2024-08-01 東邦大学
東邦大学の小沼順二准教授と京都大学の曽田貞滋教授(現・名誉教授)を中心とする研究グループは、ヒトの頭の形に関わる遺伝子と同じ遺伝子が、カタツムリ食のオサムシの1種、マイマイカブリの頭の形を決めていることを発見しました。この遺伝子の変異によって、マイマイカブリは、地域によって頭が細長い形と太短い形に分かれ、それぞれ違った方法でカタツムリを食べるように適応しています。この遺伝子は多くの動物が保有している保存性の高い遺伝子ですが、甲虫の形態の多様性に重要な役割を果たしている可能性があります。
この研究成果は、雑誌「Molecular Biology and Evolution」の2024年6月号に発表されました。

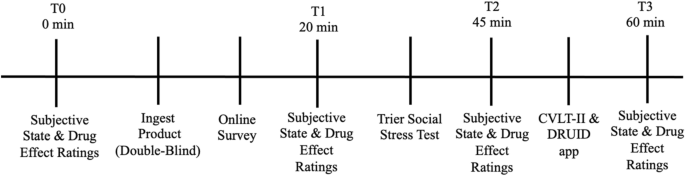
カタツムリを食べる狭頭型アオマイマイカブリ(左)と巨頭型サドマイマイカブリ(右)
発表者名
小沼 順二(東邦大学理学部生物学科 准教授)
藤澤 知親(滋賀大学データサイエンス・AIイノベーション研究推進センター 特任講師)
西山 智明(金沢大学疾患モデル総合研究センター 助教)
笠原 雅弘(東京大学大学院新領域創成科学研究科メディカル情報生命専攻 准教授)
柴田 朋子(研究当時:基礎生物学研究所生物進化研究部門 研究員)
野澤 昌文(東京都立大学大学院理学研究科生命科学専攻 准教授)
重信 秀治(基礎生物学研究所超階層生物学センター 教授)
豊田 敦(国立遺伝学研究所ゲノム・進化研究系 特任教授)
長谷部 光泰(基礎生物学研究所生物進化研究部門 教授)
曽田 貞滋(京都大学大学院理学研究科生物科学専攻 教授、現:京都大学名誉教授)
発表のポイント
- マイマイカブリのゲノム地図を作製し、形態に関わる遺伝子を特定しました。
- その遺伝子は、昆虫、軟体動物、脊椎動物など多くの動物が保有する配列保存性の高い 遺伝子で、神経や骨格などの形成に関わっています。
- 甲虫形態の適応進化を遺伝子レベルで解明するための鍵となることが期待されます。
発表概要
生物の形の違いは、多様な環境への適応を可能にし、種の多様性に関わっています。カブトムシやオサムシを含む甲虫類は、地球上の生物種の約1/4を占め、様々な環境に適応し、適応放散(注1)を遂げていますが、多様な形態の進化に関わる遺伝子はまだよく分かっていません。本研究では、日本固有のオサムシの1種で、カタツムリを専門に食べるマイマイカブリの形態変異に着目し、形の違いを決める遺伝子を探索しました。一般にマイマイカブリは、カタツムリに頭を突っ込んで食べるのに適した細長い体形をしています。しかし、一部地域のマイマイカブリは、カタツムリの殻を壊して食べるのに適した太短い頭を持ちます。全ゲノム解読と遺伝学的解析の結果、マイマイカブリの頭の形を太短く進化させた原因遺伝子はodd-pairedという遺伝子(以下opa遺伝子と表記する)(注2)であることが分かりました。opa遺伝子と相同な遺伝子は昆虫や軟体動物、ヒトを含めた脊椎動物など多くの動物に存在する保存的な遺伝子で、ヒトでは、頭の形の先天性病変と関連していることが知られています。本研究の結果、opa遺伝子が甲虫の形態の多様性に重要な働きをしている可能性が示唆されました。
発表内容
生物形態の多様性とその進化速度は、種の豊かさと相関していることが知られています。しかし、形態の多様化の根底にある分子遺伝学的メカニズムは十分に理解されていません。特に、適応放散などの近縁種間の分化がどのような遺伝的基盤によって生じているかは、未解決の問題です。この問題の解決のために、本研究は、オサムシ(オサムシ科オサムシ亜科オサムシ属)に焦点を当てました。オサムシは、体の形や大きさ、色が多様化しており、北半球を中心に約1,000種が知られています。それらは系統によって、昆虫幼虫食、ミミズ食、カタツムリ食のオサムシに分かれています(図1)。このうち、カタツムリ食のオサムシは、オサムシ全体の約半分の種数を占め、形態が著しく多様化しています。特に「狭頭型」と「巨頭型」と呼ばれる形態の分化が、異なる系統で繰り返し生じています。狭頭型とは、頭部と胸部が細長い形のオサムシで、巨頭型とは、頭部と胸部が太短い形のオサムシです。なぜカタツムリを主食とするオサムシにおいてのみ、このような形態変異が生じているのでしょうか。

図1. カタツムリを餌とするオサムシの適応放散
日本の固有種、マイマイカブリにおいても、狭頭型と巨頭型の形態変異が亜種間で生じています(図2)。行動実験の結果、狭頭型と巨頭型は、カタツムリの食べ方が大きく異なることが分かりました。狭頭型は、大きいカタツムリの殻に頭を突っ込んで食べますが、小さいカタツムリの殻に頭を入れることはできません。反対に、巨頭型は、強靭な大あごでカタツムリの殻を壊して食べることができますが、太短い頭部をカタツムリの殻に突っ込むことが下手です。このように、「頭を突っ込む」、「殻を壊す」、という2つの行動がトレードオフになっているために、カタツムリを食べるオサムシで狭頭型と巨頭型という形態の分化が起こっていると考えられます。

図2. 狭頭型アオマイマイカブリ(左)と巨頭型サドマイマイカブリ(右)
本研究では、この狭頭型と巨頭型の分化に関わる遺伝子の探索を行いました。全ゲノム配列から原因遺伝子を探し出すことを目標に、まず狭頭型の亜種アオマイマイカブリのゲノム解読(注3)を行いました。解読したゲノム配列は、複数の部分配列で構成されており、それらを染色体上に配置してつなげるために、連鎖地図の作製に取り組みました。アオマイマイカブリと巨頭型のサドマイマイカブリの交雑集団を作出し、RAD-Seq法(注4)による分子マーカーを使って連鎖解析(注5)を行いました。その結果、マイマイカブリの染色体数に対応した14連鎖群から成る高密度の連鎖地図が得られました。この連鎖地図上に解読したゲノム配列を配置して、ゲノム地図を完成させることができました(図3)。

図3. マイマイカブリのゲノム地図。14本の染色体の連鎖地図(左)と物理地図(右)
狭頭型アオマイマイカブリと巨頭型サドマイマイカブリを交配して生まれた雑種個体は中間的な形をしていました(図4のF1雑種)。形態変異に関わる遺伝子が染色体上のどこにあるかを推定するために、亜種間の交雑集団を用いてQTLマッピング(注6)を行いました。その結果、頭部形態の変異に関わる遺伝子が第6染色体上の1領域に存在していることが分かりました。さらに遺伝子発現解析(注7)を行い、ショウジョウバエなどの先行研究とも比較した結果、その領域内にあるopa遺伝子が原因遺伝子と推定されました。

図4. 狭頭型アオマイマイカブリと巨頭型サドマイマイカブリと雑種個体
狭頭型と巨頭型のopa遺伝子から作られるタンパク質のアミノ酸配列は同じだったため、遺伝子発現量の違いが形態的な差異を生み出していると考えられました。実際、opa遺伝子は、蛹になる直前の幼虫(前蛹)において、巨頭型よりも狭頭型で高度に発現していました。opa遺伝子の発現差が形態の違いを生じさせているかを検証するため、RNA干渉実験(注8)を行いました。その結果、opa遺伝子の発現を抑制した狭頭型の幼虫は、巨頭型のように太短い形態の成虫になることを確認することができました。発現を抑制した個体は、体が小さく、脱皮不全も見られたことから、opa遺伝子は、体の大きさや羽化にも関わる遺伝子と考えられます。また、野生集団を用いて狭頭型と巨頭型のマイマイカブリのゲノムを比較した結果、opa遺伝子の非コード配列に自然選択の痕跡(注9)を見つけることができました。特に、3’側非翻訳領域(注10)内の転写因子結合部位にある一塩基多型が、亜種間の形態差をもたらしている可能性が考えられました。
opa遺伝子と相同な遺伝子は、Zic遺伝子ファミリーとして昆虫や軟体動物、脊椎動物など、多くの動物分類群に見られる保存性の高い遺伝子です。ヒトでは、この遺伝子の変異が頭蓋縫合早期癒合症、全前脳胞症、ダンディー・ウォーカー症候群などの頭の形に関わる先天性病変と関連していることが報告されています。opa遺伝子のように広範な生物で共有される保存的遺伝子では、近縁な種間でのアミノ酸配列の変異はほとんどありません。マイマイカブリでもアミノ酸配列の変異はありませんでしたが、遺伝子の発現量を調節している領域に変異が生じることで、亜種間で形態や行動、餌資源の違いが生じたと考えられます。これは、適応放散の遺伝的基盤として、遺伝子の発現量を調節する「シス調節エレメントの変異」(注11)が重要であるという仮説を裏付ける結果です。今後、様々な甲虫におけるopa遺伝子の働きを調べることにより、この遺伝子が甲虫全体の形態多様化に果たしてきた役割を解明できるものと期待されます。
発表雑誌
雑誌名
「Molecular Biology and Evolution」(2024年6月10日)
論文タイトル
odd-paired is involved in morphological divergence of snail-feeding beetles
著者
Junji Konuma*, Tomochika Fujisawa, Tomoaki Nishiyama, Masahiro Kasahara, Tomoko F. Shibata, Masafumi Nozawa, Shuji Shigenobu, Atsushi Toyoda, Mitsuyasu Hasebe, Teiji Sota*
DOI番号
10.1093/molbev/msae110
論文URL
https://academic.oup.com/mbe/article/41/6/msae110/7690704
研究支援
本研究はMEXT/JSPS科研費(JP24770020, JP15K18615, JP21K06340, JP23128507, JP25128707, JP18H04010, JP22128001, JP22128008, JP221S0002)などの支援を受け行われました。
用語解説
(注1)適応放散
急速に系統分岐する系統群において、生態的な多様性が増加するとともに、形態や体色などの表現型の多様性も増加する過程を指します。
(注2)odd-paired遺伝子(opa遺伝子)
Zic遺伝子ファミリーの一つで、C2H2型ジンクフィンガー構造をもつ転写因子(遺伝子の発現を制御するタンパク質)の遺伝子です。相同な遺伝子は、後生動物の多くで見つかっており、神経や骨格などの形成に関わることが知られています。
(注3)ゲノム解読
生物の全遺伝情報(ゲノム)を解読し、その塩基配列を明らかにする過程です。本研究では、イルミナ社製シーケンサーによる短いDNA塩基配列1,450億塩基とPacBio社製シーケンサーによる長いDNA塩基配列110億塩基を使ってゲノム解読を行いました。
(注4)RAD-Seq法
RAD-Seq(Restriction-site Associated DNA Sequencing)法とは、ゲノムDNAを特定の制限酵素で切断し、制限部位に隣接するDNA断片の塩基配列情報を取得する手法です。本研究では、この手法を使って134個体から770億塩基の配列情報を取得し、1,533個の分子マーカーを獲得しました。
(注5)連鎖解析
遺伝的連鎖を調べ、同じ染色体上に乗っている遺伝子や、遺伝子間の距離を推定する研究手法を指します。本研究では、RAD-Seq法によって獲得した分子マーカーの連鎖解析を行い、マーカー間の相対的位置を一直線上に表した連鎖地図を作製しました。
(注6)QTLマッピング
QTLマッピング(Quantitative Trait Locus Mapping)とは、量的形質に関連する遺伝子座を特定するための統計遺伝学的手法です。本研究ではF1雑種のメスをアオマイマイカブリのオスに戻し交雑した雑種個体集団を用いて、QTLマッピングを行いました。
(注7)遺伝子発現解析
特定の遺伝子が細胞や組織においてどの程度発現しているかを測定し、その差異を解析するための手法です。本研究では、前蛹から得られた220億塩基のトランスクリプトーム情報を使って、狭頭型マイマイカブリと巨頭型マイマイカブリとの間で発現が異なる遺伝子を特定しました。
(注8)RNA干渉実験
二本鎖RNAを使って特定のmRNAを分解させ、遺伝子発現を抑制する実験です。本研究では、opa遺伝子の配列を持つ短い二本鎖RNAをアオマイマイカブリの前蛹に注入し、opa遺伝子の発現を抑制した成虫個体を作出しました。
(注9)自然選択の痕跡
自然選択の過程で有利な遺伝子変異が集団内で急速に増加すると、当該遺伝子変異とその近傍の遺伝子座における遺伝的多様性が著しく減少します。このような自然選択の痕跡を調べるために、本研究では、ZengのE値、標準化されたFayとWuのH値、田嶋のD値を計算し、中立進化を仮定したコアレセントシミュレーション値と比較しました。
(注10)3’側非翻訳領域
転写によって生じたメッセンジャーRNAのなかで、コード領域より下流側(3’側)の翻訳されない領域を指します。
(注11)シス調節エレメントの変異
遺伝子発現の調節に関わるDNA領域の変異を指します。コード配列の変異はタンパク質の構造を変えますが、非コード配列の変異はタンパク質構造を変えず発現量のみを調節する場合があります。そのようなシス調節エレメントの変異は、生物の様々な機能に調和した動的な微調整を可能にし、特異的な環境への適応など、近縁種の分化に重要である可能性が指摘されています。
以上
お問い合わせ先
【研究に関するお問い合わせ】
東邦大学理学部生物学科
准教授 小沼 順二
【報道に関するお問い合わせ】
学校法人東邦大学 法人本部経営企画部
京都大学 渉外・産学官連携部広報課 国際広報室
滋賀大学 広報課
金沢大学 医薬保健系 事務部総務課総務係
東京大学 大学院新領域創成科学研究科 広報室
基礎生物学研究所 広報室
東京都公立大学法人 東京都立大学管理部企画広報課広報係
国立遺伝学研究所 広報室