2025-08-29 スイス連邦工科大学ローザンヌ校(EPFL)
<関連情報>
- https://actu.epfl.ch/news/an-open-source-ai-platform-to-democratize-protei-2/
- https://www.nature.com/articles/s41586-025-09429-6
BindCraftを用いた機能性タンパク質バインダーの一発設計 One-shot design of functional protein binders with BindCraft
Martin Pacesa,Lennart Nickel,Christian Schellhaas,Joseph Schmidt,Ekaterina Pyatova,Lucas Kissling,Patrick Barendse,Jagrity Choudhury,Srajan Kapoor,Ana Alcaraz-Serna,Yehlin Cho,Kourosh H. Ghamary,Laura Vinué,Brahm J. Yachnin,Andrew M. Wollacott,Stephen Buckley,Adrie H. Westphal,Simon Lindhoud,Sandrine Georgeon,Casper A. Goverde,Georgios N. Hatzopoulos,Pierre Gönczy,Yannick D. Muller,Gerald Schwank,… Bruno E. Correia
Nature Published:27 August 2025
DOI:https://doi.org/10.1038/s41586-025-09429-6
Abstract
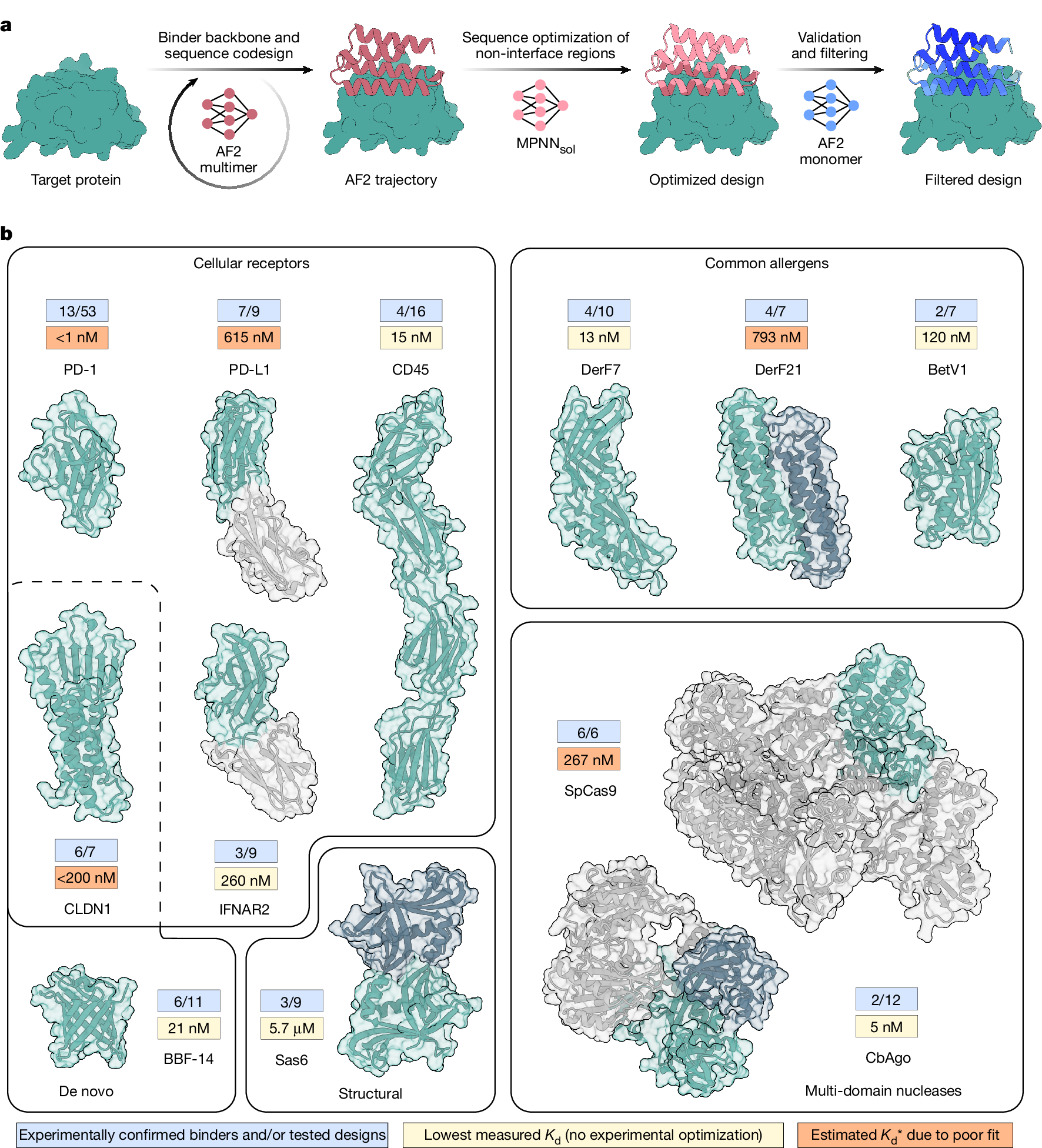
Protein–protein interactions are at the core of all key biological processes. However, the complexity of the structural features that determine protein–protein interactions makes their design challenging. Here we present BindCraft, an open-source and automated pipeline for de novo protein binder design with experimental success rates of 10–100%. BindCraft leverages the weights of AlphaFold2 (ref. 1) to generate binders with nanomolar affinity without the need for high-throughput screening or experimental optimization, even in the absence of known binding sites. We successfully designed binders against a diverse set of challenging targets, including cell-surface receptors, common allergens, de novo designed proteins and multi-domain nucleases, such as CRISPR–Cas9. We showcase the functional and therapeutic potential of designed binders by reducing IgE binding to birch allergen in patient-derived samples, modulating Cas9 gene editing activity and reducing the cytotoxicity of a foodborne bacterial enterotoxin. Last, we use cell-surface-receptor-specific binders to redirect adeno-associated virus capsids for targeted gene delivery. This work represents a significant advancement towards a ‘one design-one binder’ approach in computational design, with immense potential in therapeutics, diagnostics and biotechnology.


