希少なサンプルの長鎖DNAメチル化解析が可能に
2021-05-25 東京大学
発表のポイント
◆これまでの長いDNAのメチル化(注1)解析手法では、1マイクログラム程度のDNAが必要でしたが、その1/100の量(10ナノグラム)のDNAから実施できる方法を開発しました。
◆少量の乳がんサンプルについて実施することに成功し、現在良く行われている短いDNAを読み取る法では解析ができなかったDNA上の場所のメチル化解析に成功しました。
◆今回の方法により、大量のDNAが取れない小さな臨床検体や、わずかにしか存在しない種類の細胞などで長いDNAのメチル化解析を行うことができるようになり、さまざまな病気や細胞分化について新たな知見を生むことが期待されます。
発表概要
東京大学大学院新領域創成科学研究科の鈴木 穣教授と関 真秀特任准教授らは、少量のDNAから実施できる長いDNAのメチル化解析手法nanoEMを開発して、乳がんのサンプルに応用しました。
DNAメチル化は、合成されるRNA量の調節などに中心的な役割を果たしていて、さまざまな病気や細胞の分化などに重要であることがわかっています。これまでのDNAメチル化解析手法は、短いDNAを解析する方法が主で、1本の長いDNAがどのようにメチル化されているのかは十分にわかっていませんでした。近年、ナノポアシークエンサー(注2)といった長いDNAを読み取ることのできる機械が登場し、長いDNAのメチル化の解析が可能となりましたが、たくさんのDNAを必要とするため解析できるサンプルの種類が限られていました。
今回、酵素を利用した塩基変換法とナノポアシークエンスを組み合わせて、通常のナノポアシークエンスの1/100程度のDNA量から実施できる全ゲノムDNAメチル化解析手法nanoEMを開発しました。さらに、nanoEMを少量の臨床サンプルでも実施できることを示しました。
今回開発した方法により、大量のDNAが取れない小さな臨床サンプルやわずかにしか存在しない種類の細胞などで長いDNA上のメチル化状態の解析を行えるようになり、さまざまな病気や細胞分化についての新たな知見を生むことが期待されます。
本研究成果は、「Nucleic Acids Research」2021年5月21日版に掲載されました。
本研究は、日本学術振興会科学研究費助成事業 若手研究(21K15074及び19K16108)、文部科学省科学研究費助成事業 新学術領域研究(16H06279及び17H06306)、学術変革領域研究(A)(20H05906)、国立がん研究センター研究開発費(29-A-06)の支援を受けて行われました。
発表内容
研究の背景・先行研究における問題点
DNA塩基のシトシンのメチル化修飾(注1)は、遺伝子の発現量の調節などに中心的な役割を果たしており、さまざまな疾患や発生・分化に重要な役割を担っています。これまでのDNAメチル化解析は短いDNAを読み取る短鎖シークエンサーやメチル化アレイを用いた方法が用いられてきました。そのため、1本の長いDNAのメチル化パターンや、短鎖シークエンスで検出が難しいトランスポゾンなどの繰り返し配列や構造変異の周りのメチル化状態については、ほとんどわかっていませんでした。
一般的に使用されているDNAメチル化解析手法として、塩基変換した短いDNAを短鎖シークエンサーでシークエンスするバイサルファイトシークエンス法(BS-seq)などの方法が存在します。BS-seqでは、バイサルファイト処理(注3)によりメチル化されていないシトシンのみをウラシルに変換した後、PCRでチミンに置換します。その結果、塩基置換の有無として、非メチル化とメチル化シトシンを区別することができます。しかし、バイサルファイト処理は強い化学反応であるため、DNAがバラバラに分解されてしまい長いDNAを得ることが難しいという問題点が存在していました。近年、Enzymatic Methyl sequencing(EM-seq)と呼ばれる方法が開発され、同様の塩基変換を酵素反応によって穏やかに行うことが可能となり、バイサルファイト処理よりも長いDNAを得られるようになってきました。
現在、1分子シークエンサーであるナノポアシークエンサーによって、長い1本のDNA上のメチル化の解析が可能となっていますが、必要なDNA量が1マイクログラム程度のため、小さな臨床検体や希少な種類の細胞では実施することが不可能でした。微量のDNAから、EM-seqやバイサルファイト処理などで塩基変換したDNAを領域特異的なPCRで増幅して長鎖シークエンスする方法が最近発表されました(注4)が、全ゲノムを対象とした方法は存在していませんでした。また、塩基変換を介した全ゲノム長鎖DNAメチル化解析に用いることのできる実用的なデータ解析パイプラインも存在していませんでした。
研究内容
2種類の乳がん細胞株を用いて、微量DNAからの全ゲノム長鎖DNAメチル化解析法nanoEM及びデータ解析パイプライン(注5)の開発を行い、3症例の乳がん臨床検体に適用しました。
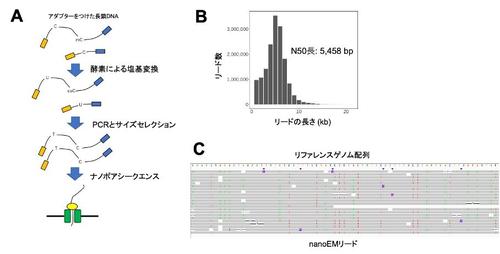
まず、2種類の乳がん細胞株を使用して検討を行いました。短鎖シークエンサーを利用したEM-seqとBS-seqを比較することで、手法間で検出されるメチル化率の相関が高いことやEM-seqの方がゲノム全体を偏りなく検出できることを確認しました。次に、1-50ナノグラムのゲノムDNAをEM-seqにより塩基変換し、ロングPCRにより全ゲノム増幅後、ナノポアシークエンスすることにより、5 kb程度の長さのリードが得られることを示しました(図1)。nanoEMデータを解析するために、Bismarkなどの短鎖シークエンス用解析パイプラインにも用いられるデータ解析戦略を援用することで、新たに長鎖DNAメチル化解析用のパイプラインを開発しました。
このパイプラインを用いてnanoEMデータを解析し、短鎖と長鎖シークエンスを用いた既存の3つの方法で測定したメチル化状態との比較を行ったところ、いずれの方法とも高い相関を示しました。さらに、インプットDNA量の異なるnanoEMデータ同士を比較したところ、10ナノグラム以上のゲノムDNAからであれば、カバーされる領域の偏りが少ないデータが得られることを確認しました。また、染色体ごとに異なるメチル化パターンを持つインプリンティング(注6)領域や、短鎖シークエンスでは検出が難しいリピート領域内や構造変異周辺のメチル化パターンの検出が行えることを示しました(図2)。
さらに、3症例の乳がんの臨床検体のがん部と非がん部について、25ナノグラム程度のゲノムDNAを使用してnanoEMを実施しました。nanoEMを用いて、がん特異的なメチル化パターンや構造変異周辺のメチル化パターンの検出が行えることを示しました。このことから、nanoEMを微量の臨床検体にも応用できることが確認できました。
社会的意義・今後の予定など
今回の方法により、大量のDNAが取れない微量な臨床検体やわずかにしか存在しない希少な細胞種などで全ゲノム長鎖DNAメチル化解析を行えるようになり、さまざまな疾患や細胞分化についての新たな知見を生むことが期待されます。
発表雑誌
雑誌名:「Nucleic Acids Research」(オンライン版:5月21日)
論文タイトル:Long-read whole-genome methylation patterning using enzymatic base conversion and nanopore sequencing
著者:Yoshitaka Sakamoto, Suzuko Zaha, Satoi Nagasawa, Shuhei Miyake, Yasuyuki Kojima, Ayako Suzuki, Yutaka Suzuki*, Masahide Seki*
DOI番号:10.1093/nar/gkab397
URL:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab397/6279847?searchresult=1
発表者
鈴木 穣(東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 教授)
関 真秀(東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 特任准教授)
用語解説
(注1)DNAメチル化:真核生物のDNAのシトシン塩基は高い頻度でメチル化修飾を受けています。多くの場合、このシトシンのメチル化修飾をDNAメチル化と呼びます。DNAメチル化は合成されるRNA量の抑制などの機能があり、さまざまな病気や細胞の発生・分化に重要なことが知られています。
(注2)ナノポアシークエンサー:ナノポアと呼ばれる数ナノメートルの穴をDNAやRNAが通るときの電流の変化を読み取ることで、DNAやRNAの配列を読み取ることのできる機械です。ナノポアを使用して配列を読み取ることをナノポアシークエンスといいます。
(注3)バイサルファイト処理:バイサルファイト(亜硫酸水素塩)でDNAのシトシンをウラシルに化学的に変換する処理です。メチル化されているシトシンはウラシルに変換されないため、シトシン塩基がメチル化されているかどうかを調べるために利用されています。
(注4)領域特異的なPCRによる長鎖DNAメチル化解析法:バイサルファイト処理やEM-seqの酵素反応などによって塩基変換したDNAについて、特定のゲノム上の領域に設計したプライマーを使用してPCR増幅し、シークエンスする方法で、SMRT-BS、LR-EM-seqなどの手法が存在しています。これらの方法では、測定したい領域を選べるというメリットが存在しますが、領域ごとにプライマーの作成する必要があるなど、ゲノム全体を網羅した解析はできませんでした。
(注5)今回開発したデータ解析パイプライン:これまでの塩基変換を利用した長鎖DNAメチル化解析には、短鎖シークエンス用のソフトウェアをそのまま転用されることが多かったのですが、全ゲノムレベルの解析に使用することは困難でした。そこで、短鎖シークエンスでも利用される塩基変換を考慮したアラインメントの方法を長鎖用のマッピングソフトウェアminimap2に適用することで、新たなパイプラインを開発しました。開発したデータ解析パイプラインはGitHubに公開しています(https://github.com/yos-sk/nanoEM)。
(注6)インプリンティング:ヒトのDNAは、父親由来と母親由来の2組存在していて、多くの遺伝子は両方のDNAから発現しますが、一部の遺伝子については片方の親由来のDNAからのみ選択的に発現します。この現象のことをインプリンティング(あるいは、ゲノムインプリンティング)と呼びます。このインプリンティング制御をDNAのメチル化が行っていることが知られています。
添付資料
図1.長鎖DNAの全ゲノムメチル化解析法nanoEM
(A)nanoEMの概略図。(B)乳がん細胞株のnanoEMリードの長さごとの数。
(C)nanoEMリードのリファレンスゲノムへのマッピング結果。リファレンスゲノムのCの位置にTの塩基置換、もしくはG(Cの相補塩基)の位置にA(Tの相補塩基)の塩基置換があることが確認できます。
図2.nanoEMによるインプリンティング領域や構造変異周辺のメチル化の検出
(A)染色体ごとにメチル化状態の異なるインプリンティング領域にマップした乳がん細胞株由来のnanoEMリード。メチル化されていないリードのみに、片方の染色体にしか存在しないCからGの一塩基多型が存在することがわかります。(B)乳がん臨床検体において、nanoEMで検出された大規模な欠失の周辺のメチル化パターン。欠失をカバーしたリードでは、四角で囲った場所がメチル化されていないことがわかります。
お問い合わせ
新領域創成科学研究科 広報室