2018-08-08 理化学研究所
理化学研究所(理研)生命機能科学研究センターエピジェネティクス制御研究ユニットの梅原崇史ユニットリーダー、生命医科学研究センターエピゲノム技術開発ユニットの蓑田亜希子ユニットリーダー、ゲノム制御ネットワーク研究チームのボグミル・カチコフスキー研究員、ライフサイエンス技術基盤研究センター機能性ゲノム解析部門エピゲノム技術開発ユニット(研究当時)のルーシー・ハンドコ研究員らの共同研究グループ※は、細胞のがん化に関わるエピゲノム[1]の目印を検出する技術を開発し、ヒトの肺がん細胞株における「エピゲノム地図[1]」を解明、公開しました。
本研究成果は、がん治療薬などの効果をエピゲノムのレベルで精密に判定する手法の開発につながると期待できます。
多くのがんにおいて、エピゲノムの異常によるがん遺伝子の発現上昇が確認されています。BETファミリータンパク質[2]は高アセチル化[3]ヒストン[4]への結合を介してがん化に関わり、この結合を阻害する薬剤をがん細胞に投与すると、遺伝子発現パターンが正常細胞に近づくケースが知られています。しかし、このタンパク質がエピゲノムに結合するときの主要標的である、ヒストンH4の高アセチル化修飾に対する阻害剤の効果は不明でした。今回、共同研究グループは、BETファミリータンパク質に対する阻害剤をヒト肺がん細胞株に投与してもヒストンH4の高アセチル化は影響を受けず、この修飾が極めて頑強な「がんエピゲノム」の目印であることを突き止めました。
本研究は、英国の科学雑誌『Epigenetics』の掲載に先立ち、オンライン版(8月6日付け)に掲載されました。
図 ヒストンとDNAの立体模型
(白がDNA、緑、青、赤、黄がヒストン8量体で、緑がヒストンH4である)
※共同研究グループ
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門
エピゲノム技術開発ユニット(研究当時)
研究員 ルーシー・ハンドコ (Lusy Handoko)
生命医科学研究センター
ゲノム制御ネットワーク研究チーム
研究員 ボグミル・カチコフスキー (Bogumil Kaczkowski)
ゲノム情報解析チーム
チームリーダー チョウヂョン・ホン (Chung-Chau Hon)
特別研究員 マリーナ・リツィオ (Marina Lizio)
エピゲノム技術開発ユニット
ユニットリーダー 蓑田 亜希子(みのだ あきこ)
研究パートタイマーI プラシャンティ・ジャヤモハン (Prashanti Jeyamohan)
(以上、旧所属はライフサイエンス技術基盤研究センター 機能性ゲノム解析部門)
生命機能科学研究センター
エピジェネティクス制御研究ユニット
ユニットリーダー 梅原 崇史 (うめはら たかし)
技師 若森 昌聡 (わかもり まさとし)
翻訳構造解析研究ユニット
ユニットリーダー 伊藤 拓宏 (いとう たくひろ)
非天然型アミノ酸技術研究チーム
チームリーダー 坂本 健作 (さかもと けんさく)
(以上、旧所属はライフサイエンス技術基盤研究センター 構造・合成生物学部門)
ライフサイエンス技術基盤研究センター 構造・合成生物学部門
タンパク質機能・構造研究チーム(研究当時)
研究員 松田 貴意 (まつだ たかよし)
科技ハブ産連本部 バトンゾーン研究推進プログラム 横山特別研究室
特別招聘研究員 横山 茂之 (よこやま しげゆき)
(旧横山構造生物学研究室 上席研究員)
東京工業大学 科学技術創成研究院 細胞制御工学研究センター
助教 佐藤 優子 (さとう ゆうこ)
教授 木村 宏 (きむら ひろし)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究B「超活性クロマチンの高分解能検出を介した疾患エピゲノム制御機構の理解(研究代表者:梅原崇史)」、科学技術振興機構(JST)戦略的創造研究推進事業さきがけ「「エピヌクレオソーム」の精密な再構成による遺伝子発現制御解析(研究代表者:梅原崇史)」による支援を受けて行われました。
背景
私たちの体を作る細胞の一つ一つに、ゲノムDNAが入っています。ヒトゲノムには約2万個の遺伝子が存在しますが、これらの遺伝子が一斉に働いているわけではありません。細胞には、特定の遺伝子をいつ、どこで、どんな環境で発現させるかを決める仕組みがあります。
その一つが、ヒストンと呼ばれるタンパク質による調節です。ヒストンは、4種類のコアタンパク質(H2A、H2B、H3、H4)が二つずつ組み合わさった8量体として存在し、糸巻きのようにDNAを巻きつけることで、長いDNAを核内にコンパクトに収納しています(クロマチン[4]構造)。またヒストンは、アセチル化やメチル化[5]、リン酸化などの修飾を受けてクロマチン構造を変化させ、ゲノムの特定領域に存在する遺伝子の発現を制御しています。このようなヒストンに対する化学修飾は可逆的であり、DNAに対する可逆的な化学修飾であるメチル化とともに「エピジェネティクス制御」と呼ばれています。エピジェネティクス制御は、個体の一生を通じて不変であるDNAの塩基配列とは異なり、可逆的に遺伝情報を制御する仕組みです。
ヒトをはじめとする真核生物のエピジェネティクス制御には、ヒストンのコアタンパク質H3とH4に対するアセチル化修飾が重要な役割を担っています。特に、H4のN末端側に存在する二つのリシン残基[6](K5とK8)が同時にアセチル化された高アセチル化状態(H4K5acK8ac)のヒストンには、「BETファミリータンパク質」が結合し、がん遺伝子の発現を活性化することが知られています。
そこで、がん抑制効果の研究として、ヒストンとBETファミリータンパク質の結合を阻害する低分子化合物(BET阻害剤)が注目されています。例えば、代表的なBET阻害剤であるJQ1[7]は、ある種のがん細胞に投与すると腫瘍促進遺伝子の発現を選択的に抑制することから、がん治療薬としての可能性が期待されています。JQ1をがん細胞に投与すると、ヒストンとBETファミリータンパク質の結合を競合的に阻害することは分かっていますが、BETファミリータンパク質がもともと結合していたヒストンH4の高アセチル化修飾のパターンがどのように変化するかなど、細胞レベルでの詳しい作用は分かっていません。特に、細胞内での高アセチル化状態のヒストンH4を特異的に検出する良い手法がこれまでなかったため、このような阻害剤ががん治療薬としてエピジェネティクス制御効果を持つかどうかを精密に判定することが困難でした。
研究手法と成果
共同研究グループはまず、高アセチル化ヒストンをエピゲノム全体にわたって詳細に解析するため、K5とK8が同時にアセチル化されたヒストンH4を特異的に認識する抗体の作製を試みました。抗体の作製には抗原が必要となるため、非天然アミノ酸をタンパク質の任意の場所に導入する理研の独自技術注1)を利用し、リシンの代わりにアセチル化リシンをK5とK8の位置に組み込んだヒストンH4を試験管内で合成し、このアセチル化ヒストンH4を抗原としてモノクローナル抗体[8]を作製しました。この抗体が高アセチル化状態(H4K5acK8a)を選択的に認識することを、X線結晶構造解析[9]により原子分解能で確認しました(図1)。この抗体を利用することで、ヒストンH4が高アセチル化された領域をゲノム規模で高精度に特定することが初めて可能となり、がん治療薬によるエピジェネティクス制御効果をより精密に分析できるようになりました。
注1)2015年5月18日「DNA情報の変換ルールを人為的に改変」
次に、このH4K5acK8ac認識抗体をChIP-seq技術[10]に適用して、ヒトの肺がん細胞のH23細胞株[11]のエピゲノムを解析しました。そして、明らかになったエピゲノムのパターンをゲノムDNAの塩基配列上に記載し、「エピゲノム地図」を作成しました。遺伝子のエンハンサー領域とプロモーター領域[12]において、今回明らかになったヒストンH4の高アセチル化修飾(H4K5acK8ac)と他の代表的なエピゲノム修飾を比較した結果、H4K5acK8acはエンハンサーとプロモーターの両方に分布しており、これまで知られているエピゲノム修飾の中ではヒストンH3のアセチル化修飾(H3K27ac)の局在と最も一致度が高いことが分かりました(図2a)。
そこで、BETファミリータンパク質の一種であるBRD2とH4K5acK8ac、または、H3K27acとの共局在を解析した結果、BRD2との共局在はH4K5acK8acの方がH3K27acよりも高く相関することが分かりました(図2b)。このことより、細胞核内でBRD2がH4K5acK8acと選択的に結合することが示されました。また、高アセチル化と遺伝子発現の関係を調べるため、CAGE法[13]を用いた遺伝子発現解析を行ったところ、遺伝子の転写産物量とH4K5acK8acのChIP-seqシグナルが強い正の相関関係にあることを確認しました。
これらの結果は、従来のエピゲノム研究において活性化エンハンサーおよびプロモーターの標準的な修飾として位置づけられてきているヒストンH3のアセチル化修飾(H3K27ac)に対して、ヒストンH4の高アセチル化修飾(H4K5acK8ac)が同等以上の活性化マークとして利用可能であることを示しています。
さらに、BET阻害剤をヒトの肺がん細胞株に添加したときに、ヒストンH4の高アセチル化修飾がどのように変化するか調べるため、代表的なBET阻害剤であるJQ1をヒトの肺がん細胞株に添加しました。JQ1の添加によって、BETファミリータンパク質のBRD2はほぼ完全にゲノムDNAから解離しましたが、ヒストンH4の高アセチル化修飾(H4K5acK8ac)は、プロモーター領域でもエンハンサー領域でもほとんど変動しないことが分かりました(図3上段)。この傾向は個々の遺伝子を解析した場合でも同様であり、代表的ながん遺伝子c-Myc[14]について調べたところ、JQ1を添加するとc-Myc遺伝子やその近くのゲノム領域においてBRD2の結合はほぼ完全に消失しましたが、H4K5acK8acのパターンには影響が見られませんでした(図3下段)。
一連の結果をまとめると、BET阻害剤を単剤で投与すると、BETファミリータンパク質はエピゲノムから解離するため、BETファミリータンパク質に依存するがんについては一時的な治療効果が期待できるが、その背景にある悪性のエピゲノムは根治されていないことが示されました。
今後の期待
今回開発したヒストンH4の高アセチル化修飾検出技術は、がん細胞に投与した薬剤がエピゲノムの変化を伴わずに一時的な作用にとどまるのか、エピゲノムの変化を伴う持続的な作用なのかを短時間で精密に判定する手法の開発につながると期待できます。また、ヒトのヒストンH4の高アセチル化修飾に関する精密なエピゲノム地図を初めて公開したことにより、他のエピゲノム修飾との相関解析が可能になり、ヒトの正常細胞と疾患細胞でのエピゲノムの相違についての理解が一層深まると考えられます。
今回、BETファミリータンパク質に対する代表的な阻害剤JQ1を単剤で投与しても、肺がん細胞株のヒストンH4の高アセチル化修飾は影響を受けず、極めて頑強な「がんエピゲノム」の目印であることが判明しました。この知見は、ヒストンH4の高アセチル化修飾の制御ががんの根本治療に必要な可能性を示しており、エピジェネティクス制御を介した新しいがん治療法の開発に向けての手がかりとなると期待できます。
原論文情報
Lusy Handoko#, Bogumil Kaczkowski#, Chung-Chau Hon, Marina Lizio, Masatoshi Wakamori, Takayoshi Matsuda, Takuhiro Ito, Prashanti Jeyamohan, Yuko Sato, Kensaku Sakamoto, Shigeyuki Yokoyama, Hiroshi Kimura, Aki Minoda*, Takashi Umehara*, “JQ1 affects BRD2-dependent and independent transcription regulation without disrupting H4-hyperacetylated chromatin states”, Epigenetics, 10.1080/15592294.2018.1469891
発表者
理化学研究所
生命機能科学研究センター エピジェネティクス制御研究ユニット
ユニットリーダー 梅原 崇史(うめはら たかし)
(ライフサイエンス技術基盤研究センター 構造・合成生物学部門 生命分子制御研究グループ エピジェネティクス制御研究ユニット ユニットリーダー(研究当時))
生命医科学研究センター エピゲノム技術開発ユニット
ユニットリーダー 蓑田 亜希子(みのだ あきこ)
(ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 オミックス応用技術研究グループ エピゲノム技術開発ユニット ユニットリーダー(研究当時))
生命医科学研究センター ゲノム制御ネットワーク研究チーム
研究員 ボグミル・カチコフスキー (Bogumil Kaczkowski)
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 エピゲノム技術開発ユニット(研究当時)
研究員 ルーシー・ハンドコ (Lusy Handoko)
お問い合わせ先
理化学研究所 生命機能科学研究センター センター長室 報道担当
山岸 敦(やまぎし あつし)
報道担当
理化学研究所 広報室 報道担当
補足説明
-
- エピゲノム、エピゲノム地図
- 細胞内の全DNAの塩基配列情報を指す「ゲノム」に対し、DNAやヒストンの化学修飾などで細胞の個性を記憶する情報を「エピゲノム」とよぶ。エピゲノムのパターンは細胞ごとに特徴があり、このパターンをゲノムDNAの塩基配列上に記載したものが「エピゲノム地図」である。本研究では特に、これまで明らかになっていなかったヒストンH4の高アセチル化修飾(H4K5acK8ac)を記載したエピゲノム地図を作成し、他のヒストン修飾や遺伝子の転写活性などの情報と合わせて解析を行なった。
-
- BETファミリータンパク質
- クロマチン関連タンパク質の一種。ヒトの核内に存在する一部のタンパク質に共通して含まれるブロモドメインと余剰末端(Bromodomain and Extra-Terminal)を持つ一連のタンパク質。ヒトでは4種類(BRD2、BRD3、BRD4、BRDT)が知られ、腫瘍促進遺伝子の発現調節に関与することから、NUT中線がん(NMC)や急性骨髄性白血病(AML)をはじめとする多くのがんの治療標的と見なされている。
-
- アセチル化
- 分子にアセチル基(CH3CO-)を結合する反応。ヒストンはアセチル基転移酵素と脱アセチル化酵素により、可逆的なアセチル化修飾を受ける。
-
- ヒストン、クロマチン
- DNAを巻き付けることで、長大なDNAを核内に納めるタンパク質をヒストンという。代表的なヒストンはH1、H2A、H2B、H3、H4の5種類があり、H2A、H2B、H3、H4の4種類(コアヒストン)が二つずつ集まってヒストン8量体を形成する。ヒストン8量体の周りにDNAが巻き付いた構造をヌクレオソームと呼び、真核生物においてヌクレオソームを基本単位とするゲノムDNAとタンパク質の高次複合体をクロマチンと呼ぶ。
-
- メチル化
- 分子にメチル基(CH3-)を結合する反応。ヒストンはメチル基転移酵素と脱メチル化酵素により、可逆的なメチル化修飾を受ける。また、哺乳類のDNAは主にシトシン・グアニン(CG)配列のCがメチル化される。ヒトゲノムの60~90%以上のCG配列がメチル化している一方、遺伝子発現調節領域ではしばしば低メチル化状態が観察される。
-
- リシン残基
- タンパク質を構成するアミノ酸の一つ。1文字略称はK。側鎖にもアミノ基を持つため、タンパク質中の残基としてアミノ基へのアセチル化やメチル化修飾の標的となる。
-
- JQ1
- 膜透過性を持つ低分子であり、BETファミリータンパク質のアセチル化リシン認識部位に結合してヒストンとBETファミリータンパク質の結合を競合的に阻害する。多くのがんや一部の免疫疾患に対する治療効果が期待されている。
-
- モノクローナル抗体
- 抗体産生細胞とがん細胞が融合したハイブリドーマが産生する抗体。抗体産生細胞は主として1種類の抗体を作るため、特定の抗体を大量に得ることができる。
-
- X線結晶構造解析
- 物質の結晶を作り、それにX線を照射して得られる回折データを解析することにより、物質の内部構造を調べる方法。タンパク質の立体構造を原子レベルの分解能で詳細に解明するための最も有力な方法の一つである。
-
- ChIP-seq技術
- ChIPはクロマチン免疫沈降(Chromatin immunoprecipitation)のこと。クロマチンに含まれる分子を抗体で免疫沈降し、その領域のDNA配列を解読する方法。共沈降したDNAを次世代シーケンサーを用いて、数千万本から数億本のDNA配列の断片を並列処理で解読・分析する技術をChIP-seqと呼ぶ。ChIP-seq技術をエピゲノム解析に応用すると、特定のエピゲノム修飾が存在するゲノム領域を高分解能で特定化することができる。
-
- H23細胞株
- KRAS遺伝子およびTP53遺伝子に変異を持ち、代表的なBET阻害剤であるJQ1やiBETに対して感受性を示す非小細胞肺がん(NSCLC)の一種。
-
- エンハンサー領域とプロモーター領域
- どちらも遺伝子発現を制御する機能を持つDNA配列。主に遺伝子の上流や下流に位置し、遺伝子の転写効率を変化させるDNAの特定の配列のうち、転写効率を著しく高める部分をエンハンサー領域(配列)という。これに対し、ゲノムDNA上でRNAとして転写される領域の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーター領域(配列)という。
-
- CAGE法
- ゲノムから転写された遺伝子産物(RNA)の開始点を捕捉し、転写産物および転写されたエンハンサーの開始位置をゲノム規模で正確に決定する手法。理研が2003年に開発した。CAGEは、Cap Analysis Gene Expressionの略。
-
- c-Myc
- 細胞増殖などに関わる遺伝子の発現を促進する転写因子Mycをコードする遺伝子。がん化にも関与する。
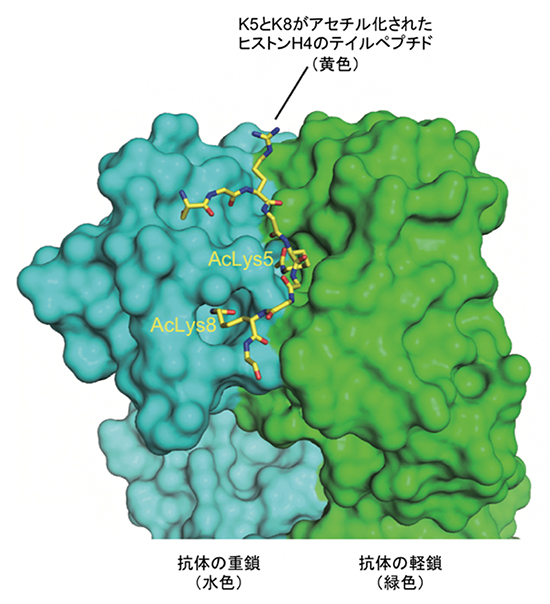
図1 ヒストンH4の高アセチル化修飾(H4K5acK8ac)を認識する抗体
今回開発したモノクローナル抗体の抗原認識部位断片(Fab)とヒストンH4のN末端側テイルペプチドとの複合体のX線結晶構造を示す。ヒストンH4のテイルペプチド(黄色)は5番目のリシン(K5; Lys5)と8番目のリシン(K8; Lys8)がアセチル化(Ac)されている。今回開発した抗体は重鎖(水色)と軽鎖(緑色)で構成する中央の溝に2カ所の凹みを持っており、ヒストンH4のK5とK8の2カ所のリシン残基がアセチル化された高アセチル化状態を選択的に認識する仕組みを備えていた。
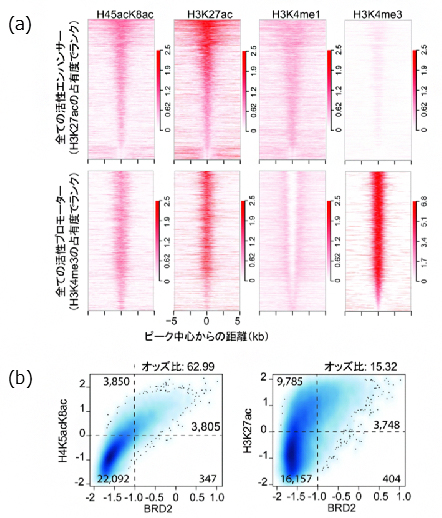
図2 ヒストンH4の高アセチル化修飾(H4K5acK8ac)のゲノム規模解析
(a)ヒト肺がん細胞株H23のエンハンサー領域またはプロモーター領域におけるエピゲノム修飾の可視化マップ。左から、ヒストンH4の高アセチル化修飾(H4K5acK8ac)、ヒストンH3のアセチル化またはメチル化修飾(H3K27ac、H3K4me1、H3K4me3)をそれぞれ示している。横軸は各ピークの中心からの距離(-5kbから+5kbまで)を示し、縦軸はH3K27ac(上側のエンハンサーのパネル)またはH3K4me3(下側のプロモーターのパネル)のシグナル強度でランク付けされたエンハンサーまたはプロモーターを上位(上)から下位(下)へ並べて示している。H4K5acK8acはエンハンサーおよびプロモーターの両方に分布しており、またH3K4me1、H3K4me3H3よりもH3K27acの局在と一致していることも分かる。
(b)BETファミリータンパク質(BRD2)とH4K5acK8ac、またはH3K27acとの共局在密度プロット。横軸は規格化されたBRD2のChIP-seqシグナル(log10 RPM + 0.25)を示し、縦軸は規格化されたH4K5acK8ac(左)またはH3K27ac(右)のChIP-seqシグナルを示す。BRD2シグナルの閾値(-1.0)、H4K5acK8acおよびH3K27acシグナルの閾値(0)をそれぞれ点線で示している。オッズ比(ある事象の起こりやすさについて二つの群で比較したときの違いを示す統計学的尺度)は、この分割表を用いてFisher Exact Testで計算した。この解析から、BRD2との共局在は、オッズ比の高いH4K5acK8acの方がH3K27acよりも高く相関することが判明した。
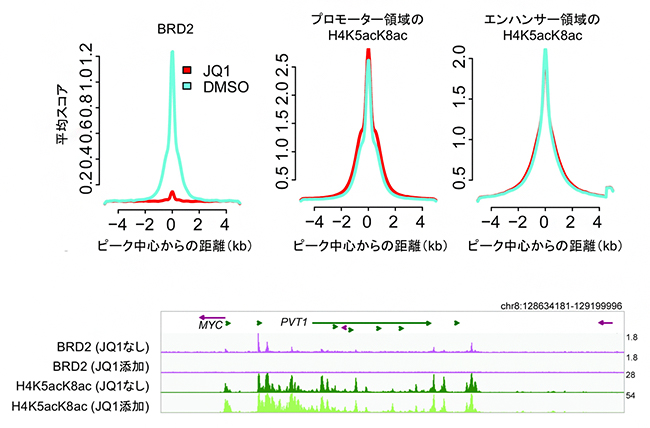
図3 BET阻害剤によるヒストンH4の高アセチル化修飾(H4K5acK8ac)への影響
(上段)BET阻害剤のJQ1を500nM(ナノモーラー、10億分の1モル濃度)の濃度で、ヒト肺がん細胞株H23に24時間添加した後のChIP-seqスコアを示す。左はBRD2、中央はプロモーター領域のH4K5acK8ac、右はエンハンサー領域のH4K5acK8acのデータで、水色が溶媒のDMSO溶液のみを添加したコントロール条件、赤色がJQ1を添加した条件でのプロットを示す。横軸は各ピークの中心からの距離(-4kbから+4kbまで)を示し、縦軸はChIP-seqの平均スコアを示す。この解析の結果、BET阻害剤のJQ1を添加するとBRD2はゲノムから解離する(左)一方、ヒストンH4の高アセチル化修飾(H4K5acK8ac)はプロモーター領域でもエンハンサー領域でも変動しないことが分かる(中央、右)。
(下段)JQ1添加によるヒト8番染色体c-Myc遺伝子周辺のChIP-seqシグナルの変動を示す。c-Myc遺伝子の位置を紫色の矢印、その近くのPVT1遺伝子の位置を緑色の矢印で示す。JQ1を添加するとこのゲノム領域のBRD2の結合はほぼ完全に消失するのに対し、ヒストンH4の高アセチル化修飾(H4K5acK8ac)は影響を受けていないことが確認された。