2024-03-14 理化学研究所,東京大学,愛知学院大学
理化学研究所(理研)開拓研究本部 岩崎RNAシステム生化学研究室の岩崎 信太郎 主任研究員、アントニオス・アポストロプロス 国際プログラム・アソシエイト(研究当時、現研究生、東京大学 生産技術研究所 特任研究員)、河本 尚大 学振特別研究員PD、七野 悠一 研究員、東京大学 生産技術研究所の池内 与志穂 准教授、周 小余 特任助教、愛知学院大学 薬学部の築地 仁美 教授らの共同研究グループは、目的の遺伝子の翻訳[1]を極めて特異的に抑制する新手法CRISPRδ(クリスパー・デルタ)を開発しました。
本研究成果は、遺伝子の機能を理解するという基礎生物学の発展に貢献するだけでなく、原因遺伝子の機能抑制を通じた疾患治療など、さまざまな応用につながることが期待されます。
遺伝子の機能を理解するためには、何らかの手法でその遺伝子の発現量を抑制し、抑制による影響を調べる必要があります。しかし、既存の手法では目的の遺伝子以外も抑制してしまうことがあり、問題となっていました。
共同研究グループは、dCas13[2]というタンパク質を標的メッセンジャーRNA(mRNA)[3]に結合させ、その翻訳を非常に特異的に抑制する新手法CRISPRδを開発しました。CRISPRδは、ゲノムに通常存在する内在性の遺伝子だけでなく、ウイルス特異的な翻訳様式や神経変性疾患の原因となり得る特殊な翻訳様式も抑制できることが分かりました。
本研究は、科学雑誌『Nature Communications』オンライン版(3月11日付)に掲載されました。
CRISPRδ(クリスパー・デルタ)による超特異的翻訳抑制
背景
あらゆる生物はDNA上の遺伝子情報をもとにタンパク質を合成することで生命を維持しています。ある遺伝子の機能を理解するためには、その遺伝子から合成されるタンパク質量を特異的に減少させ、その影響を調べる必要があります。このような方法は遺伝子ノックダウンと呼ばれ、生命現象の基本原理の理解だけでなく、バイオテクノロジーや疾患治療などの応用面においても大きく貢献してきました。
遺伝子発現では、まずDNAからmRNAが合成され、その後リボソーム[4]がmRNAの配列を読み取りタンパク質を合成します。そこで、中間産物であるRNAを狙って分解できれば、特異的な遺伝子ノックダウンを行うことができます。例えば、短い二本鎖RNAと相補的な配列を持つRNAが分解されるRNA干渉[5]という現象を利用した方法は世界中で広く使われています。しかし、この方法では認識に必要な相補的配列が短く、似た配列を持つ標的以外のRNAも分解されてしまい、問題となっていました。
現在、CRISPR-Cas[2]と呼ばれる細菌の免疫に関わるシステムが注目を浴びています。最近、このシステムの一部としてCas13[2]というタンパク質が発見されました。Cas13はガイドRNAという短いRNAと結合すると、その配列に相補的なRNAを特異的に認識して切断する酵素です(図1)。Cas13は標的RNAの認識により長い相補的配列を必要とするため、RNA干渉よりも特異性の高いノックダウン効果が期待されました。しかし、ひとたびCas13が標的RNAと結合すると、その近くにいるRNAを非特異的に切断してしまうことが分かり、Cas13を用いて特異的な遺伝子ノックダウンを行うのは難しい状況が続いていました。
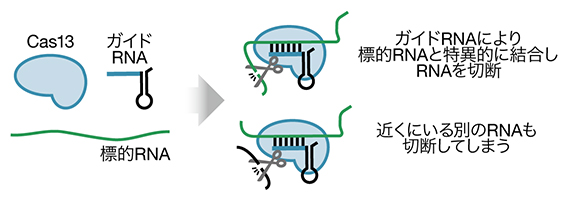
図1 Cas13によるRNA切断
Cas13はガイドRNAと複合体を作り、ガイドRNAの配列と相補的な標的RNAと強く結合する。その後、RNA切断活性を示して標的RNAを分解するが、その際近くに存在する標的以外のRNAも切断してしまう。
研究手法と成果
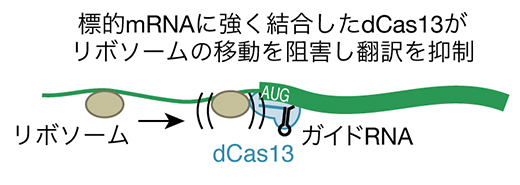
共同研究グループはCas13のRNA結合における高い特異性を生かし、RNAを分解させるのではなく翻訳阻害を引き起こす新手法CRISPRδ(クリスパー・デルタ、deltaはDEpLetion of Translation by blockAdeの略)を開発しました。翻訳の開始段階ではリボソームがmRNAの末端に誘導され、塩基配列をスキャンしながら開始コドンを探索します。その途中にRNA切断活性を失ったCas13変異体(dCas13)が強固に結合していると、リボソームの移動の邪魔になり翻訳が阻害されるのではないかと考えました(図2A)。実際にレポーターmRNA[6]の開始コドン近辺にdCas13を結合させると、レポーターmRNAから翻訳されるタンパク質量が減少しました(図2B左)。その際、RNA量の減少は起こりませんでした(図2B右)。
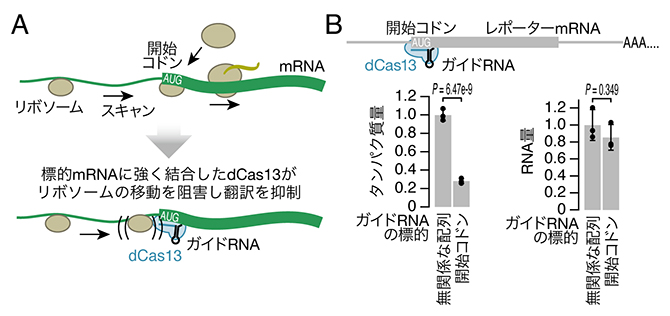
図2 CRISPRδによる翻訳抑制
(A)翻訳開始ではmRNA上をリボソームが移動し開始コドン(AUG)を探索する必要がある。この途中にdCas13が強く結合していると、リボソームの移動が阻害され翻訳が抑制される。
(B)CRISPRδによるレポーターmRNAの翻訳抑制。開始コドンを標的としたガイドRNAを導入した場合にのみ、レポーターmRNAから翻訳されるタンパク質量が減少した。一方、RNA量にはほとんど変化がなかった。
次にCRISPRδの特異性を調べるため、次世代シーケンサー[7]を用いて細胞内の全遺伝子の翻訳状況を網羅的に解析するリボソームプロファイリング法[8]を行いました。その結果、標的としたレポーターmRNAの翻訳のみが特異的に低下し、細胞内のその他のmRNAの翻訳にはほぼ影響しないことが分かりました(図3A)。一方、RNA切断活性を持つCas13で同様の実験を行うと、レポーターmRNAだけでなく、さまざまな遺伝子のRNA量が変動していました(図3B)。このことからCRISPRδはCas13を用いたRNA切断をはるかに上回る特異性を持つことが示されました。
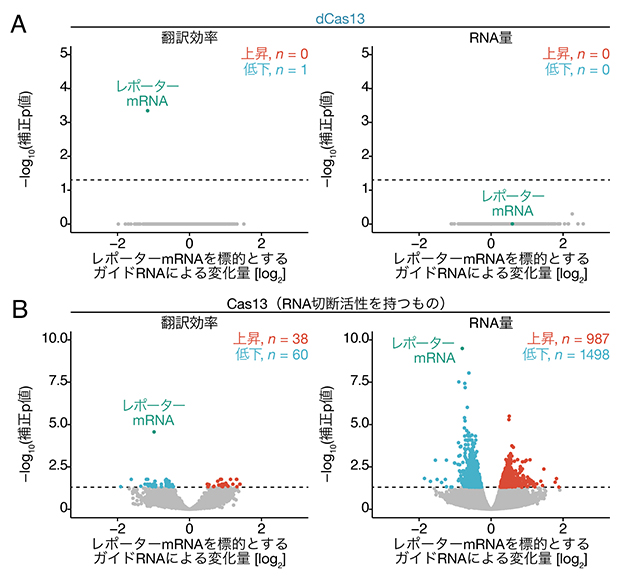
図3 リボソームプロファイリングによるCRISPRδの特異性の検証
(A)dCas13とレポーターmRNAを標的としたガイドRNAを細胞に導入し、リボソームプロファイリングを行った結果。レポーターmRNAの翻訳効率のみが有意に低下していた。補正p値=0.05を破線で示している。
(B)RNA切断活性を持つCas13で(A)と同様の実験を行った。レポーターmRNA以外にもさまざまな細胞内の遺伝子の翻訳効率やRNA量が有意に変動していた。
さらに、CRISPRδがさまざまなmRNAの翻訳を抑制できるか調べました。ウイルスなどではIRES[9]という配列を用いて、末端でなく途中からmRNAにリボソームを送り込んで翻訳を行っていますが、この特殊な翻訳もCRISPRδで抑制できました(図4A)。また、ある遺伝子に存在する繰り返し配列ではRAN翻訳[10]と呼ばれる特殊な翻訳によって3種類のタンパク質ができ、神経変性疾患の原因となることが示唆されていますが、CRISPRδは3種類のタンパク質の翻訳全てを抑制できることが分かりました(図4B)。最後に、内在の遺伝子についてもCRISPRδで翻訳を抑制できることを確かめました(図4C)。このとき、翻訳抑制に関わる4EHP[11]と呼ばれるタンパク質をdCas13タンパク質に融合させると、CRISPRδの効果が増強されることが分かりました。
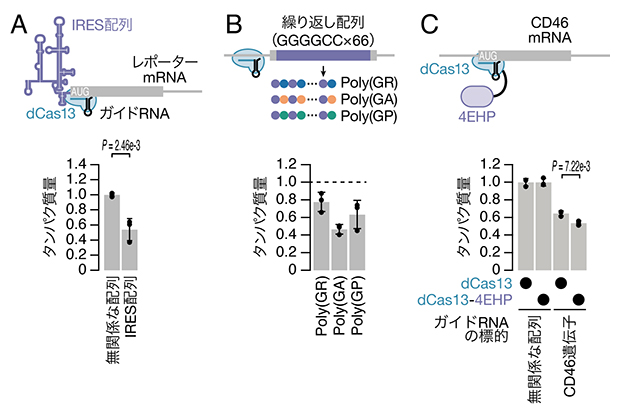
図4 さまざまなmRNAの翻訳に対するCRISPRδの効果
(A)IRES配列を持つレポーターmRNAに対するCRISPRδの効果。
(B)RAN翻訳レポーターmRNAに対するCRISPRδの効果。GGGGCCの繰り返し配列からは3種類のタンパク質が翻訳されるが、そのいずれもCRISPRδで抑制できる。
(C)内在の遺伝子CD46に対するCRISPRδの効果と4EHPを融合させることによる効果の増強。
各グラフは、無関係な配列のガイドRNAを導入した場合のタンパク質量を1とし、その変化量を示した。
今後の期待
本研究は翻訳を対象とした新しい遺伝子ノックダウン手法CRISPRδを開発し、非常に高い特異性で翻訳抑制できることを明らかにしました。遺伝子ノックダウン技術は遺伝子機能の理解に基づく基礎生物学の発展と、バイオテクノロジーや疾患治療などの応用的な側面にも貢献してきました。実際にRNA干渉を用いた医薬品もすでに実用化されています注)。今回、CRIPSRδはウイルスや神経変性疾患と関連する特殊な翻訳様式も抑制できることが分かりました。よって、ウイルス感染や神経変性疾患に対して、より特異性が高く副作用の低い治療を提供できる可能性も期待されます。
注)家族性アミロイドポリニューロパチーの治療薬であるパチシランなど。
補足説明
1.翻訳
生物学における翻訳とは、メッセンジャーRNA(mRNA)へコピーされた塩基配列をアミノ酸配列へ変換して、リボソームでアミノ酸を順番につなげてタンパク質を合成すること。
2.dCas13、CRISPR-Cas、Cas13
CRISPR-Casは細菌の持つ獲得免疫システムで、基本的にはガイドRNAと呼ばれる短いRNAとCasタンパク質が複合体を形成し、ガイドRNAと相補的な標的DNAまたは標的RNAに特異的に結合してその切断を行うもの。Cas13はⅥ型CRISPR-Casシステムを成すタンパク質で、RNAを切断する活性を持つ。Cas13に変異を導入してRNA切断活性を失わせたものをdCas13(catalytically-dead Cas13)と呼ぶ。CRISPR(クリスパー)はclustered regularly interspaced short palindromic repeats、CasはCRISPR-associatedの略。
3.メッセンジャーRNA(mRNA)
タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAのこと。リボソームによってそのコドンが読み取られ、タンパク質が合成される。
4.リボソーム
リボソームRNA(rRNA)とリボソームタンパク質から構成される超巨大複合体。リボソームはメッセンジャーRNA(mRNA)にコードされているコドンを読み取り、タンパク質を合成する。
5.RNA干渉
特定の配列を持つ短い二本鎖RNAを導入すると、それと相補的な配列を持つRNAが分解される現象。
6.レポーターmRNA
遺伝子の発現を簡便に確認するために用いられる、外来性のmRNA。代表例としてはルシフェラーゼや緑色蛍光タンパク質(GFP)が挙げられる。
7.次世代シーケンサー
DNAの塩基配列を決定するための装置。複数のDNA断片の塩基配列を同時並行で、高速・高精度に決定できる。
8.リボソームプロファイリング法
組織や細胞から翻訳装置であるリボソームを抽出し、リボソームと結合しているRNA配列を同定することで、どの遺伝子がどの程度の効率で翻訳されているかを知る解析法。リボソームは大きな複合体であるため、一定のmRNA領域を覆うように結合する。これらのリボソームとmRNAの複合体をRNA分解酵素で処理すると、リボソームが保護するmRNA断片だけが分解されずに回収される。
9.IRES
真核生物の通常の翻訳開始過程は、mRNAの5’末端のキャップ構造を翻訳開始因子群が認識し、リボソームを呼び込む。一方で、IRES配列を持つmRNAは、IRES RNAが立体構造を組むことで、直接リボソームを呼び込む。そのため、キャップ構造非依存的に翻訳過程を開始できるので、一部のRNAウイルスが、自身の遺伝子から効率よくタンパク質を生成するのに用いている。IRESは、Internal Ribosome Entry Siteの略。
10.RAN翻訳
特定の長い繰り返し配列によって生じる非典型的な翻訳開始機構。通常開始コドンはAUGだがRAN翻訳ではそれ以外の配列から開始され、3種類の繰り返しアミノ酸配列を持つタンパク質が翻訳される。RANは、Repeat-associated Non-AUGの略。
11.4EHP
mRNAの末端に結合する翻訳開始因子eIF4Eによく似た翻訳抑制を行うタンパク質。mRNAの5’末端のキャップ構造に結合する能力を持ち、eIF4EとmRNAの結合を阻害することで翻訳抑制する。4EHPは、eIF4E-homologous proteinの略。
研究支援
本研究は、理化学研究所運営費交付金RIKEN Pioneering Projects「Biology of Intracellular Environments(研究分担者:岩崎信太郎)」、国際プログラム・アソシエイト(Antonios Apostropoulos)で実施し、日本学術振興会(JSPS)科学研究費助成事業学術変革領域研究(A)「APEX-Ribo-Seq: 近傍標識による非典型局所翻訳の網羅解析(研究代表者:七野悠一)」「Heroタンパク質の動作機構解明と神経変性疾患への適用(研究分担者:築地仁美)」、同学術変革領域研究(B)「新規Disome-Seq法: パラメトリックなリボソーム渋滞の網羅的探索(研究代表者:岩崎信太郎)」「柔軟な神経らしさを作り出すパラメトリック翻訳制御の解明(研究代表者:池内与志穂)」、同基盤研究(B)「ミトコンドリア特異的リボソームプロファイリング法によるオルガネラ翻訳網羅解析(研究代表者:岩崎信太郎)」、同基盤研究(C)「細胞内顆粒P-bodyによるmRNAの空間的制御を介した細胞周期調節機構(研究代表者:七野悠一)」「RNA結合タンパク質量の増減による、筋萎縮性側索硬化症ALSの治療法開発(研究代表者:築地仁美)」、同特別研究員奨励費「青色光適応メカニズムから概日的遺伝子発現制御の意義を理解する(研究代表者:河本尚大)」、日本医療研究開発機構革新的先端研究開発支援事業(AMED-CREST)「神経変性疾患におけるアグリゲーションと翻訳の陰陽(研究代表者:岩崎信太郎、研究分担者:池内与志穂、築地仁美)」、同革新的先端研究開発支援事業(PRIME)「非膜性オルガネラによるRNA時空間制御を介したDNA損傷応答(研究代表者:七野悠一)」、生命創成探求センター先端共創プラットフォーム「オルガネラの時空間アトラス編纂(研究分担者:七野悠一)」による助成、および理化学研究所脳神経科学研究センター生体物質分析支援ユニットの支援を受けて行われました。
原論文情報
Antonios Apostolopoulos, Naohiro Kawamoto, Siu Yu A. Chow, Hitomi Tsuiji, Yoshiho Ikeuchi, Yuichi Shichino, Shintaro Iwasaki, “dCas13-mediated translational repression for accurate gene silencing in mammalian cells”, Nature Communications, 10.1038/s41467-024-46412-7
発表者
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(イワサキ・シンタロウ)
研究員 七野 悠一(シチノ・ユウイチ)
学振特別研究員PD 河本 尚大(カワモト・ナオヒロ)
国際プログラム・アソシエイト(研究当時)アントニオス・アポストロプロス(Antonios Apostropoulos)
(現 研究生、東京大学 生産技術研究所 特任研究員)
東京大学 生産技術研究所
准教授 池内 与志穂(イケウチ・ヨシホ)
特任助教 周 小余(チャウ・シウユ)
愛知学院大学 薬学部
教授 築地 仁美(ツイジ・ヒトミ)
報道担当
理化学研究所 広報室 報道担当
東京大学 生産技術研究所 広報室
愛知学院大学 総務部総務課