2024-03-28 東京大学
発表のポイント
◆リボソーム上でタンパク質が合成される際に、tRNAの塩基修飾がmRNAの遺伝暗号を解読する様子を、クライオ電子顕微鏡を用いて可視化することに成功しました。
◆tRNAに特徴的なシチジン修飾がリボソーム上でmRNAを正しく認識し、効率的にタンパク質を合成するしくみが明らかとなりました。
◆遺伝暗号の解読機構という生命の基本原理を解き明かした点で画期的な成果です。また将来的に合成生物学やtRNA創薬にも応用が期待されます。
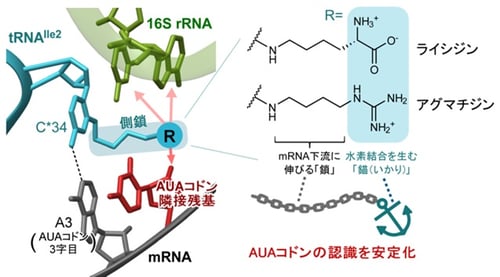
長い側鎖を持つtRNAのシチジン修飾がリボソーム上でmRNAを認識する
発表概要
東京大学大学院工学系研究科の秋山奈穂大学院生、鈴木勉教授の研究グループは、理化学研究所生命機能科学研究センターの石黒健介訪問研究員(研究当時、現:客員研究員/東京大学特任助教)、横山武司研究員(研究当時、現:東北大学助教)、白水美香子チームリーダーらとの共同で、クライオ電子顕微鏡(注1)を用いた構造解析により、リボソーム(注2)上でtRNA(注3)修飾がAUAコドン(注4)を解読するしくみを明らかにしました。
tRNAにはさまざまな化学修飾が含まれており、これらはタンパク質合成を行うために重要な役割を担っています。特に、アンチコドン領域に見られる修飾は、tRNAがmRNA上のコドンを読み取る能力を付与することで、正確かつ効率的なタンパク質合成を可能にしています。
細菌やアーキア(古細菌)では、AUAコドンをイソロイシン(Ile)に解読するtRNA(tRNAIle2)のアンチコドン1字目(34位)に特徴的なシチジン修飾(注5)を持っています。細菌のtRNAIle2はライシジン(L)(図1、2)、アーキアのtRNAIle2にはアグマチジン(agm2C)(図1、2)と呼ばれるシチジン修飾を用いています。Lとagm2Cはいずれも長い側鎖の末端に塩基性の官能基を持つ特徴的な化学構造を持ちますが、AUAコドンを解読する際にこれらがどのような役割を担うかは明らかになっていませんでした。

長い側鎖を持つtRNAのシチジン修飾がリボソーム上でmRNAを認識する
本研究では、リボソーム上で細菌およびアーキアのtRNAIle2がAUAコドンを解読する様子を、クライオ電子顕微鏡を用い、高分解能な構造解析で明らかにしました。Lとagm2Cのシトシン環は、同一の配向でAUAコドンの3字目のアデニン塩基と1本の水素結合を形成することがわかりました(図1)。また、Lとagm2Cの長い修飾側鎖がmRNAの下流に向かって伸び、末端の塩基性官能基が、AUAコドンの隣の残基と水素結合する様子が観察されました(図1)。実際に、mRNAの変異体を用いた実験で、この水素結合が形成されないと、AUAコドンの認識効率が低下することが判明しました。
さまざまなtRNA修飾がタンパク質合成の精度や効率に重要であることは知られていますが、具体的にどのようなしくみでその機能が発揮されるかは、よくわかっていませんでした。本研究は、細胞の生育に必須なシチジン修飾が、どのようにコドン解読を行うかを、分子レベルで解き明かしました。特に、tRNA修飾の側鎖が、mRNAのコドン以外の残基と相互作用するという発見は、tRNA修飾の全く新しい機能であり、生命科学の基本原理を解き明かした成果であると言えます。
発表内容
〈研究の背景〉
mRNAの塩基配列をタンパク質のアミノ酸配列に変換する「翻訳」は、すべての生物に必須の過程です。翻訳の精度は、リボソームにおいてtRNAがmRNA上のコドンを如何に正しく認識するかに大きく依存しています。tRNAにおいてコドンの読み取りを行う3文字の塩基配列はアンチコドンと呼ばれ、さまざまな化学修飾を受けています。特にアンチコドン1字目(34位)のtRNA修飾は、コドンとアンチコドンの相互作用を制御することにより、tRNAのコドン認識能を決定づけています(図2)。さらに、アンチコドンの3’側の隣接部位である37位にも複雑な修飾が見られ、コドン-アンチコドン対合を安定化する働きが知られています(図2)。これまでに、さまざまな生物から多種多様なtRNA修飾が見つかっていますが、それらがいったいどのようにコドンとアンチコドンの相互作用に関与するかについては、理解が進んでいないのが現状です。
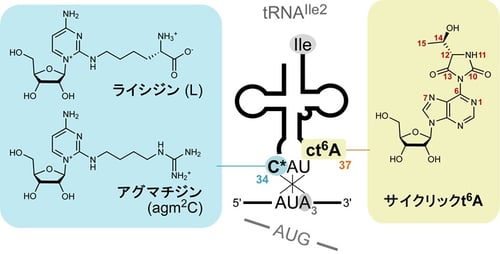
図2:AUAコドンを読むtRNAIle2に存在するアンチコドン修飾
遺伝暗号の基本的なルールとして、3字目がアデニンまたはグアニンのコドン(NNAまたはNNG)は34位にウリジン修飾を持つtRNAによってまとめて解読され、同じアミノ酸に翻訳されます。しかし例外的に、AUAとAUGコドンは異なるtRNAによって別々に解読され、それぞれIleとメチオニン(Met)に翻訳されるため、これら2つのコドンは遺伝暗号の中でも最も読みわけが難しいことが知られています。そこで、生物はtRNA修飾を用いて、AUAとAUGを正確に読み分けるシステムを築いています。
細菌およびアーキアなど原核生物は、AUAコドンを専属で読むtRNAIle2を持ち、その34位にはシチジン修飾(C*)が存在します(図2)。細菌のtRNAIle2は、リジンがシトシン2位に結合したLを持ち、アーキアtRNAIle2はアルギニン代謝物であるアグマチンがシトシン2位に結合したagm2Cを持ちます(図2)。Lとagm2Cは、いずれもシトシン環から長い側鎖が伸び、その末端に極性官能基があるという点で化学的性質が類似しています。ところが、これらは全く異なる種類のtRNA修飾酵素によって導入されることが知られ、生命の共通祖先から細菌とアーキアが分岐した後で、収しゅう斂れん 進化(注6)によって獲得されたtRNA修飾であると考えられています。未修飾のtRNAIle2前駆体はメチオニンを受容し、かつCAUアンチコドンを持つため、AUGコドンを認識してメチオニンに翻訳し、あたかもメチオニンtRNAのように振舞います。tRNAIle2は修飾されたのちに、イソロイシンを受容するようになり、はじめてAUAコドンを認識できるようになります。したがって、Lまたはagm2C修飾は、tRNAIle2のアミノ酸受容能とコドン解読能の2つの性質を一度にスイッチさせる重要な働きがあります。Lやagm2Cの修飾酵素はいずれも必須遺伝子にコードされていることから、これらのtRNA修飾はAUAコドンの解読に必須であり、生命の維持に不可欠であることが知られています。非常に重要なtRNA修飾であるにも関わらず、Lやagm2CがどのようなしくみでAUAコドンを解読するかについては、きちんと理解されていませんでした。
また、細菌や真菌、植物などのtRNAIle2には、アンチコドンに隣接する37位にサイクリックt6A(ct6A; cyclic N6-threonylcarbamoyladenosine)という、別のtRNA修飾が見られます(図2)。ct6Aは化学的に不安定ではあるものの、細胞内では高い割合でtRNAに導入されています。これまでに、ct6Aはコドン認識効率を向上させるという報告がありますが、そのしくみについては不明のままでした。また、ct6Aは嵩かさ高いだかい ヒダントイン環(注7)を含んでおり、この特徴的な化学構造がどのようにコドン解読に関わるのか、という興味深い課題がありました。
〈研究の内容〉
本研究では、大腸菌およびアーキアの一種であるHaloarcula marismortuiからtRNAIle2を単離精製し、AUAコドンを含むmRNAおよびリボソームと複合体を形成させ、クライオ電子顕微鏡(300kV)により2.3~2.7Å(オングストローム、1Åは0.1ナノメートル)の高分解能で構造解析を行いました。リボソームのA部位(注8)において、Lとagm2Cのシトシン環はいずれも、AUAコドン3字目のアデニン(A3)と1本の水素結合を介したC-A塩基対を形成することがわかりました(図3A)。この対合様式においては、Lまたはagm2Cの修飾側鎖が、AUGコドン3字目のグアニン(G3)と空間的にぶつかるため、AUGコドンの誤認識を妨げていると考えられます。さらに、Lおよびagm2Cの修飾側鎖はmRNAの下流(3’側)に向かって伸び、その末端の極性官能基がAUAコドンに隣接する残基のリボース2’-ヒドロキシ基と追加的に水素結合を形成する様子が観察できました(図3A)。さらにagm2Cは末端のグアニジノ基を介してA部位周辺のリボソームRNA(rRNA)とも水素結合をつくり、コドン-アンチコドン対合をさらに安定化していることが示唆されました(図3A)。これらの相互作用は、C-A塩基対には1本しか水素結合がないため、この弱い対合を補強する役割があると考えられます。また、細菌とアーキアでAUAコドンの認識機構が保存されていることも明らかとなりました。
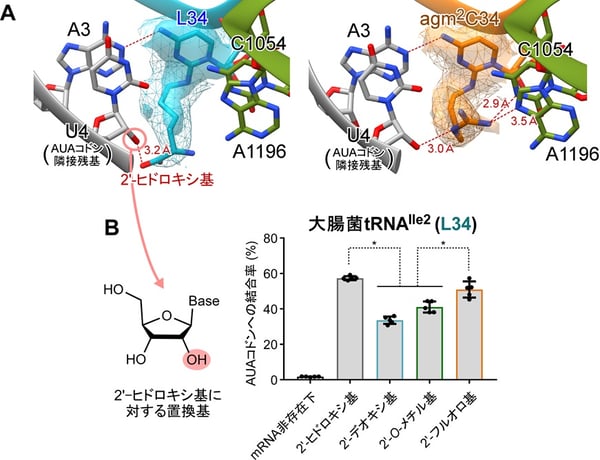
図3:シチジン修飾による効率的なAUAコドン認識のしくみ
(A)AUAコドンを認識するライシジン(L34;左)、アグマチジン(agm2C34;右)の構造。
(B)大腸菌tRNAIle2のAUAコドン結合効率。AUAコドンに隣接するmRNA 4字目の2′-ヒドロキシ基に対し置換を加えている。データは平均値±標準偏差で示した(n=5)。*p<0.01(two-tailed Student’s t-test)
Lおよびagm2Cの修飾側鎖とmRNAの相互作用が、実際にAUAコドン認識に影響を与えるかを調べるため、AUAコドンの3’隣接残基を置換した化学合成mRNAを用いて、リボソーム上におけるtRNAIle2のAUAコドン結合効率を評価しました(図3B)。3’隣接残基のリボースに2’-デオキシ基および2’-O-メチル基への置換を加えると、tRNAIle2のAUAコドンへの結合率が顕著に低下しました(図3B)。一方で、同じ位置を2’-フルオロ基に置換すると天然のmRNA同様に、AUAコドンによく結合することがわかりました(図3B)。2’-デオキシ基や2’-O-メチル基は極性側鎖との水素結合を阻害し、2’-フルオロ基は水素結合のアクセプターとして働くので、極性側鎖との水素結合が可能になると考えられます。これらの結果は、シチジン修飾の極性側鎖とコドンに隣接するmRNA残基との相互作用によって、tRNAによるAUAコドンの解読が効率よく行われることを強く示唆します。シチジン修飾は生命に必須のtRNA修飾でありながら、AUAコドンの解読に関してどのような役割を果たしているかわかっていませんでした。Lやagm2Cのシチジン修飾において、mRNAの下流に手を伸ばす炭素鎖部分を「鎖」、周囲のmRNAやrRNAと水素結合を作る末端の極性基を「錨(いかり)」に例えると、tRNAがリボソームに錨を下ろして、AUAコドンを認識しているように捉えることができます(図1)。このようなtRNA修飾の機能は前例がなく、従来の概念を覆す発見です。
さらに、今回の構造解析によって大腸菌tRNAIle2の37位に存在するct6A修飾を可視化することにも成功しました。ct6Aのヒダントイン環がアデニン塩基部分とおおよそ同一平面に配置された構造をとることがわかりました(図4A)。この結果から、ct6Aは、嵩高いヒダントイン環をうまくアンチコドンループ内に収め周辺の塩基(A38など)とスタッキング効果(注9)を生むことにより(図4B)、コドン-アンチコドン相互作用を安定化させる効果があると考えられます。
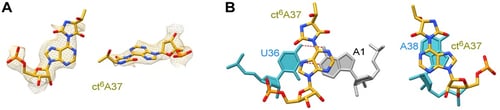
図4:37位のct6A修飾の構造
(A)ct6A37の構造。ヒダントイン環はアデニン塩基とおおよそ同一平面に配置されている。
(B)ct6A37と周辺のU36-A1(mRNA)(左)やA38(右)とのスタッキング。
〈今後の展望〉
本研究の成果は、遺伝暗号の中でも読みわけが難しいAUAコドンの解読機構の謎に迫ったものであり、タンパク質合成の基本的なしくみやtRNA修飾の機能を分子レベルで明らかにした成果と言えます。今後は、ヒトや他の生物種に見られるtRNA修飾についても、同様の解析を行うことで、遺伝暗号解読機構の全体像を理解することが重要です。
発表者
東京大学大学院工学系研究科 化学生命工学専攻
秋山 奈穂 博士課程/日本学術振興会特別研究員
鈴木 勉 教授
理化学研究所 生命機能科学研究センター タンパク質機能・構造研究チーム
白水 美香子 チームリーダー
論文情報
〈雑誌〉Nature Structural & Molecular Biology
〈題名〉Structural insights into the decoding capability of isoleucine tRNAs with lysidine and agmatidine
〈著者〉Naho Akiyama, Kensuke Ishiguro, Takeshi Yokoyama, Kenjyo Miyauchi, Asuteka Nagao, Mikako Shirouzu, *Tsutomu Suzuki
〈DOI〉10.1038/s41594-024-01238-1
研究助成
本研究は、日本学術振興会JSPSの特別研究員奨励費「原核生物におけるAUAコドン解読の分子基盤」(代表:秋山奈穂、23KJ0409)、基盤研究(S)「RNAエピジェネティックスと高次生命現象」(代表:鈴木勉、26220205)、基盤研究(S)「RNA修飾の変動と生命現象」(代表:鈴木勉、18H05272)、新学術領域研究 研究領域提案型「ncRNAのケミカルタクソノミ」(代表:鈴木勉、26113003)、および科学技術振興機構(JST)の戦略的創造研究推進事業(ERATO)「鈴木RNA修飾生命機能プロジェクト」(研究総括:鈴木勉、JPMJER2002)などの支援を受けて実施されました。
用語解説
(注1)クライオ電子顕微鏡
生体分子の試料に低温下(約-200℃)で電子線を照射し、その構造を観察できる電子顕微鏡。試料を水溶液中で瞬間凍結することで、生体内に近い環境で目的分子の構造解析を行うことができる。
(注2)リボソーム
RNAとタンパク質からなる複合体でタンパク質合成の場である。大小2つのサブユニットからなり、大サブユニットはペプチジル転移反応を、小サブユニットはmRNAとtRNA間のコドンーアンチコドン対合を監視するといった重要な役割をもつ。
(注3)tRNA
Transfer RNA(転移RNA)。タンパク質合成において、コドンとアミノ酸を対応させるアダプター分子として働く。70〜90塩基長の短い一本鎖RNAで、二次構造としては特徴的なクローバー葉様構造をとり、それが折りたたまれてL字型の立体構造を取る。tRNAは3’末端に対応するアミノ酸を受容し、20種類のアミノ酸に対応して異なるtRNA種が存在する。tRNAはコドンと対合するアンチコドンを持ち、リボソーム上でmRNA(伝令RNA)上のコドンと結合することで、対応するアミノ酸を伸長中のタンパク質へと導入する。
(注4)AUAコドン
コドンとはRNAの4種類の塩基(A,U,G,C)が三つ組みになって構成される遺伝暗号の単位であり、4×4×4=64通りのコドンが存在する。各コドンはタンパク質を構成する20種類のアミノ酸およびタンパク質合成の終結を指定している。AUAコドンはイソロイシンを指定し、後出するAUGコドンはメチオニンを指定する。
(注5)シチジン修飾
シチジンとはRNAを構成する4種類のヌクレオシドの1つであり、修飾されたシチジンのことを指す。
(注6)収斂進化
異なる系統の生物や生体分子が、類似した形態に進化する現象。例えば、哺乳類のイルカと魚類のサメは、水中での生活に適応するために似通った形の尾ひれを獲得している。
(注7)ヒダントイン環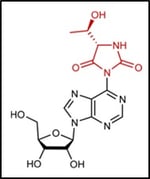
複素環式構造の一種。ct6A修飾に含まれている分子骨格である。
(注8)A部位
リボソームにおいてtRNAが結合する3つの部位の1つ。A部位にはアミノアシルtRNAが結合し、ここでtRNAのアンチコドンがコドンを認識し、遺伝暗号の解読が行われる。
(注9)スタッキング効果
2つの芳香環が積み重なるように配置するときに生じる相互作用。芳香環のπ電子同士が重なり合うことで分子間力を生じ、その配置を安定化する効果がある。
プレスリリース本文:PDFファイル
Nature Structural & Molecular Biology:https://www.nature.com/articles/s41594-024-01238-1


