2024-09-20 理化学研究所
理化学研究所(理研)生命機能科学研究センター 転写制御構造生物学研究チームの関根 俊一 チームリーダー、柳沢 達男 研究員、村山 祐子 上級テクニカルスタッフらの研究チームは、RNAポリメラーゼⅡ(RNAPⅡ)[1]に転写終結因子「Rat1-Rai1[2]」が結合した構造を解明し、真核生物における転写終結反応の新たなモデルを提唱しました。
本研究成果は、転写[1]の最終段階である転写終結[2]のメカニズムの理解に貢献し、がんやウイルス感染症など転写が関わる疾病メカニズム解明や生命進化の理解につながると期待されます。
RNAPⅡは真核生物[3]において遺伝情報の転写をつかさどる、巨大なタンパク質複合体です。転写を終えたRNAPⅡが鋳型DNAや転写されたRNAから解離して転写装置を解体する転写の最終段階が転写終結です。真核生物に広く保存された転写終結因子Rat1-Rai1はRNAPⅡの転写終結を促進するタンパク質複合体であることが報告されていましたが、その詳しい仕組みは不明でした。
今回、研究チームは、クライオ電子顕微鏡[4]を用いた構造解析により、Rat1-Rai1がRNAPⅡに結合して転写の終結反応をつかさどる姿を初めて捉えました。その結果、RNAPⅡ-Rat1-Rai1複合体は転写終結の「前」と「後」で全く異なる複合体構造をとることが判明し、転写を完了させた後もRat1-Rai1はRNAPⅡを保護する機能を持つことが示唆されました。
本研究は、科学雑誌『Nature Communications』オンライン版(9月8日付)に掲載されました。
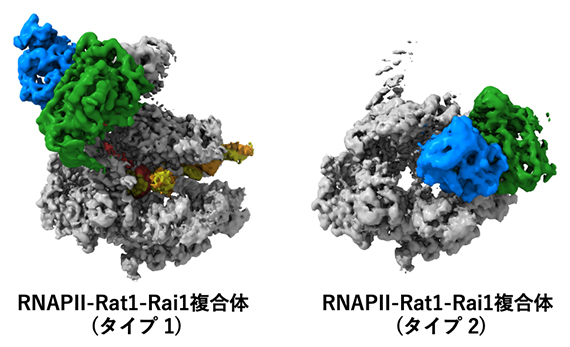
転写終結の前(タイプ1)と後(タイプ2)のRNAPⅡ-Rat1-Rai1複合体構造
背景
RNAポリメラーゼ(RNAP)[1]は巨大なタンパク質複合体(分子量約40~50万)で、DNA上の遺伝情報をRNAへと転写する役割を担っています。真核生物に存在する3種類のRNAPのうちの一つ、RNAポリメラーゼⅡ(RNAPⅡ)は鋳型DNA上の特定の領域から読み取りを開始し、転写伸長複合体[5]となったRNAPⅡがDNAに沿って伝令RNA(mRNA)を合成、最終的にRNAPⅡがDNAから解離して転写が完了します。転写の最終段階は「転写終結」と呼ばれ、遺伝子の読み取りを適切に終わらせることで遺伝子発現を制御し、mRNAの長さの調節やゲノム安定性に寄与する重要なステップです。真核生物の転写終結は22~24種類ものタンパク質が関わる多段階の反応であることが知られており、5’→3’エキソリボヌクレアーゼ[2]活性を持つRNA分解酵素Rat1と、その補助因子であるRai1の複合体「Rat1-Rai1」もその一つです。
真核生物の遺伝子は、タンパク質の情報を持つ領域の下流に「ポリAシグナル[6]」と呼ばれる保存された6塩基配列を持ちます。RNAPⅡがポリAシグナルを通過してポリAシグナルを含むmRNAが転写されると、その配列の10~30塩基下流でmRNAが切断され、切断により生じたmRNAの3’末端に50~200塩基のポリAが付加されます(図1A、B、C)。一方、切断によって転写伸長複合体に残されたRNAの5’末端には、Rat1-Rai1が結合してRNAを5’末端側から分解していきます。Rat1-Rai1は転写中のRNAPⅡに追い付いて「魚雷」のように激突し、RNAPⅡをDNA/RNAから解離させると考えられています(図1D)。しかしながら、転写終結に至る複合体の構造情報を含め、Rat1-Rai1による転写終結の詳細な分子メカニズムについてはよく分かっていませんでした。そこで本研究では、クライオ電子顕微鏡を用いて、Rat1-Rai1が結合したRNAPⅡ複合体の構造解析を試みました。
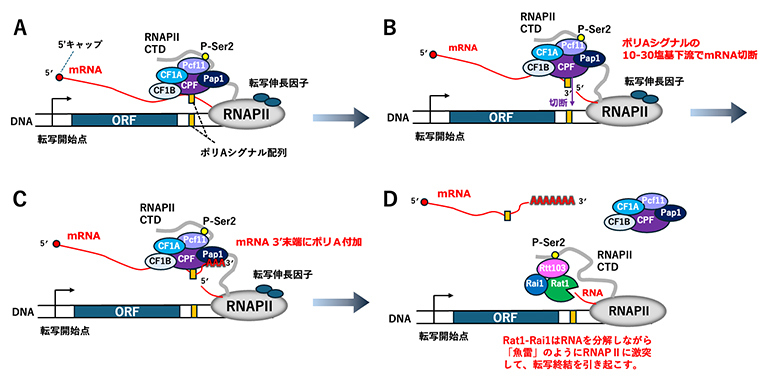
図1 転写終結の概略図
出芽酵母において、mRNAを転写したRNAPⅡの転写終結に至る各ステップを模式図で示す。ORFはアミノ酸配列をコードする領域(Open reading frame)、黄色はDNA上のポリAシグナルと、mRNAに転写されたポリAシグナルを示す。
(A)転写伸長複合体がポリAシグナルを超えて転写を続けている。切断・ポリA化因子複合体(CPF)、および切断因子(CF)複合体の構成要素であるCF1A、CF1Bは、mRNAの3’非翻訳領域のポリAシグナルを認識して結合する。mRNAの5’末端はキャップ構造(7メチルGppp)構造を持つ。
(B)mRNAがポリAシグナルの10~30塩基下流(ポリA付加サイト)で切断される。転写伸長複合体に残されたRNA断片の5’末端には、キャップ構造は存在しない。
(C)mRNAの3’末端に、ポリAポリメラーゼPap1によって出芽酵母では約50塩基のポリAが付加される(ヒトでは200~250塩基)。
(D)Rtt103の介在により、Rat1-Rai1がRNAPⅡに動員される。mRNA切断後に残ったRNAは、Rat1-Rai1によって5’末端側から分解される。Rat1-Rai1は「魚雷」のようにRNAPⅡに激突して転写終結を引き起こす。本研究では、この過程を捉えた。
研究手法と成果
研究チームは、まずRat1-Rai1による転写終結を試験管内で再現するための実験系を構築しました。RNAを分解する活性を持つRat-Rai1は、転写伸長複合体から送り出されたRNAを介して転写中のRNAPⅡに動員されるはずです。そこでRat1-Rai1をRNAPⅡに結合させるため、長さの異なる5種類のRNA(31、26、22、20、17塩基長)を用意しました。これらのRNAと、鋳型となるDNAおよび酵母細胞から取り出したRNAPⅡを混ぜることで、転写伸長複合体を調製しました(図2A上)。これらのRNAPⅡの転写伸長複合体にRat1、Rai1を加えた試料を用いて、クライオ電子顕微鏡による構造解析を行いました。その結果、RNAが31、26、22、20塩基の条件では、大きく分けて2種類(タイプ1、タイプ2)の複合体が存在することが分かりました(図2A下、B)。一方、RNAが17塩基、およびDNAとRNAが存在しない条件ではタイプ2の複合体しか観察されませんでした(図2A下、B)。
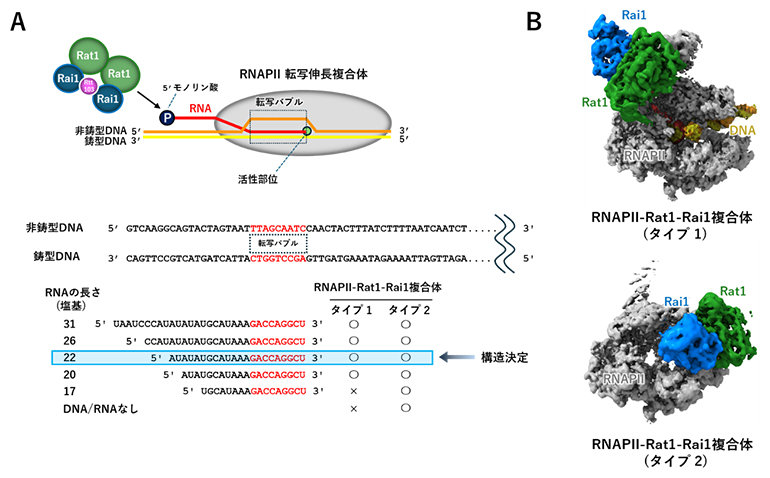
図2 試験管内で再構成した転写終結
本研究では、2本鎖DNAの一部に対合しない配列を持たせることで、RNAPⅡ内での転写の際に二重らせん構造が部分的に解かれた構造(転写バブル)を疑似的に再現した。この2本鎖DNAと転写バブル内の鋳型DNAに対合する31、26、22、20、17塩基のRNA、および酵母細胞(メタノール資化酵母)から取り出したRNAPⅡを混ぜることで、転写伸長複合体を調製した。RNAの5’末端は、切断された状況としてキャップ構造を持たせていない(5’モノリン酸化RNA)。
(A)上:RNAPⅡ転写伸長複合体、RNAPⅡ-Rat1-Rai1複合体の調製と本研究で使用したDNA、RNAの模式図。下:◯はクライオ電子顕微鏡解析で検出された複合体、×は検出されなかった複合体。Rat1-Rai1は5’モノリン酸化RNAを認識して結合する。2本鎖DNAは3’側の35塩基を省略して表記した。
(B)RNAPⅡ-Rat1-Rai1複合体(タイプ1およびタイプ2)の構造。22塩基RNAを用いてクライオ電子顕微鏡データを取得し、タイプ1およびタイプ2複合体の構造を決定した。
タイプ1複合体では、Rat1-Rai1がRNAPⅡ転写伸長複合体のRNA送出トンネル[5]を覆うように結合し、転写されたRNAがこのトンネルから送り出される経路を大きく変えられてRat1の活性部位ポケット(RNA分解活性の中心部)へと誘導されていました(図3A左、B、C)。またタイプ1複合体が存在する条件では、DNAとRNAが含まれないタイプ1様の複合体も見られました(タイプ1b複合体)(図3A右)。一方、タイプ2複合体はタイプ1とは全く異なり、RNAPⅡ内でDNAが結合するための溝(DNA結合クレフト、以下クレフト)が大きく開いて、そこにRat1-Rai1が結合していました(図2B下、図4)。
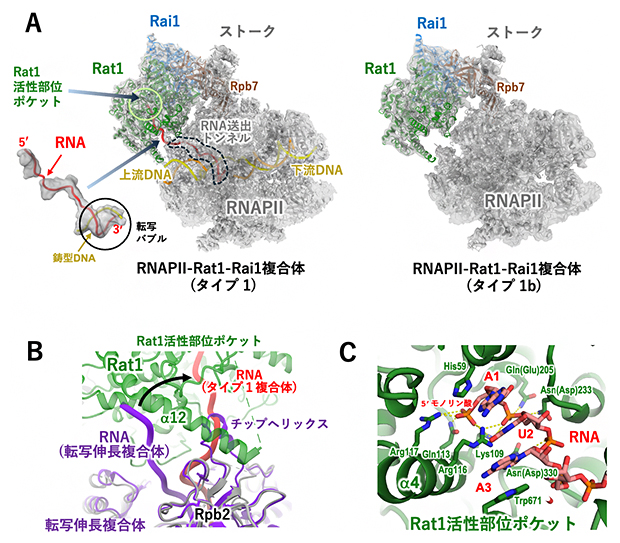
図3 Rat1-Rai1とRNAPⅡの結合―タイプ1
(A)Rat1-Rai1と結合したRNAPⅡ複合体の構造。タイプ1はDNAとRNAを含み、タイプ1bは含まない。クライオ電子顕微鏡で得られた構造に、タンパク質の骨格を示すリボンモデルを重ね、RNA送出トンネル内のRNAとRat1活性部位を透過して示した。Rat1-Rai1は、RNAPⅡのストークと呼ばれる部分構造のサブユニット(Rpb7)と相互作用している。参考として、転写バブルに結合した状態のRNAの全体像を別に表示させた。
(B)RNAPⅡ転写伸長複合体からタイプ1複合体への構造変化。RNAPⅡから送り出されたRNA(赤)が、Rat1の活性部位ポケットへと大きく向きを変えていることが分かる。
(C)5’モノリン酸化RNAが結合したRat1活性部位ポケットの構造。5’リン酸と最初の3塩基(AUA)がRat1の各アミノ酸残基によって認識されている様子を示す。アミノ酸残基の略号は以下の通り。Asn:アスパラギン、Asp:アスパラギン酸、Arg:アルギニン、Gln:グルタミン、Glu:グルタミン酸、His:ヒスチジン、Lys:リジン、Trp:トリプトファン。
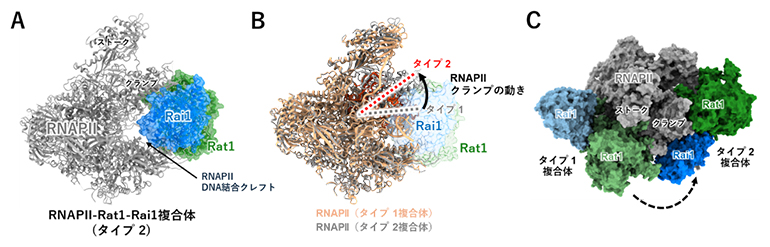
図4 Rat1-Rai1とRNAPⅡの結合―タイプ2
(A)RNAPⅡはリボンモデルで、Rat1-Rai1はリボンモデルに表面モデルを重ねて表示した。
(B)タイプ1複合体とタイプ2複合体を、RNAPⅡを基準にして重ね合わせた。タイプ2複合体では、RNAPⅡの開閉部分(クランプ)が大きく開いてできたクレフト(溝)にRat1-Rai1が結合している。
(C)(B)の重ね合わせ構造を表面モデルで表示し、別の方向から見たもの。タイプ1複合体とタイプ2複合体のRat1-Rai1はRNAPⅡ上では全く違う位置に結合していることが分かる。
本研究で得られた構造から、Rat1-Rai1が関わるmRNA転写終結のメカニズムを次のように説明できます(図1、図5)。
(ⅰ)ポリAシグナル下流でmRNAの切断が起こると、Rat1がRNAの5’末端を捕捉し、転写を続けるRNAPⅡを追跡してRNAの分解を開始する。
(ⅱ)Rat1がRNAを20~22塩基の長さまで分解すると、Rat1-Rai1はRNAPⅡと接触する。さらにRat1がRNAを分解していくと、Rat1とRNAPⅡ間のRNAのひずみが増大し、転写バブル内における対合したDNA/RNAが不安定化する。
(ⅲ)転写バブルの崩壊とDNA/RNAの放出が引き起こされる。
(ⅳ)Rat1-Rai1は、DNA/RNAが放出されて空になったクレフトに再配置されるか、あるいはDNA/RNAがまだ残っている場合はクレフトに移動して、クレフトから完全にDNA/RNAを取り除く。
こうして転写が終結した後、クレフトに結合したRat1-Rai1は、次の転写ラウンドで転写開始複合体にリサイクルされるまでRNAPⅡを保護すると考えられます(図5)。
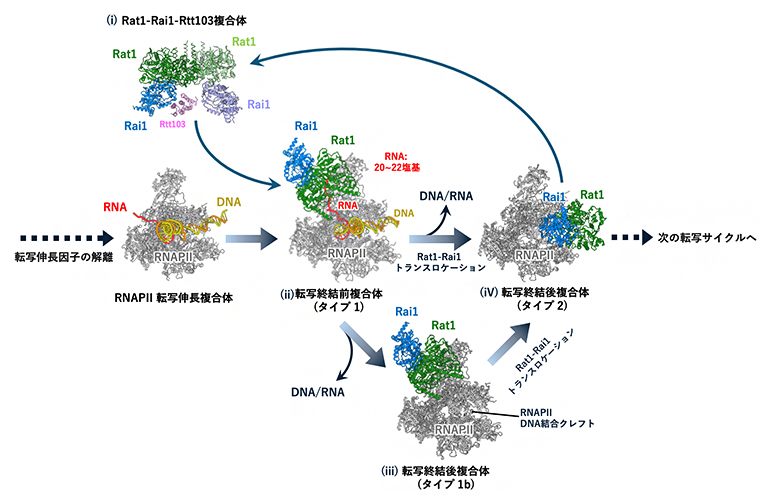
図5 Rat1-Rai1依存的転写終結のモデル
今回の解析で得られた複数の構造を、転写終結の各ステップに当てはめた新たなモデルを示す。
(ⅰ)RNAPⅡに結合する前のRat1-Rai1はヘテロ4量体、あるいはRat1-Rai1-Rtt103のヘテロ5量体複合体として存在しているが、RNAPⅡに結合する際にはRat1-Rai1ヘテロ2量体の形をとって、リン酸化されたRNAを捕捉し、「魚雷」のように5’末端側からRNAを分解していく。
(ⅱ)Rat1-Rai1がRNAを分解しながらRNAPⅡに追い付き、転写が終結する前の構造(タイプ1複合体)。
(ⅲ)タイプ1bは、タイプ1複合体の構造を保ったままDNA/RNAが解離した転写終結後の構造。この後、空になったクレフトにRat1-Rai1が再配置(トランスロケーション)されることで、タイプ2複合体(ⅳ)に移行する。
(ⅳ)タイプ2複合体は転写終結が完了した後、不必要な転写開始を抑えるための構造と考えられる。タイプ1からタイプ2への移行は、タイプ1b(ⅲ)を経由せず、直接タイプ1(ⅱ)からタイプ2(ⅳ)へと移行するパターンもあり得る(Rat1-Rai1がRNAを分解しながらクレフトに移動し、直接DNA/RNAを取り除く)。クレフトに結合したRat1-Rai1は、RNAPⅡが次の転写サイクルで転写開始複合体にリサイクルされるまでRNAPⅡを保護すると考えられる。
今後の期待
今回明らかになった複合体構造から、Rat1-Rai1を介したRNAPⅡによる転写の終結に関して新たな知見を得ることができました。Rat1とRai1はその発見から約30年以上にわたって研究されてきたタンパク質ですが、どのような結合様式でRNAPⅡや新生RNAに結合し、転写終結を引き起こすのかについてはよく分かっていませんでした。本研究は、Rat1-Rai1がRNAPⅡやRNAに結合し転写終結をつかさどる姿を分子レベルで初めて捉えたもので、転写終結という細胞の基本的なプロセスの理解に大きく貢献すると考えられます。驚くことに、Rat1による転写終結のメカニズムは、以前に研究チームが明らかにした細菌の転写終結因子Rhoによるもの注1)と類似性が見られ、Rat1とRhoは進化的にも機能的にも全く異なるにもかかわらず、原核生物から真核生物まで共通の仕組みで転写を終わらせていることが示唆されます。また、細胞はこれらのプロセスを状況に応じて調節しながら実行することでその機能を維持しており、転写が関連する疾病のメカニズム解明、さらには生命進化の理解にもつながると期待されます。
注1)2023年2月9日プレスリリース「遺伝子の読み取りを終わらせるメカニズム」
補足説明
1.RNAポリメラーゼⅡ(RNAPⅡ)、転写、RNAポリメラーゼ(RNAP)
DNA配列を鋳型にしてRNAが合成されることを転写と呼び、細胞内ではDNA依存性RNAポリメラーゼ(RNAポリメラーゼ、RNAP)により行われる。RNAPは複数のタンパク質(サブユニット)が集合してできた巨大な複合体(分子量約40~50万)で、細菌からヒトまで共通した”カニのはさみ”のような形をしている。およそ10塩基対のDNAをほどいて「転写バブル」を形成し、一方のDNA鎖を鋳型にしてRNAを合成する。このとき鋳型DNAのG(グアニン)はC(シトシン)、CはG、T(チミン)はA(アデニン)、AはU(ウラシル)と読み取られる。細菌などの原核生物はRNAPを1種類だけ持つのに対し、真核生物は3種類のRNAP(RNAPⅠ、RNAPⅡ、RNAPⅢ)を持ち、それぞれrRNA(リボソームRNA)、mRNA(伝令RNA)、tRNA(転移RNA)と5S rRNA(リボソームの大サブユニットの構成成分であるリボソームRNAの一つ)の合成を分担する。
2.Rat1-Rai1、転写終結、エキソリボヌクレアーゼ
転写終結は転写を終えたRNAPが鋳型DNAや新生RNAから解離し、転写装置が解体されること。Rat1(ヒトではXrn2)は、真核生物に広く保存された5’→3’型のRNA分解酵素(エキソリボヌクレアーゼ)で、Rai1(ヒトではDXO/DOM3Z)というRNAの5’脱キャッピング活性を持つタンパク質と複合体(Rat1-Rai1)を形成して働く。
3.真核生物
すべての生物は真核生物と、原核生物である古細菌と真正細菌の三つのグループに分類される。真核生物の特徴は細胞内に核を持ち、細胞のそれ以外の部分からは膜で仕切られていることである。核の中には遺伝情報が書き込まれたDNAが収められている。すべての動物と植物、カビ、キノコ、酵母などの真菌類、ゾウリムシやアメーバなどの原生生物は真核生物である。
4.クライオ電子顕微鏡
タンパク質などの生体試料を観察するために開発された電子顕微鏡。タンパク質などの試料を含んだ溶液を薄く展開し、液体エタン(-183~-160℃)中で急速凍結して試料をごく薄い氷の層に閉じ込めた上、液体窒素温度(-196℃)で電子顕微鏡により観察する。タンパク質試料を生理的な(細胞の状態に近い)条件で観察できる、低温のため電子線による試料の損傷が軽減される、といった利点がある。近年著しい発展を見せている構造解析技術であり、2017年のノーベル化学賞の対象ともなった。
5.転写伸長複合体、RNA送出トンネル
転写の過程においてRNAPは、転写伸長に必要なさまざまなタンパク質と結合した巨大な複合体となって機能する。このタンパク質複合体にさらに鋳型DNAと新生RNAが結合している状態が、細胞内の転写反応の真の姿と考えられ、これを転写伸長複合体と呼ぶ。関根チームリーダーらの先行研究注2)では、転写伸長複合体には、新生RNAの効率的な排出を促進する構造が存在することを解明し、これをRNA送出トンネルと名付けた。
注2)2017年8月4日プレスリリース「転写中のRNAポリメラーゼIIの構造を解明」
6.ポリAシグナル
真核生物では、多くのmRNAの3’末端にポリAと呼ばれるAMP(アデノシン一リン酸)のポリマーが付加されている。ポリAシグナルはmRNAの非翻訳領域にあるAAUAAA、AUUAAAなどの保存された配列で、ポリA化因子複合体や切断因子複合体により、このシグナル配列の10~30塩基下流でRNAが切断されポリAが付加される。
研究チーム
理化学研究所 生命機能科学研究センター 転写制御構造生物学研究チーム
チームリーダー 関根 俊一(セキネ・シュンイチ)
研究員 柳沢 達男(ヤナギサワ・タツオ)
上級テクニカルスタッフ 村山 祐子(ムラヤマ・ユウコ)
上級研究員 江原 晴彦(エハラ・ハルヒコ)
テクニカルスタッフⅠ 五島 美絵(ゴトウ・ミエ)
テクニカルスタッフⅠ 青木 真理(アオキ・マリ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(S)「転写と中核的な生命機能を結びつける高次複合体の構造基盤(研究代表者:関根俊一)」「クロマチンを背景とした転写の構造基盤(研究代表者:関根俊一)」による助成を受けて行われました。
原論文情報
Tatsuo Yanagisawa, Yuko Murayama, Haruhiko Ehara, Mie Goto, Mari Aoki, and Shun-ichi Sekine, “Structural basis of eukaryotic transcription termination by the Rat1 exonuclease complex”, Nature Communications, 10.1038/s41467-024-52157-0
発表者
理化学研究所
生命機能科学研究センター 転写制御構造生物学研究チーム
チームリーダー 関根 俊一(セキネ・シュンイチ)
研究員 柳沢 達男(ヤナギサワ・タツオ)
上級テクニカルスタッフ 村山 祐子(ムラヤマ・ユウコ)
報道担当
理化学研究所 広報室 報道担当