2018-10-09 理化学研究所,沖縄美ら島財団,海遊館,大阪市立大学,東京大学
要旨
理化学研究所(理研)生命機能科学研究センター分子配列比較解析ユニットの工樂樹洋ユニットリーダー、沖縄美ら島財団総合研究センターの佐藤圭一上席研究員(沖縄美ら海水族館副館長)、海遊館の西田清徳館長、大阪市立大学大学院理学研究科の小柳光正准教授、東京大学大気海洋研究所の兵藤晋教授らの共同研究グループ※は、「イヌザメ」「トラザメ」「ジンベエザメ」の全ゲノム配列を解析しました。
本研究成果から得られた高精度のゲノム情報は、脊椎動物の進化および海の生態系において独特の位置を占める「軟骨魚類」の発生や生態、また水族館などでの飼育・繁殖に関する多様な研究に貢献すると期待できます。
軟骨魚類のうち、サメ類とエイ類が属する「板鰓(ばんさい)類[1]」は、ゲノムサイズ[2]が大きいことなどが障壁となり、これまで、高精度な全ゲノム配列情報が得られていませんでした。今回、共同研究グループは、約1,200種知られる板鰓類のうち、卵生[3]で繁殖力の高いイヌザメとトラザメに着目し、全ゲノム配列を解読しました。また、現生で最大の魚類ジンベエザメについては、既に公開されていたゲノム配列をより高精度の情報に整えました。これらのゲノム情報に加えて、生体から採取した細胞のRNAデータを活用することで、形態形成や視覚などをつかさどる遺伝子群の進化を詳しく解析し、サメ類とヒトを含む他の脊椎動物との共通点と相違点を多数発見しました。
本研究は、英国のオンライン科学雑誌『Nature Ecology and Evolution』(10月8日付け:日本時間10月9日)に掲載されます。

図 今回全ゲノム解析したイヌザメ成魚、トラザメ幼魚、ジンベエザメ成魚(左より)
※共同研究グループ
理化学研究所 生命機能科学研究センター
分子配列比較解析ユニット
ユニットリーダー 工樂 樹洋(くらく しげひろ)
形態進化研究チーム
チームリーダー 倉谷 滋(くらたに しげる)
沖縄美ら島財団総合研究センター
上席研究員 佐藤 圭一(さとう けいいち)
(沖縄美ら海水族館 副館長)
海遊館
館長 西田 清徳(にしだ きよのり)
大阪市立大学 大学院理学研究科
生物地球系専攻 生体高分子機能学II研究室
教授 寺北 明久(てらきた あきひさ)
准教授 小柳 光正(こやなぎ みつまさ)
東京大学大気海洋研究所
海洋生命科学部門 生理学分野
教授 兵藤 晋(ひょうどう すすむ)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(C)課題「進化上保存されていない「若い」遺伝子の発生プログラムにおける役割を探る(研究代表者:工樂樹洋)」、JSPS科学研究費助成事業基盤研究(B)課題「川を遡上するオオメジロザメの広塩性に関する生理生態学的研究(研究代表者:兵藤晋、研究分担者:工樂樹洋、佐藤圭一)」、JSPS科学研究費助成事業基盤研究(B)課題「“抗利尿ホルモン”の起原と進化:新規受容体研究がもたらす新展開(研究代表者:兵藤晋、研究分担者:工樂樹洋)」、JSPS科学研究費助成事業挑戦的萌芽研究課題「単離腎ネフロン分節からのトランスクリプトーム:広塩性のマスター分子解明に向けて(研究代表者:兵藤晋、研究分担者:工樂樹洋)」による支援を受けて行われました。
背景
現存の「軟骨魚類」の共通祖先は、哺乳類の祖先を含む硬骨魚(硬骨脊椎動物)と約4億5,000万年前に分岐したと考えられています。軟骨魚類のうち、ギンザメ類[4]を除く「板鰓(ばんさい)類(サメ類とエイ類、図1)」は、サンゴ礁のような浅海から水深1,000メートルを超える深海に、そして極海から赤道に、1,200種余りが生息しています。一般的なサメのイメージである肉食性の大型種は、食物連鎖の上位として生態系で重要な地位を占めています。また、体長が20センチメートルほどにしか成長しない種から、「ジンベエザメ(Rhincodon typus)」のように体長が10メートルを超えるプランクトン食性の種まで存在し、繁殖様式についても卵生と胎生[3]があります。また近年では、寿命が約400年近い種の存在が話題になりました注1)。
これらの種間の系統関係や生息域などを調べるだけなら、ゲノムDNAの一部の情報を利用すればよいのですが、寿命を決める仕組みなど複雑な生命現象のメカニズムを分子レベルで理解するためには、それらの現象に関わる遺伝子群を網羅的に同定する必要があります。さらに、哺乳類では、多様なホルモン[5]によって、成長や繁殖に加え、食欲のコントロールや浸透圧調節など「恒常性」と呼ばれる体の活動を維持するシステムが制御されていますが、軟骨魚類のように脊椎動物の中で早い時期に分岐した生物でもこのような制御システムを持っているか否かについての包括的な知見はありません。これらの生命機能を体系的に調べる手がかりとなるのが、全ての遺伝子の情報を含む「全ゲノム」の塩基配列です。
これまで多様な生物の全ゲノム配列情報が公表されてきましたが、軟骨魚類のゲノム解析は、ゲノムサイズ[2]が小さい(約12億塩基対)「ゾウギンザメ」について行われたのみでした注2)。しかし、ゾウギンザメが属するギンザメ類の仲間は50種程度に過ぎないうえ、本来の「サメ」の系統とも異なっており(図1)、軟骨魚類の多様性を理解するための分子レベルの研究は手つかずのままでした。またゾウギンザメの生息域は南半球の一部地域に限られ、生体を確保することが難しいなどの理由で、得られたゲノム情報の生命科学研究への利用は狭い範囲にとどまっていました。
こういった軟骨魚類研究の現状を打破すべく、共同研究グループは、卵生で繁殖力の高いサメに着目しました。日本近海に生息し、以前から国内で研究に用いられてきた「トラザメ(Scyliorhinus torazame)」を解析対象として選んだほか、アジア・オセアニア海域に広く生息し、各地の水族館で飼育されている「イヌザメ(Chiloscyllium punctatum)」に新たに着目し、生体を研究に使用するための準備を整えてきました。孵化までの発生段階の詳細なモニタリングを行った先行研究では、温暖域に棲むイヌザメは、冷水域に棲むトラザメより約1.5倍速く成長することを明らかにしました注3)。この研究の過程で、理研生命機能科学研究センターと海遊館は、魚類をはじめとする野生動物の生命科学研究や教育普及などの取り組みを強化するために学術交流協定を締結しました。
注1)Nielsen J et al. 2016. Eye lens radiocarbon reveals centuries of longevity in the Greenland shark (Somniosus microcephalus). Science. 353(6300):702-4.
注2)Venkatesh B. et al. 2014. Elephant shark genome provides unique insights into gnathostome evolution. Nature. 505:174–179.
注3)Onimaru K. et al. 2018. A staging table for the embryonic development of the brownbanded bamboo shark (Chiloscyllium punctatum). Developmental Dynamics. 247(5):712-723.
研究手法と成果
本研究では、海遊館のイヌザメと、理研生命機能科学研究センター水棲動物飼育施設のトラザメから採取した血液試料や胚に加え、海遊館および沖縄美ら海水族館のジンベエザメから採取した血液試料が用いられました。ジンベエザメの血液については、健康管理のため両水族館で定期的に採血[6]を実施しており、その一部を活用することにより、生育個体の生命を犠牲にすることなく生体試料を利用することができました。 まず共同研究グループは、対象とする3種のサメについて、細胞核に含まれるDNAの量を測り、ゲノムサイズを推定しました。その結果、総塩基対数が約32億塩基対のヒトゲノムに対し、ジンベエザメが38億、イヌザメが47億、トラザメに至っては67億であり、大きなゲノムを持つことが分かりました。
本研究では、生体試料の扱いから長大なゲノム配列の解析までを、超並列DNAシーケンサー[7]を効率的に運用する体制を備えている分子配列比較解析ユニット内で行うことにより、比較的少ない読み取りDNAデータ量から、高い完成度の全ゲノム配列情報を得ることができました。
サメゲノムの特徴を調べるため、本研究で得られたイヌザメおよびトラザメの全ゲノム配列と、既に公開されていたゲノム配列をつなぎ直したジンベエザメの全ゲノム配列を、他の脊椎動物のゲノム情報と比較しました。その結果、サメ類の肥大したゲノムは、ゲノム全体が倍加する全ゲノム重複[8]ではなく、反復配列の増幅などで起こる遺伝子間領域[9]の拡大によるものであることが分かりました。また、サメ類の系統では、進化にかかった時間の割に、DNAの塩基配列の変化が少なかったことから、突然変異率が低下していることが分かりました。ゲノムの倍加が起こっている両生類アフリカツメガエルや硬骨魚類ゼブラフィッシュなどと比べて、サメ類は塩基配列や遺伝子セットの変化が小さく、脊椎動物の祖先の特徴をより色濃く残していると推測されます。さらに、これまではサメ類やエイ類が進化の過程で失ったとされてきたHoxC遺伝子群[10]の少なくとも一部を、3種のサメが保持していることも明らかになりました。
サメ3種の全ゲノム情報が明らかになったことにより、それぞれの種が持っている遺伝子のカタログ作りと、その種間比較が可能になりました。そこで、サメ類の野生集団の生態の理解に加え、水族館でのより計画的な飼育・繁殖の基礎となる知見を得るため、食欲、成長、繁殖などを制御するさまざまなホルモンについて調べました(図2)。主に哺乳類で知られている成長ホルモン、オキシトシン、プロラクチン、レプチン、グレリン、オレキシンなどのホルモンを作る遺伝子の分子系統を推定するとともに、これらの遺伝子のゲノム上での配置を調べたところ、これらのホルモン遺伝子のほとんどについて、対応するサメ遺伝子を同定することができました。さらに、トラザメの脳や各臓器での遺伝子発現プロファイリング[11]により、同定した遺伝子が産生するホルモンの多くは、哺乳類で働くことが知られている脳下垂体や視床下部、腸や生殖巣など体の各部位で分泌されていることが推測されました。このことから、サメ類や哺乳類を含む有顎脊椎動物[12](図1)の共通祖先の時点で、既に内分泌システムを介した脳や消化器官の間の体内情報伝達ネットワークの基本パーツが確立されていたと考えられます。
次に、さまざまな環境へ適応していったサメ類の進化の痕跡を探るため、感覚系の遺伝子に注目しました。まず、一般に匂い分子の受容体[13]として機能することが知られている遺伝子の多様性を調べたところ、イヌザメでわずか3つしかなく、他の2種でも遺伝子の数が極端に少ないことが分かりました。このことから、サメ類の嗅覚は他の遺伝子群の働きによって担われているか、あるいは、電磁場を検知するなどの嗅覚以外の感覚に大きく依存していることが考えられます。
さらに、視覚をつかさどるオプシン[14]遺伝子の多様性を調べました。視覚をつかさどるオプシン遺伝子は、脊椎動物の進化の初期に起きた全ゲノム重複によって5つのタイプに分かれたことが知られています。今回得た全ゲノム情報を調べたところ、イヌザメとジンベエザメは、明暗視をつかさどるタイプのオプシンであるロドプシン遺伝子のほかに、長波長(赤色)吸収タイプ(long wavelength-sensitive、LWS)のオプシンを作る遺伝子を保持していることが分かりました(図3)。一方、トラザメのゲノム配列中には、基本5タイプのうちロドプシン遺伝子しか見つからなかったことから、色覚を持たない可能性が示されました。現生のトラザメは浅瀬から水深300メートル程度の深い海まで広く生息しますが、その祖先種は深海に定住していたと考えられており、赤い光が届かない環境にいた祖先の時代にLWSタイプのオプシンを失った可能性が考えられます。
また、同定した遺伝子配列から人工的にロドプシンタンパク質を合成して吸収波長を調べたところ、通常のロドプシンが500ナノメートル(nm、1nmは10億分の1メートル)の光を最も効率よく吸収するのに対して、ジンベエザメのロドプシンは深い海にも届きやすいとされる短めの波長(約480ナノメートル)の光を吸収するように変化していることが分かりました(図3)。このことは、海水面付近で捕食する傍らで1000メートル以深へ潜水するという、データロガー[15]の活用などによって近年明らかになってきたジンベエザメの生態についての知見と整合的でした。このように、ゲノム情報と分子生物学実験を組み合わせることにより、行動実験が困難なため明らかになっていない種の生態に迫ることができる可能性が示されました。
今後の期待
今回の解析により、サメ類と他の脊椎動物とで共通な部分と大きく異なる部分の両方がゲノム中に見いだされました。今後、さらに多様な種の情報を加えることによって、5億年に及ぶ壮大な脊椎動物の進化におけるゲノムの変遷の過程がより詳しく再構築されると期待できます。
サメ類の野生集団の動態は、食物連鎖のバランスに影響するため、ヒトの食料の確保や海水圏の生態系の維持と切っても切れない関係にあります。本研究によって得られたゲノム情報は、これまでDNA配列情報が限られていたサメ類に対する分子マーカー[16]選定の可能性を広げ、絶滅危惧種や乱獲の対象となっている種の効率的なモニタリングの助けにもなると期待できます。野生集団を脅かすことなく生物多様性に触れる機会を確保する上で、水族館における展示は今後も重要な役割を果たすと考えられます。特にサメ類の性行動や繁殖形態については不明な点が多く、今回得られたゲノム情報を用いた今後の研究が、水族館における計画的な飼育・繁殖にも貢献すると期待できます。
本研究で得られたサメ類の遺伝子配列情報は、既に10以上の研究室へ供与されており、今後、より多くの研究に役立てられると推測されます。
原論文情報
Yuichiro Hara, Kazuaki Yamaguchi, Koh Onimaru, Mitsutaka Kadota, Mitsumasa Koyanagi, Sean D. Keeley, Kaori Tatsumi, Kaori Tanaka, Fumio Motone, Yuka Kageyama, Ryo Nozu, Noritaka Adachi, Osamu Nishimura, Reiko Nakagawa, Chiharu Tanegashima, Itsuki Kiyatake, Rui Matsumoto, Kiyomi Murakumo, Kiyonori Nishida, Akihisa Terakita, Shigeru Kuratani, Keiichi Sato, Susumu Hyodo, and Shigehiro Kuraku, “Shark genomes provide insights into elasmobranch evolution and the origin of vertebrates”, Nature Ecology and Evolution, 10.1038/s41559-018-0673-5
発表者
理化学研究所
生命機能科学研究センター 分子配列比較解析ユニット
ユニットリーダー 工樂 樹洋(くらく しげひろ)
沖縄美ら島財団総合研究センター
上席研究員 佐藤 圭一(さとう けいいち)
(沖縄美ら海水族館 副館長)
海遊館
館長 西田 清徳(にしだ きよのり)
大阪市立大学 大学院理学研究科
生物地球系専攻 生体高分子機能学II研究室
教授 寺北 明久(てらきた あきひさ)
准教授 小柳 光正(こやなぎ みつまさ)
東京大学大気海洋研究所
海洋生命科学部門 生理学分野
教授 兵藤 晋(ひょうどう すすむ)
お問い合わせ先
理化学研究所 生命機能科学研究センター センター長室 報道担当
山岸 敦(やまぎし あつし)
報道担当
理化学研究所 広報室 報道担当
沖縄美ら島財団 企画広報課
菅間 加奈子(すがま かなこ)・安里 美希(あさと みき)
海遊館 広報チーム
村上 寛之(むらかみ ひろゆき)
大阪市立大学 広報室
三苫 優人(みとま ゆうと)
東京大学大気海洋研究所 広報室
補足説明
-
- 板鰓(ばんさい)類
- 軟骨魚類(軟骨魚綱)を構成する二つのグループのうちの一つ(板鰓亜綱)で、サメ類とエイ類が含まれる。板鰓とは、板状のエラのこと。板鰓類はエラ蓋を持たず、5~7対のエラ穴が外から見える。
-
- ゲノムサイズ
- 特定の種の一つの細胞の核に含まれるDNAの総量。通常は単相(n)に相当する量を表示する。種によってゲノムサイズの値が異なるが、その大小関係は生物の複雑性とは必ずしも対応してはいない。
-
- 卵生、胎生
- 動物の繁殖様式で、卵を産み体外で孵化させる「卵生」、卵を体外に出さず雌の体内で孵化させ、母体を通して胚発生に必要な栄養供給やガスの交換を行い出産に至る「胎生」がある。サメ類やエイ類ではさまざまな繁殖様式が見られ、胎盤を持つ種も存在する。なお、ジンベエザメはかつて卵生と考えられていたが、1995年に台湾で妊娠中の雌個体が捕獲され、胎生と判明した。このとき採取された胎仔の標本は、沖縄美ら海水族館で展示されている。
-
- ギンザメ類
- 軟骨魚類(軟骨魚綱)の全頭亜綱に含まれるグループ。板鰓類とは異なり、硬骨魚類のようにエラ蓋を持つ。
-
- ホルモン
- 体内の特定の器官で合成、分泌され、血液や体液を巡ったのち、別の特定の細胞で機能を発揮する生理活性物質。ステロイドホルモンのような脂溶性低分子とペプチドからなるホルモンがあり、ここでは後者を指す。
-
- ジンベエザメの採血
- ヒトの健康管理で行われているように、性ホルモン量などを測るために行われている。個体ごとにハズバンダリートレーニング(健康管理のための訓練)を行った上で、個体にストレスがかからないよう、保定をすることなしに水槽中で飼育員が採血を行う。
-
- 超並列DNAシーケンサー
- 次世代シーケンサーとも呼ばれる、断片化された数千万~数億本のDNA配列を一度に解読する装置。1配列あたりの解読できる長さは必ずしも長くはないものの、総和として膨大なDNA配列情報が産出される。全ゲノム配列の解読だけではなく、遺伝子発現の定量化、遺伝情報の個人差の同定など、現代の生物学や医学に広く活用されている。
-
- 全ゲノム重複
- 進化の過程でゲノム全体が倍加する現象。現存する脊椎動物の共通祖先や硬骨魚類真骨魚の共通祖先で起きたとされる比較的古い全ゲノム重複に加え、サケ類やフナ類の系統で起きたより新しい全ゲノム重複が知られている。
-
- 遺伝子間領域
- 長大なゲノムDNAのうち、特定の機能を持つタンパク質やRNAを指定するなどの機能を持っている遺伝子に挟まれたDNA領域。ヒトゲノムの場合、ゲノムの半分以上がこういった領域であり、多数コピー存在する反復配列やエンハンサーなど遺伝子の働きを制御する領域などが含まれる。
-
- HoxC遺伝子群
- 動物の前後軸に沿った領域特異的な構造の形成に関わる転写因子群の一部。通常、ホメオボックスと呼ばれるDNA結合ドメインを指定する共通の領域を持つ10個程度の遺伝子が、染色体上に一列に並んでクラスターを形成している。多くの脊椎動物は、二度の全ゲノム重複の結果、A、B、C、Dという4つのクラスターを持つ。今回サメで見つかったHoxC遺伝子群は、長大な遺伝子間領域に分断されるなど、一般的なHox遺伝子クラスターの特徴からは逸脱した性質を持っている。
-
- 遺伝子発現プロファイリング
- 超並列DNAシーケンサーなどを用いて、多数遺伝子の働く場所と時期を網羅的に調べる解析アプローチ。ゲノム中のそれぞれの遺伝子は、体のどこでいつ発現するか細かな制御を受けており、その情報を得ることでDNA配列情報からだけでは分からない遺伝子の機能を類推することができる。
-
- 有顎脊椎動物
- 脊椎動物(脊椎動物亜門)は、あごを持たない円口類(無顎上綱)とあごを持つ有顎類(顎口上綱)に大別される(図1)。円口類に属するヌタウナギとヤツメウナギを除く現存の全ての脊椎動物が有顎脊椎動物に含まれる。
-
- 匂い分子の受容体
- 正確には、7回膜貫通型Gタンパク質共役型受容体のうち、匂い分子を受容するもの。嗅覚受容体とも呼ばれる。ヒトは約400種の嗅覚受容体遺伝子を持つ。
-
- オプシン
- 視細胞で光をキャッチする視物質に代表される光受容タンパク質。明暗を感じる視細胞には弱い光にも反応できる桿体(かんたい)オプシン(ロドプシン)が発現し、色覚を担う視細胞には、強い光に反応する錐体(すいたい)オプシンが発現する。オプシンは、それぞれの固有の波長(色)感受性を持つため、色感受性の異なる複数のオプシンを使って色の違いを見分けることが可能となる。したがって、オプシン遺伝子を一つしか持たない動物は色の違いを見分けることができない。
-
- データロガー
- 温度、加速度、深度などのセンサーにより計測・収集した各種データを保存する装置。近年、野外の生物に取り付けることにより、生態調査に利用されるようになった。このような手法はバイオロギングと呼ばれている。ジンベエザメについては2015年、この手法により、最大潜行深度1,928メートルが記録されている。
-
- 分子マーカー
- 生物が持つ遺伝子や分子のうち、細胞種や細胞状態の区別、あるいは個体や種の識別に有用な特徴を持つものを分子マーカーと呼ぶ。
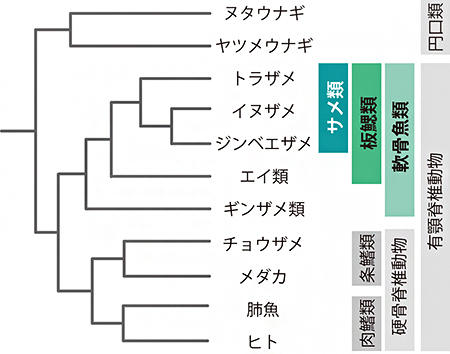
図1 最新の分子系統学的知見による系統関係
サメ類とエイ類を合わせたグループが板鰓(ばんさい)類、それにギンザメ類を合わせたグループが軟骨魚類と呼ばれる。系統学的には、ギンザメ類はサメ類とは異なる。軟骨魚類に対置する硬骨脊椎動物には、硬骨を持つ全ての魚類と四肢動物が含まれる。円口類は、顎(あご)を持たない脊椎動物である。
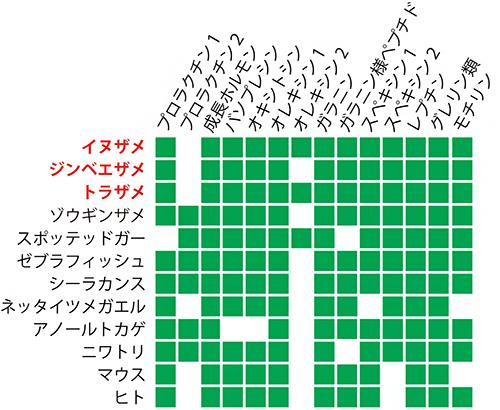
図2 ホルモン遺伝子セットの種間比較
緑の四角は、共通祖先から由来し種間で対応する遺伝子が少なくとも1つ存在すること示す。

図3 サメ類のオプシン遺伝子
5色で塗りつぶした四角は、5つのタイプのオプシン遺伝子のうち、それぞれの動物が持つ遺伝子を、そして点線の四角は遺伝子がゲノム中に見つからないことを表す。生息深度は、文献に基づいた概数。ゾウギンザメのロドプシンの最大吸収波長は既報注4)に、また、それ以外のサメ3種のロドプシンの最大吸収波長は本研究における分光測定の結果に基づく。
注4)Davies W. et al. 2009. Into the blue: Gene duplication and loss underlie color vision adaptations in a deep-sea chimaera, the elephant shark Callorhinchus milii. Genome Research. 19:415-426.