2025-02-06 理化学研究所,東京大学
理化学研究所(理研)生命機能科学研究センター 合成生物学研究チームの上田 泰己 チームリーダー(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 教授)、松本 桂彦 客員研究員(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 講師)、原田 頌子 テクニカルスタッフ(研究当時)、吉田 将太 リサーチアソシエイトらの共同研究グループは、抗体が認識するエピトープ[1]を、単一アミノ酸レベルの分解能でハイスループット[2]に解析する新手法を開発しました。
本研究成果は、抗体を利用する生物学・医学研究の再現性向上に貢献するとともに、血中抗体から未知の抗原を探索する応用技術となることが期待されます。
抗体は、生物学・医学研究や診断・治療に幅広く利用されていますが、市販されているものを含めて十分な品質評価がなされているとは言い難く、研究結果の再現性や信頼性の低下が危惧されています。この問題を解決する上で、抗体と標的タンパク質との結合に関わるエピトープ情報を知ることは非常に重要ですが、膨大な種類の抗体に対して網羅的かつ詳細に解析することは困難でした。
今回、共同研究グループは、mRNA display[3]のプロトコルの改良により高速化・単純化したペプチドセレクション[4]系「DECODE法」を開発しました。この方法により、モノクローナル抗体[5]またはポリクローナル抗体[5]が認識するエピトープのパターン(認識モチーフ[1])を単一アミノ酸分解能で特定し、タンパク質データベース全域に対する交差反応性[6]を予測することが可能になりました。さらに、自己免疫疾患[7]モデルマウスの血中抗体から病原ペプチドを同定することに成功し、DECODE法が未知の自己抗原の同定に役立つ将来性を示しました。
本研究は、科学雑誌『PLOS Biology』オンライン版(1月23日付)に掲載されました。
背景
免疫細胞が作る抗体は、標的分子(抗原)への結合に対する高い特異性や親和性を持つことから、生物学・医学研究のみならず、臨床現場でも診断薬・治療薬として幅広く応用されており、現在世界には700万種類を超える市販抗体が普及しています注1)。一方で、市販抗体の増加に対して品質評価が追い付いているとは言い難く、抗体が標的以外のタンパク質を認識する場合(交差反応性)や、ロットごとに染色性や特異性が変わる場合があることも知られています。このような個々の抗体の性能に関する情報不足が原因で、抗体を使用した研究成果の再現性や信頼性の低下が指摘されるようになりました注2)。
一般的に抗体は、標的となるタンパク質の大きさにかかわらず、5~8個のアミノ酸で構成される「エピトープ」と呼ばれる特定部位に結合します。抗体がタンパク質を認識するアミノ酸配列の特徴(エピトープ中の認識モチーフ)が分かれば、研究や治療の目的に応じた最適な抗体を選定することが可能になります。さらに、タンパク質の配列データベースとエピトープ情報から抗体の交差反応性を知ることで、免疫染色[8]などの実験において目的とする標的だけを特異的に認識する抗体を選択できるようになります。
このように抗体のエピトープ情報を詳細に知ることは非常に重要ですが、既存のエピトープデータベースに登録されている情報は、膨大な市販抗体数に比べて少なく、かつ認識モチーフや交差反応性に関する情報はほとんど公開されていません注3)。
注1)CiteAb – Reagent Search & Data Services
注2)Baker M. Nature. 2015 May 1;521 (7552) :274-6.
注3)Potocnakova L, Bhide M, Pulzova LB. J. Immunol. Res. 2016 Dec 29;2016:e6760830.
研究手法と成果
共同研究グループは、正確なエピトープ情報を網羅的に解析する新手法を開発し、モノクローナル抗体とポリクローナル抗体を含む市販抗体について、結合部位、認識モチーフ、タンパク質データベース全域に対する交差反応性といった詳細なエピトープ情報を取得しました。この手法を用いて自己免疫疾患モデルマウス血中の抗体群の網羅的なエピトープ解析を実施し、疾患の原因となる自己抗原の同定を行いました。
抗体と結合するタンパク質を解析する手法として、ランダムなアミノ酸配列を持つ短いタンパク質(ペプチド)を人工的に大量合成し、その中から抗体と結合したものを選択・回収する方法(ペプチドセレクション)があります。本研究では、この一種であるmRNA display法のプロトコルを最適化・単純化することで、ハイスループットなエピトープペプチドセレクション系「DECODE(Decoding Epitope Composition by Optimized-mRNA-display, Data analysis, and Expression sequencing)法」を開発しました。DECODE法では、カスタムPUREシステム[9]の使用などで、mRNA display法のプロトコルから複雑な工程を省略し、マルチウェルプレート[10]と自動分注機による並列化を実現して、セレクションの精度を低下させずに従来法の100倍以上のスループット化を実現しました(図1)。
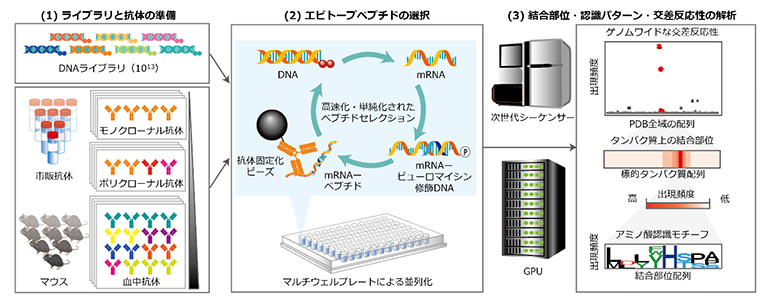
図1 DECODE法によるエピトープ解析の概要
(1)本手法では、mRNA display法において転写されたmRNAとピューロマイシン修飾DNAとの連結効率を高めるため、鋳型DNAのアンチセンス鎖の5’末端に二つの2′-O-メチルグアノシン(Gm)を含む1013以上のDNAライブラリを使用する。標的抗体は、モノクローナル抗体だけでなく、複雑なポリクローナル抗体や血中抗体にも適用できる。
(2)mRNA-ピューロマイシン修飾DNA複合体から翻訳されたペプチドは、ピューロマイシンの作用によりmRNAとペプチドが解離せず、両者の複合体として抗体固定化ビーズに回収される。この過程の並列性を高めるために、核酸分解酵素やタンパク質分解酵素を用いない無細胞翻訳システム(PUREシステム)を採用するなど従来のmRNA displayを改良し、精製過程を省略してもペプチド回収効率を落とさないプロトコルを構築し、マルチウェルプレートを使用したペプチドセレクションを実現した。
(3)ゲノムワイドなタンパク質データベースに対するGPU(画像処理装置)ベースのエピトープ解析。濃縮されたペプチド配列は次世代シーケンサーによって読み取られ、結合対象のタンパク質、結合部位、抗体によって認識されるアミノ酸残基のパターンなどのエピトープ情報を高解像度で特定する。
DECODE法により、200種類以上の市販抗体の詳細なエピトープ情報(1アミノ酸単位での結合部位配列と認識モチーフ)を網羅的に同定したところ、さまざまな発見がありました。例えば、市販されている8種の抗p53モノクローナル抗体[11]を調べたところ、クローンが異なり、抗体の配列も異なっているにもかかわらず、認識モチーフレベルでエピトープが同じものが発見されました(図2(1))。実際に実験を行う場合でも、別クローンの抗体を比較して適する抗体を選定することがよくありますが、ほとんど同じ性能のこれらのクローンを比較することはコストと時間の無駄になります。また、ポリクローナル抗体に関しては、型番が同じでもロットが異なるとほとんど違うエピトープを認識するため、再現性に乏しい研究になる可能性があることを確認しました(図2(2))。これらの結果は、エピトープを正確に把握した上で、市販抗体を選定することが重要であることを示しています。
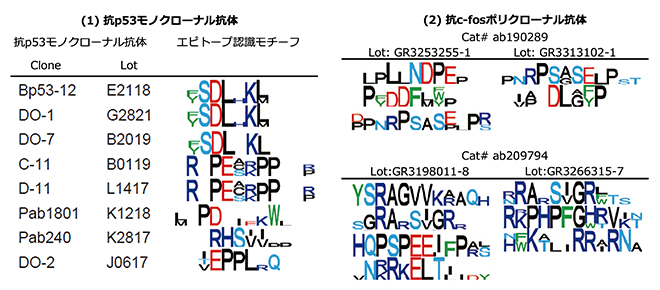
図2 エピトープの類似点と相違点の解明
(1)抗p53モノクローナル抗体について、クローン名が異なっているにもかかわらず、認識モチーフレベルで同じエピトープに結合する抗体が存在することが分かった。
(2)神経活動のマーカーである転写因子c-fosに対する抗c-fosポリクローナル抗体について、同じカタログナンバー(Cat#)の製品でも、ロット(Lot)が異なると認識するエピトープが異なることが分かった。エピトープ認識モチーフのアルファベットは連続するアミノ酸配列の1文字記号を示し、フォントの大きさはアミノ酸の重要度を視覚的に表したもの。二つ以上のアミノ酸が縦に並んでいるものは、同じ位置に複数のアミノ酸のバリエーションが存在することを示す。
抗体を用いた実験の一つに、組織や細胞内の特定のタンパク質に抗体を結合させて可視化する、免疫染色があります。免疫染色では、染色の前段階としてホルマリンなどによる生体試料の固定がしばしば行われますが、固定により標的タンパク質の抗原性が変化してしまうことがあります。本研究で得られたエピトープ情報は、固定による抗原性の低下の程度や、低下した抗原性を復活させる処理の効果が、抗体によって異なる原因の解明に貢献しました(図3(1))。
また、立体的な臓器を丸ごと観察するための新たな3D免疫染色方法の開発にも成功しました(図3(2))。これまで、マウス全脳や大きい組織の3D免疫染色を行う際、抗体が組織表面にとどまって内部まで浸透せず、均一な染色が難しい場合があるという課題を抱えていました。従来の3D免疫染色法(CUBIC-HV[12]法)では、浸透性を向上させる添加剤の濃度や種類を細かく調製する必要があり、複数の抗体を同時に用いて3D染色する場合は、この条件が一致する抗体同士でないと共染色できませんでした。一方、本研究で得られた詳細なエピトープ情報を基に抗体と結合するペプチドを合成し、染色液に添加すると、抗体の見かけの結合親和性が低下し浸透性を向上させることができました。本手法は個々の抗体ごとに高い特異性で親和性をコントロール可能であるため、どのような抗体の組み合わせでも3D免疫染色の共染色が可能になる画期的な手法です。
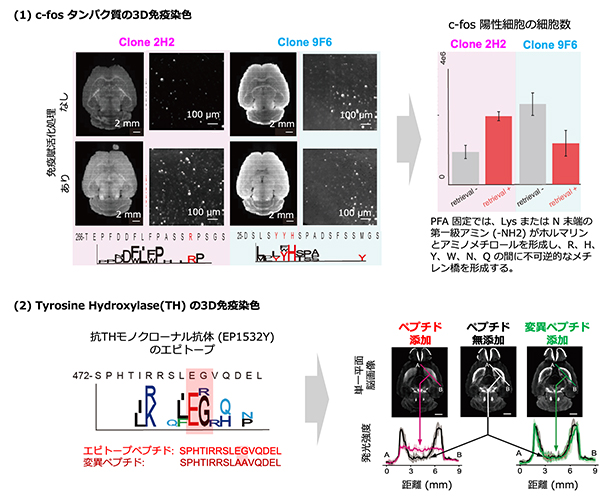
図3 エピトープを利用した免疫染色固定条件の最適化と3D免疫染色の抗体浸透率の向上
エピトープ情報の3D免疫染色への応用。
(1)抗c-fosモノクローナル抗体(クローン2H2とクローン9F6)を使用したc-fosタンパク質のマウス全脳3D免疫染色(免疫蛍光染色)の結果。全体写真は脳を背側から見た像、拡大写真は典型的な断層像を示す。ホルマリンまたはPFA固定された脳サンプルに対して、2H2と9F6で免疫染色結果に差が生じた。クローン2H2では、固定された組織や細胞の抗原を露出させるための前処理(免疫賦活化処理)をした場合にc-fos陽性細胞を多く検出したのに対し、クローン9F6ではその逆の結果となった。これは、エピトープ認識モチーフ中で特に抗体の認識に重要なアミノ酸(ホットスポット残基)に、ホルマリン(PFA)固定の際メチレン架橋を形成するアミノ酸種が含まれている場合、免疫賦活化処理後の抗原回復が難しくなることが原因であることを確認した。写真下のエピトープ情報で、赤色で示したアミノ酸(R:アルギニン、Y:チロシン、H:ヒスチジン)がホルマリンによりメチレン架橋される。
(2)神経伝達物質の酸性に関わるチロシン水酸化酵素(TH)に対する抗THモノクローナル抗体を使用したマウス全脳3D免疫染色(免疫蛍光染色)の結果。下のグラフは、写真中のA―Bを含む断層平面の蛍光強度を曲線で表したもので、AB線分の中央部の蛍光強度の低さは、抗体が脳深部に十分浸透していないことを示す。エピトープペプチドを添加しない場合、抗体は線条体表面にほとんどとどまるが、エピトープペプチドを添加すると線条体への浸透性が向上し、均一な免疫染色が可能になった。また、対照実験としてエピトープ中のホットスポット残基に変異を入れたペプチドを添加した場合はこの効果が消失し、浸透性が著しく低下した。
最後に、DECODE法の臨床応用を検討するため、血中に存在する病原ペプチドの同定が可能かを試みました。自己免疫疾患は、何らかの理由で自分自身を攻撃してしまう抗体(自己抗体)が産生されることで生じる疾患です。しかし、その原因となる自己抗原は未知の場合が多く、抗原タンパク質を使用して血中から自己抗体を濃縮するなどの研究戦略が有効ではありません。本研究では、抗原タンパク質が未知の場合でも、自己免疫疾患モデルマウスの血中から病原性エピトープを同定できるかどうかを検証しました。
具体的には、中枢神経の構成タンパク質の一部であるペプチド(MOG35-55ペプチド)を注射して実験的自己免疫性脳脊髄炎(EAE)[13]を再現したモデルマウス(EAEマウス)を作製し、その血液に含まれる抗体をそのまま磁気ビーズに結合させて、DECODE解析を実施しました。血中には、さまざまな抗原に対する多様な抗体が存在していますが、EAEマウスサンプルと健常マウスサンプルに対するDECODE解析で得られたエピトープを比較することで、EAEマウス特異的なエピトープを検出できました(図4)。このエピトープを調べた結果、注射した免疫原(MOG35-55ペプチド)の認識モチーフと一致したことから、血液から未知の病原性エピトープを同定できる可能性が示されました。
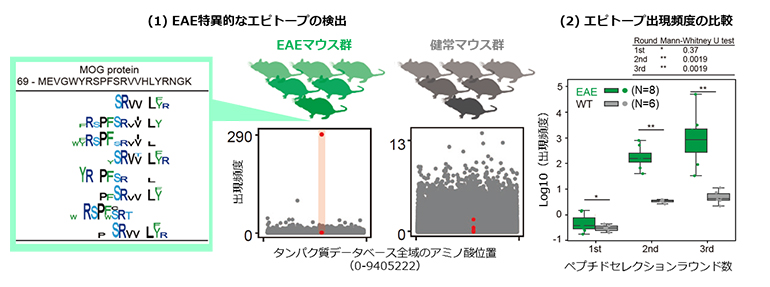
図4 EAEマウスの血中抗体特異的な病原性エピトープの特定
EAEマウス血中抗体に対するエピトープ解析の結果。健常マウスサンプルと比較してEAEマウスサンプルで顕著に出現頻度が増加したエピトープが発見された。
(1)EAEマウスサンプル特異的に出現頻度が増加したエピトープの結合部位と認識モチーフ。これは免疫原として用いたMOGペプチド配列とよく一致した。一方、健常マウスの解析サンプルにもMOGペプチド配列が一部含まれているが、タンパク質データベース上の配列全体にわたるさまざまなエピトープが非特異的に検出されている。
(2)EAEマウス(緑、N&8)と健常(WT)マウス(灰色、N&6)に間において、MOGペプチド配列が出現した頻度の比較。DECODE解析1回目のペプチドセレクションでは両者に有意差はない(MOGペプチド配列が偶然出現している可能性が高い)が、2ラウンド目からはEAEマウスサンプル特異的に病原性エピトープの出現頻度が増加する(EAEマウスでの出現頻度が有意に高い)ことが分かる。P値はMann-WhitneyのU検定によって取得された。*はP<0.05、**はP<0.001。
今後の期待
DECODE法が可能にする単一アミノ酸分解能かつ網羅的なエピトープ解析は、抗体を用いた生化学実験や病理診断などの研究から得られる結果を、より正確で安定したものにすると期待されます。さらに、血中から病原性エピトープを同定する手法は、近い将来、がん細胞や感染性病原体に由来する未知の病原の同定や、ワクチン開発、抗体医薬品の開発に大きく貢献すると期待できます。
補足説明
1.エピトープ、認識モチーフ
エピトープは、抗原タンパク質のうち抗体が結合する部位。一般的には5~8個のアミノ酸で構成される。エピトープの構造を形成するアミノ酸の重要度を示したものを認識モチーフと呼ぶ。
2.ハイスループット
従来手法の高速・効率化による大量処理を指す用語。high-throughput。
3.mRNA display
ペプチドセレクション([4]参照)の手法の一つ。mRNA displayでは、mRNAと翻訳産物(ペプチド)がピューロマイシンを介して共有結合されることで遺伝情報が保持される。このmRNA-ペプチド複合体は標的分子に結合する能力に基づいて選択された後、逆転写でcDNA-ペプチド複合体に変換され、ポリメラーゼ連鎖反応(PCR)による増幅で選択されたペプチドが濃縮される。mRNA displayは10の13乗ほどのペプチドライブラリを構築可能であり、ペプチドセレクションの中で最も高い多様性を示す。
4.ペプチドセレクション
膨大なペプチドライブラリから、特定の標的分子に高い結合親和性を持つペプチド配列を選び出す方法。
5.モノクローナル抗体、ポリクローナル抗体
単一のB細胞から作られた抗体のクローンをモノクローナル抗体、特定の抗原に対して複数の異なるB細胞クローンから産生される抗体の混合物をポリクローナル抗体と呼ぶ。
6.交差反応性
特定の抗原に対して作成された抗体が、類似した構造を持つ他の抗原にも反応すること。
7.自己免疫疾患
免疫システムに異常が生じ、自己の正常な組織に障害を与える疾患の総称。関節リウマチや1型糖尿病などがある。
8.免疫染色
抗体の特異性を利用して組織内の特定の抗原を検出し、その局在を顕微鏡下で可視化する技術。
9.PUREシステム
無細胞転写/翻訳システムの中でも、必要な要素のみ(翻訳開始因子・翻訳伸長因子・翻訳終結因子など10種類、アミノアシルtRNA合成酵素20種類、転写酵素1種類、その他エネルギー再生酵素など5種類)を一つずつ組換えタンパク質として精製し、試験管内において再構成する技術。PUREはProtein synthesis Using Recombinant Elementsの略。
10.マルチウェルプレート
複数の小さなウェル(井戸状のくぼみ)が配置されたプレート。本研究では主に96ウェルプレートを使用し、96種類のサンプルを並列処理した。
11.抗p53モノクローナル抗体
がん抑制遺伝子由来のがんマーカーp53タンパク質に特異的に結合する抗体。主にがん研究や診断に利用される。
12.CUBIC-HV
CUBICは、理研の研究チームが2014年に開発した、3次元イメージング、画像解析を組み合わせた全臓器・全身全細胞解析のためのパイプライン。CUBIC-HVは、CUBICに基づいて3D免疫染色のために開発された手法。バッファー組成や添加剤による抗体の組織への浸透性を向上させてマウス全臓器を均一に染色できる。CUBICは、Clear, Unobstructed Brain/Body Imaging Cocktails and Computational analysisの略。HVはHistoVIsionの意味。
13.実験的自己免疫性脳脊髄炎(EAE)
自己免疫疾患の研究に用いられる動物モデル。特に多発性硬化症(MS)の研究で広く利用されている。中枢神経のミエリンを構成するタンパク質の一つであるミエリンオリゴデンドロサイト糖タンパク質(MOG)を投与することにより発症誘導される。EAEはExperimental Autoimmune Encephalomyelitisの略。
共同研究グループ
理化学研究所 生命機能科学研究センター
合成生物学研究チーム
チームリーダー 上田 泰己(ウエダ・ヒロキ)
(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 教授)
客員研究員 松本 桂彦(マツモト・カツヒコ)
(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 講師)
テクニカルスタッフ(研究当時)原田 頌子(ハラダ・ショウコ)
(現 無細胞タンパク質合成研究チーム 研究員)
リサーチアソシエイト 吉田 将太(ヨシダ・ショウタ)
テクニカルスタッフ(研究当時)鳴海 良平(ナルミ・リョウヘイ)
(現 合成生物学研究チーム 客員研究員)
研修生 三谷 智樹(ミタニ・トモキ)
無細胞タンパク質合成研究チーム
テクニカルスタッフ 佐藤 文(サトウ・アヤ)
チームリーダー 清水 義宏(シミズ・ヨシヒロ)
大阪大学
大学院医学系研究科 病態病理学講座/大学医学部附属病院 病理部
教授 森井 英一(モリイ・エイイチ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、科学技術振興機構(JST)戦略的創造研究推進事業ERATO「上田生体時間プロジェクト(研究代表者:上田泰己、JPMJER2001)」、同ムーンショット型研究開発事業「恒常性の理解と制御による糖尿病および併発疾患の克服(研究代表者:片桐秀樹、JPMJMS2023)」、日本医療研究開発機構(AMED)先端的バイオ創薬等基盤技術開発事業JP21am0401011、同AMED-CREST JP21gm0610006、同Brain/MINDS JP21dm0207049(研究代表者はいずれも上田泰己)、日本学術振興会(JSPS)科学研究費助成事業基盤研究(S)「哺乳類生体リズム振動体の設計(研究代表者:上田泰己、18H05270)」、同基盤研究(C)「マウス全脳の機能的コネクトーム解析による睡眠覚醒の責任細胞の同定(研究代表者:松本桂彦、20K06885)」、同若手研究「全脳一細胞解像度の神経活動の測定による全脳全細胞の概日リズム解析(研究代表者:吉田将太、20K16626)」「全脳ニューロンアトラスによる神経変性疾患モデルの一細胞解像度全脳病態解析(研究代表者:三谷智樹、20K16498)」、Human Frontier Science Program grant 2018「Sleep, the clock, and the brain: a neuromathematical approach(研究代表者:上田泰己)」、文部科学省光・量子飛躍フラッグシッププログラム(Q-LEAP)「量子生命技術の創製と医学・生命科学の革新(研究代表者:馬場嘉信、JPMXS0120330644)」の助成を受けて行われました。
原論文情報
Katsuhiko Matsumoto*, Shoko Y. Harada*, Shota Y. Yoshida*, Ryohei Narumi, Tomoki T. Mitani, Saori Yada, Aya Sato, Eiichi Morii, Yoshihiro Shimizu, Hiroki R. Ueda#(*:共同筆頭著者、#:責任著者), “DECODE enables high-throughput mapping of antibody epitopes at single amino acid resolution”, PLOS Biology, 10.1371/journal.pbio.3002707
発表者
理化学研究所
生命機能科学研究センター 合成生物学研究チーム
チームリーダー 上田 泰己(ウエダ・ヒロキ)
(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 教授)
客員研究員 松本 桂彦(マツモト・カツヒコ)
(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 講師)
テクニカルスタッフ(研究当時)原田 頌子(ハラダ・ショウコ)
リサーチアソシエイト 吉田 将太(ヨシダ・ショウタ)
報道担当
理化学研究所 広報室 報道担当
東京大学大学院医学系研究科 総務チーム