がん免疫療法の適応診断や遺伝性腫瘍診断に有効
2020-03-25 理化学研究所,東京大学
理化学研究所(理研)生命医科学研究センターがんゲノム研究チームの藤本明洋副チームリーダー(研究当時、現 東京大学大学院医学系研究科教授)、中川英刀チームリーダーらの共同研究グループは、2,700例以上のがん全ゲノムシーケンスデータについて、「マイクロサテライト(MS)[1]」と呼ばれる繰り返し配列の変異を同定し、その特徴を明らかにしました。本研究は、国際連携による全ゲノムがん種横断的解析プロジェクト(PCAWG)[2]の一環として行われ、がん全ゲノムの「マイクロサテライト不安定性(MSI)」[3]に関するこれまでで最も網羅的な研究と診断となりました。
本研究成果は、がん免疫療法の適応診断、遺伝性腫瘍の診断に有効であり、次世代のがんゲノム医療研究の進展に貢献すると期待できます。
高変異率のMSマーカーを用いたMSI判定は、現在、遺伝性腫瘍[4]の診断やがん免疫療法の適応診断のために行われています。しかし、繰り返し配列の解析は非常に難しく、全ゲノムでどのようなMS変異が起きているかは不明でした。
今回、共同研究グループは、全ゲノムにおけるMS変異同定のための高精度解析法「MIMcall」を開発し、PCAWGで解析された2,717例の21種類のがんの全ゲノムシーケンスデータについて、1例あたり平均約765万カ所、合計約200億カ所のMS変異解析を行いました。その結果、ほとんどのMSに変異が見つかりませんでしたが、解析できたMS全体の約2.6%に当たる約20万カ所のMSにおいて複数個のがんゲノムに変異があることが分かりました。31例のがん(大腸がん、子宮体がん、胃がんなど)にMS変異が極端に多く見つかり、MSIと判定できました。さらに、高頻度に変異している20個のMSマーカーを新たに同定し、既存のMSマーカーと同程度の変異頻度を持ち、MSI判定の精度も同等であることが分かりました。
本研究は、科学雑誌『Genome Research』の掲載に先立ち、オンライン版(3月24日付:日本時間3月25日)に掲載されます。
背景
がんは、ゲノムに変異が蓄積することで発症・進行する「ゲノムの病気」であり、がんの本質は「ゲノムの不安定性」にあるといえます。現在、世界中でがんの網羅的な全ゲノムシーケンス解析[5]や、その結果得られるがんのゲノム変異に基づく薬の開発、個別化医療(がんゲノム医療)が精力的に行われています。2014~2019年には、がんゲノム分野の国際共同プロジェクト「全ゲノムがん種横断的解析(PCAWG)」により、がんの大規模な全ゲノムシーケンス解析が行われた結果、最も網羅的かつ詳細ながんゲノムカタログ[6]が発表されました注1)。
ヒトゲノムの30億塩基配列のうちの約半分を繰り返し配列が占めています。そのうち、1~6個の塩基が10~数十回繰り返した配列を「マイクロサテライト(MS)」と呼び、ヒトゲノム中には1000万個以上のMSがあると考えられています。MSは突然変異率が高く、個体間における繰り返しの長さの遺伝的多型[7]が豊富なことから、遺伝的マーカーとして集団遺伝学や家系解析などの研究に有効です。
がんの診療では、数個のMSマーカーの変異(繰り返しの長さが変化する)の有無によって「マイクロサテライト不安定性(MSI)」の判定が行われており、DNA修復機構が欠損する遺伝性腫瘍(リンチ症候群[4])の診断に利用されています。さらに近年、MSI陽性がんが免疫チェックポイント阻害剤[8]による治療によく反応することが分かってきたため、MSI判定は免疫チェックポイント阻害剤の適応基準にも使われるようになり、注目を集めています。しかし、これまでMSI判定は、高変異率を示してきた数個のMSマーカーを用いられていますが、ゲノム全体でどのようなMS変異が起きているのかは不明でした。また、MSの高変異率をもたらす機序についても多くは解明されていませんでした。
近年の次世代シーケンサー(NGS)[9]技術の急速な進展、情報解析技術やITハード面の技術革新に伴い、約30億塩基の情報からなるヒトの全ゲノムシーケンス解析が、容易かつ安価に行えるようになりました。今後は、全ゲノムシーケンス解析が研究のみならず、がんゲノム医療といった診断や個別化医療においても、重要な解析手法になると予測されています。しかし、現在のNGSの解析技術では、MSや繰り返し配列部位の解析は極めて難しく、NGSによる全ゲノムでの大規模なMS解析は行われてきませんでした。
注1)2020年2月6日プレスリリース「国際連携によるがん全ゲノムの大規模解析」
研究手法と成果
繰り返し配列であるMSには、ポリメラーゼ連鎖反応(PCR)[10]およびNGSでのシーケンス解析の段階においてエラー[11]が入りやすくなります。共同研究グループは、この測定でのエラーの頻度やパターンを解明するため、男性の正常DNAの全ゲノムシーケンスデータの中でX染色体[12]にあるMSに着目しました。男性のX染色体は1本しかなく、他の染色体に位置するMSのように二つのアレル[13]を評価する必要がないからです。そこで、男性X染色体のデータから全ゲノムシーケンスでのMSの人為的変異の割合を推定し、この情報を用いて、がんでのMS変異を同定するための解析法「MIMcall」を開発し、この手法が高い精度を持つことを確認しました。
次にMIMcallを用いて、PCAWGにて解析された2,717例に及ぶ21種類のがん全ゲノムシーケンスデータ(がんと正常)から、1例あたり平均約765万カ所、合計約200億カ所のMSを対象として変異解析を行いました。その結果、ほとんどのMSに変異は見つかりませんでしたが、解析できたMS全体の約2.6%に当たる約20万カ所のMSにおいて複数個のがんゲノムに変異があり、これら変異MSのほとんどは、タンパク質をコードしていないゲノム領域に存在していました。
また、変異MSの個数は、全ゲノムでのSNV(一塩基置換)変異[14]数とは相関していませんでしたが、indel(短い挿入・欠失)変異[14]数と強い相関があり、MS変異はindel変異と同じ機構で起こることが分かりました。さらに、変異MSの特徴を詳しく解析したところ、A(アデニン)やT(チミン)の繰り返しは変異率が高いこと(図1上)、MS変異にはDNA複製のタイミングやDNAの構造が関連することなどが示唆されました。
そして、ゲノム全体で解析できた約765万カ所のMSのうち3%以上に変異が見られた腫瘍をMSI陽性と判定し、31例の全ゲノムレベルでのMSI陽性がんを特定しました。その中には、大腸がん(59例中7例)、胃がん(78例中6例)、子宮体がん(50例中11例)が多く含まれ、他にも肝臓がん、膵臓がん、卵巣がん、腎臓がん、悪性黒色腫も1~2例ずつ含まれていました(図1下)。

図1 21のがん腫ごとの全ゲノムレベルでのマイクロサテライト(MS)の変異率
上:A(アデニン)が26回繰り返したMS(黒)に対する、挿入変異(青)と欠失変異(赤)を示した。
下:3%以上のMSに変異があるがんをマイクロサテライト不安定性(MSI; Microsatellite Instable)と判定した(赤丸)。大腸がん、子宮体がん、胃がんにMSI陽性のサンプルが複数検出できた。MSの変異が3%未満であるがんは、マイクロサテライト不安定性が認められない(MSS; Microsatellite Stable)とした(黒丸)。
この31例のMSI陽性がんのうち、DNA修復遺伝子の生殖細胞変異が2例で見つかりリンチ症候群に相当するとされ、DNA修復遺伝子の多くの体細胞変異も見つかりました。また、MSI陽性がんは変異によってネオ抗原[15]が多く発現し、抗原の産生などを誘導する免疫原性が高いと考えられています。本研究により、MSI陽性がんにはネオ抗原がMSI陰性がんよりも多く存在すること、MSI陽性がんの多くはタンパク質をコードする遺伝子内のMSあるいは繰り返し配列の変異からできたことが推測されました。
さらに、高頻度に変異している20個のMSマーカーを新たに同定し、埼玉県立がんセンターにある大腸がんと子宮体がんのDNAを用いて、そのMSIの診断精度を検証しました。その結果、新しいMSマーカーは、既存のMSマーカーと同程度の変異頻度を持ち、MSI判定の精度も同等であることが分かりました(図2)。
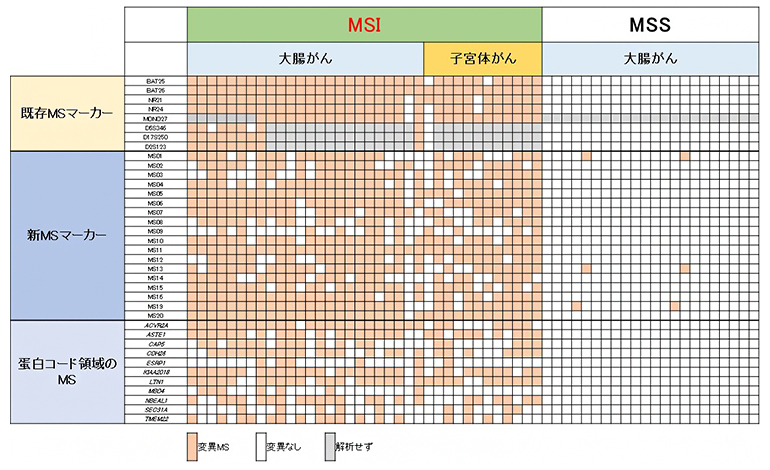
図2 新規MSマーカーによる大腸がんと子宮体がんのMSI判定
既存のMSI診断マーカー(8個)、今回の高変異率の新規MSマーカー(20個)、およびタンパク質コード領域(11個)のMSマーカーでの変異をオレンジで記している。MSI陽性の大腸がん(24例)、子宮体がん(12例)では、同程度の変異頻度を持つことが分かった。MSI陰性の大腸がん(22例)における変異頻度も同程度であることから、MSI判定の精度も同等であることが分かった。
今後の期待
今後、DNAシーケンス解析技術の革新に伴うコストの低下により、全ゲノムシーケンス解析が研究分野のみならず、がんの診断や個別化医療の分野においても標準的なゲノム解析手法になると予測されています。
MSIは、がんの本質である「ゲノムの不安定」に直接つながる現象であり、今回の大規模な全ゲノムシーケンスデータの解析によるMSIについての知見は、がんの本体の解明につながるものであり、今後、この現象を標的とした治療法が開発されると考えられます。
また、今回開発した全ゲノムシークエンスデータからのMS変異解析法(MIMcall)やMSI判定法は、全ゲノム時代でのがん免疫療法の適応および遺伝性腫瘍の診断に使われるようになると期待できます。
補足説明
1.マイクロサテライト(MS)
1~6個の塩基が10~数十回繰り返した配列のこと。ヒトゲノム中には1000万個以上のMSが存在すると考えられている。MSはMicrosatelliteの略。
2.全ゲノム横断的がん解析プロジェクト(PCAWG)
国際連携共同体であるICGC/TCGA内のがんの全ゲノムシーケンス解析のデータを集積し、ICGC/TCGAの共同作業にてがんの横断的(PanCancer)解析を行うプロジェクトとして、2014年に始動した。約2,800例のさまざまながんの全ゲノムシーケンスのビックデータを東京大学医科学研究所ヒトゲノム解析センターのスーパーコンピュータ「SHIROKANE」を含む世界10カ所のデータセンターで仮想空間を作り、分担して解析を行った。生シーケンスデータだけで約1ペタバイト(1000兆バイト)の情報量になる。PCAWGは、PanCancer Analysis of Whole Genomesの略。
3.マイクロサテライト不安定性(MSI)
細胞が分裂する際や環境曝露によって起こる、DNAの配列ミスを修復する機能が低下している状態のこと。がんでは、数個のMSマーカーの変異の有無(繰り返しの長さが変化する)によってMSIの判定が行われ、DNA修復機構が欠損する遺伝性大腸がんの一つであるリンチ症候群の診断に使われている。MSIはMicrosatellite Instabilityの略。
4.遺伝性腫瘍、リンチ症候群
がん全体の約5%は、遺伝?の配列の変化により?まれつきがんを発症しやすい体質が原因で発症すると考えられている。これを遺伝性腫瘍という。遺伝性腫瘍にはいくつかの種類があるが、遺伝性乳がん卵巣がんや「リンチ症候群」が主である。リンチ症候群は、大腸がんや子宮体がんなどの発症リスクを高める遺伝性疾患で、全大腸がんのうち2~5%の原因になっていると推定されている。
5.全ゲノムシーケンス解析
次世代シーケンサーを使って、個人(約30億塩基)やがんの全ゲノム情報を解読し、塩基配列の違いや変化を同定すること。データが大量になるため、大型計算機を使って情報解析を行うのが一般的である。タンパク質をコードする1~2%の範囲のエクソンだけでなく、遺伝子の発現を制御するゲノム領域の変異やさまざまな構造異常(大きなゲノム配列異常)も検出可能で、究極のゲノム解析手法といえる。がんの場合は、がんのDNAと同一患者由来の正常DNAの全ゲノムシーケンス解析を行い、その差分を調べる。
6.がんゲノムカタログ
30億塩基からなるヒトゲノムマップで、どの位置にどういった型の変異が蓄積しているのかを、網羅的にカタログ化したもの。例えば、最も多く見つかるKRAS遺伝子の変異は、12番染色体の25245350の位置にCからTへの変異が見られる。
7.遺伝的多型
同じヒトであっても個人個人のゲノムの塩基配列は多種多様であり、人口の1%以上の頻度で存在する配列の違いを遺伝子多型という。一つの塩基が他の塩基に置き換わっている一塩基多型(SNPs)やMSの長さの違いなどが、ゲノム解析の多型マーカーとして使われている。
8.免疫チェックポイン阻害剤
免疫のチェックポイントであるPD-1分子を抗体で抑制することで、腫瘍細胞への免疫抑制を解除し、免疫細胞が腫瘍細胞を排除できるように導く薬剤。現在、日本ではニボルマブとペムブロリズマブが、肺がん、胃がん、悪性黒色腫、腎臓がん、そしてMSI陽性の大腸がんなどについて承認され、治療に使われている。
9.次世代シーケンサー(NGS)
ヒトゲノムの全配列を1,000米ドル以下のコストで解読すべく、欧米の政府や企業が技術開発を行った結果、より高速高精度の性能を持つシーケンサーが開発された。この次世代シーケンサーは従来のものに比べ、超大量のDNAシーケンス反応を並列して行うことができる。NGSはNext Generation Sequencerの略。近い将来、100米ドル以下のコストになるものと予測されている。
10.ポリメラーゼ連鎖反応(PCR)
熱耐性を持つDNA依存性DNA合成酵素(DNAポリメラーゼ)を用いて、鋳型DNAとPCRプライマーの結合、相補DNAの合成、二本鎖DNAの解離の3ステップを繰り返すことで、DNAを連続的かつ指数関数的に増幅させることができる反応。PCRはPolymerase Chain Reactionの略。
11.シーケンスの段階におけるエラー
細胞内におけるDNAの複製や修復と同様、PCRや次世代シーケンサーでの化学反応は主にDNAポリメラーゼ(DNA合成酵素)が担っており、一定の割合で間違い(エラー)が生じる。ただし、細胞内におけるエラーの多くはDNA修復機能によって訂正される。
12.X染色体
ヒトの全遺伝情報(ゲノム)は、22本の常染色体が2セットと、2本の性染色体からなる。女性の性染色体は2本ともX染色体である一方、男性ではX染色体とY染色体が1本ずつ存在する。このことから、MSのように極めて多型に富む(長さが異なる)マーカーを解析する際には、男性のX染色体を用いれば2本の染色体における長さの変化を考える必要がない。そのため、男性のX染色体は精度解析に適している。
13.アレル
哺乳類は母親と父親から同じ遺伝子セットを持つ染色体を1組ずつ受け継ぐ。この両親から受け継いだ1対の遺伝子セットを対立遺伝子またはアレルと呼ぶ。
14.SNV(一塩基置換)変異、indel(短い挿入・欠失)変異
SNV変異は、ゲノム上の一塩基(A、T、C、G)が変換される変異。Indel変異は、1~数塩基配列の挿入または欠失による変異。SNVはSingle Nucleotide Variantの略。
15.ネオ抗原
タンパク質をコードする遺伝子に変異が入ると、アミノ酸が異なるタンパク質が発現するようになる。その一部のペプチド配列が腫瘍細胞の表面に提示されると、免疫細胞が非自己と認識し、腫瘍細胞を攻撃できるようになる。MSI陽性がんではMSI陰性がんに比べてこのような変異が圧倒的に多く、ネオ抗原を多く発現する。そのため免疫原性が高く、免疫チェックポイント阻害剤の効果が期待できる。
共同研究グループ
理化学研究所 生命医科学研究センター がんゲノム研究チーム
チームリーダー 中川 英刀(なかがわ ひでわき)
上級研究員 藤田 征志(ふじた まさし)
東京大学 大学院医学系研究科 国際保健学専攻 人類遺伝学分野
教授 藤本 明洋(ふじもと あきひろ)
東京大学 医科学研究所
ヒトゲノム解析センター DNA情報解析分野
教授 宮野 悟(みやの さとる)
ヘルスインテリジェンスセンター 健康医療データサイエンス分野
教授 井元 清哉(いもと せいや)
埼玉県立がんセンター 腫瘍診断・予防科
部長 赤木 究(あかぎ きわむ)
原論文情報
Akihiro Fujimoto, Masashi Fujita, Takanori Hasegawa, Jing Hao Wong, Kazuhiro Maejima, Aya Oku-Sasaki, Kaoru Nakano, Yuichi Shiraishi, Satoru Miyano, Go Yamamoto, Kiwamu Akagi, Seiya Imoto, and Hidewaki Nakagawa, “Comprehensive Analysis of Indels in Whole-genome Microsatellite Regions and Microsatellite Instability across 21 Cancer Types”, Genome Research, 10.1101/gr.255206.119
発表者
理化学研究所
生命医科学研究センター がんゲノム研究チーム
チームリーダー 中川 英刀(なかがわ ひでわき)
東京大学 大学院 医学系研究科 国際保健学専攻 人類遺伝学分野
教授 藤本 明洋(ふじもと あきひろ)
(研究当時:理研生命医科学研究センターがんゲノム研究チーム 副チームリーダー)
報道担当
理化学研究所 広報室 報道担当