2018-04-11 理化学研究所
要旨
理化学研究所(理研)脳神経科学研究センタータンパク質構造疾患研究チームのチェンウェン・チェン研究員と田中元雅チームリーダーの研究チームは、タンパク質合成中の転移RNA(tRNA)[1]の種類や量を詳しく解析する「tRNAリボソームプロファイリング法[2]」を新たに開発し、翻訳中のリボソーム[3]内に入っているメッセンジャーRNA(mRNA)とtRNAを同時に解析することで、タンパク質合成の過程をより詳しく調べることに成功しました。
mRNAが翻訳されてタンパク質が合成される過程は、全ての生命活動の根幹をなしています。しかし、タンパク質合成装置であるリボソームの中で、実際にどのようなことが起こっているのかは不明な点が多く残されています。特に、リボソーム内に取り込まれ、タンパク質合成に使われているtRNAの種類、性質、量については、その測定技術がないために十分に理解されていませんでした。
今回、研究チームは出芽酵母を用いて、翻訳中のリボソーム内に入っているtRNAを網羅的に解析するtRNAリボソームプロファイリング法を新たに開発しました。また、本手法と従来法を組み合わせることにより、リボソーム内に入っているtRNAとmRNAを同時に解析する「tRNA/mRNA同時リボソームプロファイリング」が可能になりました。解析の結果、開始メチオニン[4]tRNAの量が翻訳阻害のバイオマーカーになり得ることや、一部分解したtRNAが関わる新たな翻訳阻害機構を明らかにしました。
最近、特定のtRNAの発現異常や分解が、がんや神経変性疾患[5]に関与すると指摘されています。本手法は従来法に比べて、測定時間やコスト、データ解析の面ではるかにコンパクトな次世代シーケンサー[6]での解析ができます。そのため、これらの疾患に対して、tRNAの変化に着目した新たなバイオマーカーや治療法の開発に寄与すると期待できます。
本研究は、米国の科学雑誌『Cell Reports』に掲載されるのに先立ち、オンライン版(4月10日付け:日本時間4月11日)に掲載されます。
本研究は、日本学術振興会(JSPS)科学研究費補助金 新学術領域「新生鎖の生物学(領域代表:田口英樹)」などの支援を受けて行われました。
背景
生命は、多種多様なタンパク質の働きにより維持されています。そのため、メッセンジャーRNA(mRNA)が翻訳されタンパク質が合成される過程は、全ての生命活動の根幹をなしています。2009年に、リボソーム内に入って実際の翻訳に使われているmRNAを網羅的に解析する「リボソームプロファイリング法」が開発されました注1)。しかし、タンパク質の部品となるのは転移RNA(tRNA)であるにもかかわらず、翻訳中のリボソーム内に取り込まれているtRNAの種類、性質、量に関しては、測定技術がないために十分に理解されていませんでした。
一方、細胞は環境ストレスにさらされるとそのストレスに応答し、mRNAの翻訳様式を変えて、その環境に適応することが知られています。その際、翻訳の開始、伸長の各過程に関わるtRNAは、環境ストレスの標的になると考えられます。しかし、翻訳中のリボソーム内に存在するtRNAを詳しく調べる手法がないため、環境ストレスによる翻訳の変化にtRNAがどのように関わっているのか、よく分かっていませんでした。
そこで、研究チームは真核生物である出芽酵母を用いて、翻訳中のリボソーム内に取り込まれているtRNAの種類や量を網羅的に調べる手法を開発し、その手法を用いて、環境ストレス下における翻訳阻害の分子メカニズムの解明に取り組みました。
注1) Ingolia NT, Ghaemmaghami S, Newman JR, Weissman JS. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 324, 218-223 (2009).
研究手法と成果
研究チームはまず、増殖期の出芽酵母から調整した抽出液をRNA分解酵素で処理し、スクロース勾配実験[7]によってモノソーム[8]を単離しました。単離したモノソームから、mRNA(20~30塩基)とtRNA(70~100塩基)に対応する大きさの核酸を単離・精製し、逆転写酵素[9]などを用いて次世代シーケンサーで解析を行うためのライブラリー(Ribo-mRNA-seq、Ribo-tRNA-seq)を作成しました。
また、リボソーム内に入っているmRNAやtRNAの比較対照として、酵母抽出液全体からもmRNAおよびtRNAを単離・精製し、次世代シーケンサーで解析するためのライブラリー(mRNA-seq、tRNA-seq)を作成しました。このように、同一の酵母試料から、四つのライブラリーを同時に作成しました(図1)。
次に、次世代シーケンサーを用いて、Ribo-tRNA-seqとtRNA-seqの解析を行ったところ、酵母の染色体の各tRNA遺伝子座に、配列リードが特異的にマッピングできていることを確認しました。これは、tRNAを含むライブラリーが適切に作成されていることを示します。また、次世代シーケンサーでmRNAコドン[10]とtRNAアンチコドン[10]の相関を調べたところ、リボソームのP部位[11]からtRNAを多く収集できていました。この結果から、tRNA/mRNA同時リボソームプロファイリング法を用いて、実際の翻訳に使われているコドンの解析が可能になりました。
tRNAにはメチル化など多くの修飾が存在し、tRNAの安定性などに寄与しています。ライブラリー作成時に用いた逆転写反応[9]では、tRNAに一部の修飾が存在することで、その逆転写反応が途中で止まったり、塩基置換を生じたりすることが知られています。そこで、この現象を利用し、リボソーム内に入っているtRNAにどのような修飾が存在するか調べたところ、約10種類のtRNA修飾を定量的に解析できる可能性が示されました。
さらに、酵母を栄養飢餓や酸化ストレスに暴露して、翻訳がどのように変化するかをRibo-mRNA-seq、Ribo-tRNA-seq、mRNA-seq、tRNA-seqを用いて解析しました。その結果、四つのライブラリーによる解析の中で、Ribo-tRNA-seqが、環境ストレスに応答した翻訳の変化を最も敏感に反映することが分かりました。そこで、さらに環境ストレス下におけるRibo-tRNA-seqのデータを詳しく調べたところ、リボソームあたりの開始メチオニンtRNAの量が、ストレスによる翻訳阻害が翻訳開始阻害かペプチド伸長阻害かを知るための、簡易的なマーカーになることが示されました(図2)。
最後に、過酸化水素による酸化ストレスに対するtRNAへの影響を調べたところ、tRNAの3’末端[12]に存在するCCA塩基(C:シトシン、A:アデニン)の一部が酸化ストレスによって分解し、欠損していることを見いだしました。tRNAのCCA末端にはアミノ酸が結合しているため、CCAの一部が欠損していることは、そのtRNAがアミノ酸を保持していないことを示しています。そのようなtRNAがリボソーム内に取り込まれるのか、また、それによって翻訳阻害が引き起こされるのかをRibo-tRNA-seqによって調べました。その結果、CCA末端の一部を欠損したtRNAの一部は、実際にリボソーム内に取り込まれ、翻訳阻害を引き起こすことが明らかになりました。
今後の期待
近年、特定のtRNAの異常な発現や分解が、がんや神経変性疾患などのヒト疾患に深く関わることが示されています。既存のmRNAリボソームプロファイリング法に比べてtRNAリボソームプロファイリング法は、時間やコストの面ではるかにコンパクトな次世代シーケンサーでの解析ができます。そのため、tRNAリボソームプロファイリング法は、それらの疾患に対して、tRNAの変化に着目した新たなバイオマーカーや治療法の開発に寄与すると期待できます。
また、コンパクトなtRNAリボソームプロファイリング法は、次世代シーケンサーを用いた網羅的な遺伝子解析の裾野を大きく広げると期待できます。
原論文情報
Chien-Wen Chen and Motomasa Tanaka, “Genome-Wide Translation Profiling by Ribosome-Bound tRNA Capture”, Cell Reports, 10.1016/j.celrep.2018.03.035
発表者
理化学研究所
脳神経科学研究センター タンパク質構造疾患研究チーム
研究員 チェンウェン・チェン(Chien-Wen Chen)
(旧 脳科学総合研究センター タンパク質構造疾患研究チーム 研究員)
チームリーダー 田中 元雅(たなか もとまさ)
(旧 脳科学総合研究センター タンパク質構造疾患研究チーム チームリーダー)
報道担当
理化学研究所 広報室 報道担当
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
補足説明
-
- 転移RNA(tRNA)
- 転移リボ核酸(transfer RNA)の略号。アミノ酸の種類ごとに1~数種類のtRNAが存在する。tRNAはそれぞれ対応するアミノ酸を末端に結合し、リボソームまで運搬する。リボソームにアミノ酸を受け渡すと、再びアミノ酸を結合して、リボソームまで運搬する働きを繰り返す。
-
- リボソームプロファイリング法
- 細胞内に存在しているmRNAを調べるだけでは、それが翻訳に使われているか不明である。リボソーム内に入って実際に翻訳に使われているmRNA(約30塩基)を網羅的に調べる手法。
-
- リボソーム
- メッセンジャーRNA(mRNA)の翻訳を行なう(遺伝情報からタンパク質を合成する)場所。真核生物では、40Sの小サブユニットと60Sの大サブユニットからなる。
-
- 開始メチオニンtRNA
- 細胞内に二種類存在するメチオニンを結合したtRNAの一つ。開始メチオニンtRNAは、翻訳開始時に使われる。もう一つは、ペプチド伸長時に使われる伸長メチオニンtRNA。
-
- 神経変性疾患
- 神経組織の変性を伴う病気。異常なタンパク質の凝集などを伴い、神経細胞が損傷するために起きると考えられている。アルツハイマー型認知症やパーキンソン病、プリオン病、筋萎縮性側索硬化症(ALS)などが知られている。
-
- 次世代シーケンサー
- 多数のDNA断片を同時並行で解析し、大量の配列を読み取ることができるDNA配列解析装置。サンガー法を利用した蛍光キャピラリーシーケンサーである「第一世代シーケンサー」と対比して使われる用語。
-
- スクロース勾配実験
- スクロース勾配溶液を用いて、リボソームの大きさ(重さ)を指標にリボソームの大小の各サブユニットやモノソームとポリソームを分離する実験。
-
- モノソーム
- 一つ以上のリボソームがmRNAに結合して翻訳を行っている中で、mRNAに一つのリボソームが存在する状態。mRNAに複数のリボソームが連結した状態は、ポリソームという。
-
- 逆転写酵素、逆転写反応
- 逆転写反応は、RNA依存性DNAポリメラーゼによって、RNAを鋳型として相補的なDNAを合成する反応。その反応を触媒するのが逆転写酵素。
-
- コドン、アンチコドン
- mRNAが翻訳されタンパク質に変換される際、各アミノ酸を規定する三つの塩基配列のことをコドンと呼ぶ。一方、それぞれのアミノ酸に対応するtRNAも存在し、それらはコドンに相補的な塩基配列をもち、アンチコドンと呼ぶ。
-
- P部位
- リボソームの中には、アミノアシルtRNAが結合するA部位、ペプチジルtRNAが結合するP部位、ポリペプチド鎖を渡したtRNAが出ていくE部位の三つの部位が存在する。
-
- 3’末端
- RNAはヌクレオシドの2′-リボースの5’位のヒドロキシ基と3’位のヒドロキシ基がリン酸ジエステル結合によって連結されてできている。RNAの5’位のヒドロキシ基側を5’末端と呼び、3’位のヒドロキシ基側を3’末端と呼ぶ。
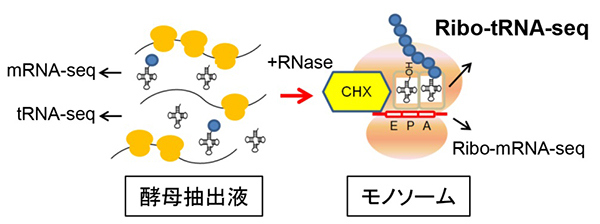
図1 tRNA/mRNA同時リボソームプロファイリング法の開発
同一酵母から四つのライブラリーを作成した。酵母抽出液からtRNA-seq、mRNA-seq用のライブラリー、RNA分解酵素(RNase)処理後のモノソームからRibo-tRNA-seq、Ribo-mRNA-seq用を作成した。
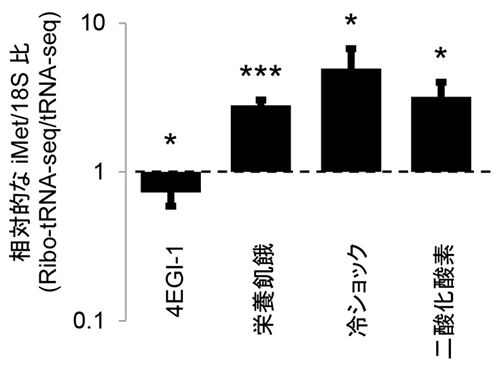
図2 翻訳阻害のバイオマーカーになる開始メチオニンtRNAの相対的な量
酵母を翻訳開始阻害剤(4EGI-1)、栄養飢餓、冷ショック、過酸化水素にさらしたときに、リボソームあたりの開始メチオニンtRNAの量を定量した。その結果、開始メチオニンtRNAの量は、翻訳開始阻害かペプチド伸長阻害かを調べるための指標になることが明らかになった。