理研を中心とする国際研究コンソーシアム「FANTOM6」
2020-07-28 理化学研究所
理化学研究所(理研)生命医科学研究センターゲノム情報解析チームのジョーダン・ラミロフスキー研究員、遺伝子制御回路研究チームのイップ・チーワイ研究員、ジェイ・シンチームリーダー、応用計算ゲノミクス研究チームのタンビル・アラム研修生(研究当時)、ミヒル・デ・ホーンチームリーダー、トランスクリプトーム研究チームのピエロ・カルニンチチームリーダーらの国際共同研究グループは、大規模なトランスクリプトーム解析[1]により、生物種を越えて細胞のアイデンティティーを形成する遺伝子発現パターンを発見し、さらに「長鎖ノンコーディングRNA(lncRNA)」[2]の解析により機能注釈を行いました。
本研究成果は、遺伝子発現パターンによる細胞種の同定や、機能性lncRNA研究の発展に貢献すると期待できます。
「FANTOM」は、ゲノムDNAから転写されるRNAの機能をカタログ化することを目的に2000年に発足した、理研を中心とする国際研究コンソーシアムです。今回、第6期目となるFANTOM6プロジェクト[3]では、FANTOM5プロジェクト[3]で得られたヒトを含む5種類の生物種の全遺伝子の発現情報(トランスクリプトーム[1])を比較し、生物種間で保存された共通のコアとなる遺伝子発現パターンがあることを明らかにしました。さらに、ヒトの285種のlncRNAについて大規模な機能解析を行った結果、発現量を減少させた119種のlncRNAのうち、13種(11%)が細胞の増殖や形態のみならず、転写、翻訳、代謝、発生などさまざまな細胞機能に関連していることを明らかにしました。
本研究の成果は、2報の論文として科学雑誌『Genome Research』に掲載されるのに先立ち、オンライン版(7月27日付:日本時間7月28日)に掲載されます。
背景
DNAから転写されるRNAには、タンパク質をコードするメッセンジャーRNA(mRNA)と、タンパク質をコードしないノンコーディングRNA(ncRNA)があります。ncRNAには、転移RNA(tRNA)[4]やリボソームRNA(rRNA)[4]などのほか、約200塩基以上の「長鎖ノンコーディングRNA(lncRNA)」があります。lncRNAの一部は、転写や翻訳、エピジェネティクス[5]の制御などを介して、細胞の分化やがん化、個体発生や疾患など、生体の多様なプロセスに関与することが知られています。しかし、大部分のlncRNAの役割はよく分かっていません。
理研は2000年から、哺乳類のゲノム機能を明らかにすることを目的にした国際研究コンソーシアム「FANTOM」を主宰し、次々にゲノムに関する謎を明らかにしてきました。プロジェクト5期目にあたるFANTOM5では、マウスやヒトの初代細胞や各種組織を用いた遺伝子発現解析により、lncRNAの発現の仕方が細胞種によって異なる傾向があることを明らかにしました。さらに、同コンソーシアムで開発されたCAGE法[6]による詳細な解析により、ヒトのlncRNA27,919種のうち19,175種が、何らかの生物学的な機能を持つ可能性があることが示されました注1)。
今期のFANTOM6では、その解析をさらに進め、ヒトを含む5種の生物種におけるトランスクリプトームの比較解析を行い、細胞種に特有の保存された発現パターンがないかを調べました。また、その大半の機能が未知であるlncRNAについて網羅的に調べるために、細胞内局在や発現量の異なる285種のlncRNAについて解析を行いました。
注1)2017年3月2日のプレスリリース「タンパク質をコードしないRNAをカタログ化」
研究手法と成果
国際共同研究グループはまず、ヒト、マウス、ラット、イヌ、トリの初代細胞[7]を用いて、それらの全遺伝子の発現情報を比較検討しました。その結果、異種動物間では、同じ細胞種であっても、平均して50%以上の遺伝子においてその発現量が異なることが分かりました。しかし、進化的に古い遺伝子や、RNAの転写やRNAプロセシング[8]などの制御に関わる遺伝子の発現レベルについては、異種間でも同じ傾向があることを見いだしました。これは、細胞種に特有の転写プログラムが進化的に保存されていることを示しており、今後、遺伝子発現パターンによる細胞種の同定に役立つと考えられます。
次に、ヒトの皮膚由来の初代線維芽細胞において、細胞内局在や発現量の異なる285種のlncRNAについて解析を行いました。ロボットによる自動化システムを構築し、各標的lncRNAに対してアンチセンスオリゴヌクレオチド[9]を用いて、遺伝子機能抑制(ノックダウン)[10]を行い、リアルタイムで細胞の増殖速度や形態を観察し、遺伝子発現の変化を解析しました。その結果、285種のlncRNAのうち194種(68%)において発現が50%以上抑制でき、そのうち15種(8%)が細胞増殖の維持に、50種(25%)が細胞の形態維持に関与することが分かりました(図1左・中)。
また、ノックダウンによるトランスクリプトーム変化の詳しいコンピュータ解析から、ノックダウンできた119種(42%)のlncRNAのうち13種(11%)が、細胞の増殖や形態のみならず、転写、翻訳、代謝、発生などさまざまな細胞機能を持つことが明らかになりました(図1右)。実際に、それら13種類のうち、ZNF213-AS1とKHDC3L-2という2種のlncRNAは、皮膚の創傷治癒の際に重要となる、細胞の増殖や遊走[11]をサポートすることや、隣接するタンパクをコードする遺伝子の発現を制御することで、細胞の機能に影響を及ぼすことを確認しました。
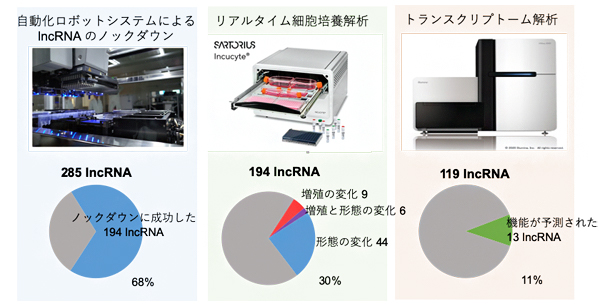
図1 解析のワークフローと長鎖ノンコーディングRNA(lncRNA)の機能注釈
左:自動化ロボットシステムにより、ヒトの皮膚由来初代線維芽細胞の285種のlncRNAについて解析した。そのうち、68%にあたる194種のlncRNAのノックダウンに成功した。
中:リアルタイムで細胞の増殖速度や形態を観察し、遺伝子発現の変化を解析した結果、194種のlncRNAのうち、30%にあたる59種のlncRNAが細胞の増殖・形態の維持に関与することが分かった。
右:コンピュータによるトランスクリプトーム解析により、ノックダウンできた119種のlncRNAのうち、11%にあたる13種がさまざまな細胞機能を持つことが分かった。
今後の期待
本研究では、RNAがDNAとタンパク質を結ぶ単なる中間生成物ではなく、生物種を越えて細胞のアイデンティティーの指標の一つとなること、また、”ジャンク”と考えられていたlncRNAが、細胞の状態を正常に保つためのさまざまな機能を持つことが明らかになりました。
また、細胞実験で観察されたデータ(細胞表現型)とコンピュータによるトランスクリプトーム解析で得られたデータ(分子表現型)を組み合わせた大規模なlncRNAの機能注釈の手法が、RNA生物学において有効な研究手法の一つになることが示されました。本手法は、今後さらに発展していくと期待できます。
今回の研究で取得した網羅的なシークエンスデータは、世界中の研究者に広く公開されます。なお、理研が開発したユーザーフレンドリーなバイオインフォマティクス・ツール「ZENBU(ゼンブ)注2)」でも参照できます。
注2)ZENBU – omics interactive visualization system(英語)![]() 」
」
補足説明
1.トランスクリプトーム解析、トランスクリプトーム
トランスクリプトームとは、一つのゲノム、または特定の細胞・組織・器官の中で生産される転写産物(転写によって合成されたRNA)全体を指す。トランスクリプトーム解析は、特定の遺伝子発現に着目するのではなく、全ての発現遺伝子に関して網羅的に解析する手法。マイクロアレイや、次世代シークエンサーを用いたRNAシークエンスなどの方法がある。
2.長鎖ノンコーディングRNA(lncRNA)
タンパク質をコードしないノンコーディングRNAの一種。一般に、約200塩基以上のものを指す。全長にわたる保存性は高くないが、反復配列やウィルス由来の配列断片を含むものも多い。転写、翻訳、エピジェネティクスなど生体内の多様なプロセスに関与するものが知られている。
3.FANTOM6プロジェクト、FANTOM5プロジェクト
FANTOMは理化学研究所が主催する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センターの林崎良英グループディレクター(現、理研予防医療・診断技術開発プログラム プログラムディレクター)が中心となり2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。5期目のプロジェクトとなるFANTOM5では、さまざまな哺乳類細胞のゲノム上の遺伝子制御部位の活性を測定し、転写状態やプロモーター活性の全容を明らかにする研究が進められた。現在のFANTOM6には20カ国、100以上の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。FANTOMはFunctional Annotation of the Mammalian Genomeの略。
詳細はFANTOMのホームページ![]() を参照。
を参照。
4.転移RNA(tRNA)、リボソームRNA(rRNA)
tRNAとrRNAは、メッセンジャーRNA(mRNA)からタンパク質が翻訳される過程で機能するRNA。いずれもタンパク質へは翻訳されないが、その生合成のプロセスや機能はよく研究されている。
5.エピジェネティクス
DNAのメチル化やヒストンのメチル化/アセチル化など、DNAの塩基配列に依存しない遺伝子の調節機構のこと。
6.CAGE法
理研が独自に開発した遺伝子解析技術で、耐熱性逆転写酵素やmRNAのキャップ構造を捕捉する技術を組み合わせて、5’末端の塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせ、どこからどのくらい転写が始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる世界唯一の解析技術。CAGEはCap Analysis of Gene Expressionの略。
7.初代細胞
組織から採取した細胞の培養を開始した場合、約60~80回分裂を繰り返したのち、老化し増殖しなくなる。このような細胞を初代細胞と呼ぶ。生体内と同様の挙動をすることが期待されている。
8.RNAプロセシング
遺伝子から転写されたmRNAの前駆体が成熟した機能分子になるための修飾過程のこと。5’末端へのキャップ構造の付加、3’末端のポリアデニル化、スプライシングなどがある。
9.アンチセンスオリゴヌクレオチド
標的塩基配列に相補的な、一本鎖の合成DNAまたはRNA。遺伝子機能発現を抑制する、アンチセンス法に用いられる。
10.遺伝子機能抑制(ノックダウン)
特定の遺伝子の転写量を減少させることにより、遺伝子の機能を阻害する方法。
11.細胞の遊走
細胞がある場所から他の場所へ移動すること。創傷治癒、胚発生、腫瘍の転移などにおいて重要な働きを持つ。
国際共同研究グループ
理化学研究所
生命医科学研究センター
ゲノム情報解析チーム
研究員ジョーダン ラミロフスキー(Jordan Ramilowski)
遺伝子制御回路研究チーム
研究員 イップ チーワイ(Yip Chi Wai)
技師 安澤 加代子(やすざわ かよこ)
特別研究員(研究当時) ジャスミン リー チン(Jasmine Li Ching)
チームリーダー ジェイ シン(Jay Shin)
応用計算ゲノミクス研究チーム
研修生(研究当時) タンビル アラム(Tanvir Alam)
研究員 ソムヤ アグラワァル(Saumya Agrawal)
技師 ジェシカ ミシェル セヴェリン(Jessica Michelle Severin)
チームリーダー ミヒル デ・ホーン(Michiel de Hoon)
エピゲノム技術研究チーム
研究員 レン チエン チャン(Jen-Chien Chang)
トランスクリプトーム研究チーム
チームリーダー ピエロ カルニンチ(Piero Carninci)
予防医療・診断技術開発プログラム
プログラムディレクター 林崎 良英(はやしざき よしひで)
エジンバラ大学 ロスリン研究所
大学院生 ニック パーキンソン(Nick Parkinson)
トロント大学 コンピューターサイエンス学科
大学院生 ミカエル メンデス(Mickael Mendez)
インペリアル・カレッジ・ロンドン
博士研究員(研究当時) レオニー ローズ(Leonie Roos)
デューク・シンガポール国立大学医学部
博士研究員 ジョン オーヤン(John F Ouyang)
トレント大学統合生物学センター
博士研究員 ヤリ シアーニ(Yari Ciani)
ロシア科学アカデミー エンゲルハルト分子生物学研究所
研究員 イバン クラコフスキー(Ivan V Kulakovskiy)
オールボー大学 医学部
助教授 アンドレアス ペトリ(Andreas Petri)
ジョンスホプキンス大学 医学部
准教授 ルイージ マルキオーニ(Luigi Marchionni)
ニューサウスウェールズ大学 医学部
教授 レボン カチジアン(Levon M. Khachigian)
原論文情報
Tanvir Alam, Saumya Agrawal, Jessica Severin, Robert S. Young, Robin Andersson, Erik Arner, Akira Hasegawa, Marina Lizio, Jordan A. Ramilowski, Imad Abugessaisa, Yuri Ishizu, Shohei Noma, Hiroshi Tarui, Martin S. Taylor, Timo Lassmann, Masayoshi Itoh, Takeya Kasukawa, Hideya Kawaji, Luigi Marchionni, Guojun Sheng, Alistair Forrest, Levon M. Khachigian, Yoshihide Hayashizaki, Piero Carninci, Michiel de Hoon, “Comparative transcriptomics of primary cells in vertebrates”, Genome Research, 10.1101/gr.255679.119
Jordan A Ramilowski, Chi Wai Yip, Saumya Agrawal, Jen-Chien Chang, Yari Ciani,Ivan V Kulakovskiy, Mickaël Mendez, Jasmine Li Ching Ooi, John F Ouyang, Nick Parkinson, Andreas Petri, Leonie Roos, Jessica Severin, Kayoko Yasuzawa, Imad Abugessaisa, Altuna Akalin, Ivan V Antonov, Erik Arner, Alessandro Bonetti, Hidemasa Bono, Beatrice Borsari, Frank Brombacher, Chris JF Cameron, Carlo Vittorio Cannistraci, Ryan Cardenas, Melissa Cardon, Howard Chang, Josée Dostie, Luca Ducoli, Alexander Favorov, Alexandre Fort, Diego Garrido, Noa Gil, Juliette Gimenez, Reto Guler, Lusy Handoko, Jayson Harshbarger, Akira Hasegawa, Yuki Hasegawa, Kosuke Hashimoto, Norihito Hayatsu, Peter Heutink, Tetsuro Hirose, Eddie L Imada, Masayoshi Itoh, Bogumil Kaczkowski, Aditi Kanhere, Emily Kawabata, Hideya Kawaji, Tsugumi Kawashima, S. Thomas Kelly, Miki Kojima, Naoto Kondo, Haruhiko Koseki, Tsukasa Kouno, Anton Kratz, Mariola Kurowska-Stolarska, Andrew Tae Jun Kwon, Jeffrey Leek, Andreas Lennartsson, Marina Lizio, Fernando López-Redondo, Joachim Luginbühl, Shiori Maeda, Vsevolod J Makeev, Luigi Marchionni, Yulia A Medvedeva, Aki Minoda, Ferenc Müller, Manuel Muñoz-Aguirre, Mitsuyoshi Murata, Hiromi Nishiyori, Kazuhiro Nitta, Shuhei Noguchi, Yukihiko Noro, Ramil Nurtdinov, Yasushi Okazaki, Valerio Orlando, Denis Paquette, Callum JC Parr, Owen JL Rackham, Patrizia Rizzu, Diego Fernando Sánchez Martinez, Albin Sandelin, Pillay Sanjana, Colin AM Semple, Youtaro Shibayama, Divya M Sivaraman, Takahiro Suzuki, Suzannah C Szumowski, Michihira Tagami, Martin S Taylor, Chikashi Terao, Malte Thodberg, Supat Thongjuea, Vidisha Tripathi, Igor Ulitsky, Roberto Verardo, Ilya Vorontsov, Chinatsu Yamamoto, Robert S Young, J Kenneth Baillie, Alistair RR Forrest, Roderic Guigó, Michael M Hoffman, Chung Chau Hon, Takeya Kasukawa, Sakari Kauppinen, Juha Kere, Boris Lenhard, Claudio Schneider, Harukazu Suzuki, Ken Yagi, FANTOM consortium, Michiel de Hoon, Jay W Shin, Piero Carninci, “Functional Annotation of Human Long Non-Coding RNAs via Molecular Phenotyping”, Genome Research, 10.1101/gr.254219.119
発表者
理化学研究所 生命医科学研究センター ゲノム情報解析チーム
研究員 ジョーダン・ラミロフスキー(Jordan Ramilowski)
遺伝子制御回路研究チーム
研究員 イップ・チーワイ(Yip Chi Wai) チームリーダー ジェイ・シン(Jay Shin)
応用計算ゲノミクス研究チーム
研修生(研究当時)タンビル・アラム(Tanvir Alam)
チームリーダー ミヒル・デ・ホーン(Michiel de Hoon)
トランスクリプトーム研究チーム
チームリーダー ピエロ・カルニンチ(Piero Carninci)
報道担当
理化学研究所 広報室 報道担当