RNA構造のライブラリ化を通じてRNA構造ごとにおけるRNA-タンパク質相互作用を大規模に解析するシステム”FOREST”の開発
2020-12-09 京都大学iPS細胞研究所
ポイント
- ゲノムに潜むRNA注1)構造データを網羅的に収集した上で、数千-数万種類のRNA構造ライブラリを作成し、 タンパク質との相互作用を生化学的に大規模に解析するシステム・FOREST(Folded RNA Element Profiling with Structure library)を開発した。
- FORESTは、タンパク質がRNAと相互作用する時に、「どのようなRNAの構造を好んで結合するのか?」というこれまで取得が困難であった情報を定量的かつ網羅的に取得できた。
- これまで大規模解析が困難であったRNA高次構造であるRG4構造注2)の定量に成功し、それらと相互作用する3つのタンパク質の結合特異性と強度を明らかにした。
- 得られた結合データを分析し、RG4構造をヒトのマイクロRNA前駆体から新たに見出すことに成功した。
- FORESTはRNA構造と様々な相互作用を大規模に生化学解析可能な幅広いプラットホームとなり、今後RNAに関連する基礎研究や創薬開発など、さまざまな領域で活用が期待できる。
1. 要旨
RNAは生体内で様々な構造をつくることが近年わかってきましたが、それらの構造がどのような機能を持つかを調べるためには、RNA1つずつを生化学的に検証するしかありませんでした。このため、RNA構造の効率的な研究を行うために、RNA標的タンパク質との相互作用を、一度の実験で数千ー数万種類の組み合わせについて定量する「RNA構造が有する機能の大規模解析」の確立が待たれていました。
今回、小松リチャード馨(CiRA未来生命科学開拓部門 大学院生、xFOREST Therapeutics 代表取締役CTO)、樫田俊一 (同社 代表取締役社長CEO、元CiRA特定研究員) および齊藤博英教授(CiRA同部門)らの研究グループは、数千-数万種類のRNA構造が行うRNA-タンパク質相互作用を1度に大規模に解析可能なシステム、FOREST(Folded RNA Element Profiling with Structure Library) を開発しました (図1)。FORESTでは通常の実験で使用するRNA1種類の代わりに、多様な生物種、細胞種、ウイルスゲノム等から抽出したRNAのモチーフ注3群であるRNA構造ライブラリを実験に使用します。ひいては、RNAを標的として結合するタンパク質が、RNA構造ライブラリ中のどのようなRNA構造にどの程度結合するのかという情報を一度の実験で大量に取得することができます。
本研究では、実証実験として、複数のタンパク質を用いてFORESTの実用性の証明と、新しい相互作用の発見を試みました。結果として、RNA構造ライブラリとタンパク質の相互作用を定量なスコアによるランクとして描出できることを確認し、機能をもつRNA構造の発見に至りました。さらに、FORESTは、従来法では解析が難しかったRG4構造の大規模定量に成功しました。そして、複数のRG4結合タンパク質に対するRNA結合強度の定量化が可能になったため、新しくRG4結合タンパク質の結合特性を明らかにできました。応用例として相互作用データをもとにヒトマイクロRNA前駆体からRG4構造を新たに見出すことに成功しました。FORESTは概して、RNA構造の相互作用を大規模に解析する幅広いプラットホームとなり、今後RNAに関連する基礎研究や創薬開発など、さまざまな領域で活用が期待されます。
この研究成果は2020年12月8日午後19時(日本時間)に英国科学誌「Nature Communications」でオンライン公開されました。
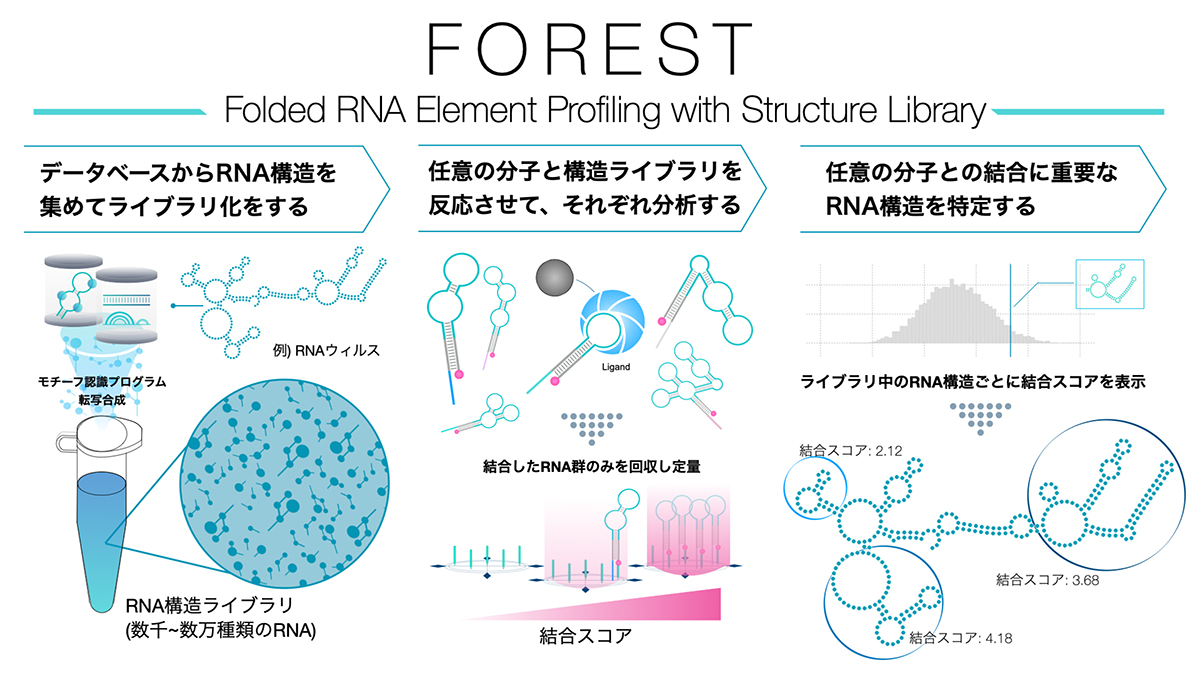
図1:FORESTの概要
FORESTはRNA構造ライブラリを用いて一度に数千ー数万種類の組み合わせの相互作用を定量する。
2. 研究の背景
RNAは配列をもとに複雑な高次構造を形成し、構造単位でユニークな機能を有することが知られています。それゆえ、多種多様なRNAの構造を特定して機能の検証を行うことは、生命を司る特別なRNAを見つけることに繋がります。これまで、並列シーケンサーとRNA生化学実験における技術の進展によって、自然界に存在する多くのRNA構造が明らかになってきました。しかし、RNA構造の種類は膨大であるために、どのような特徴や機能を有しているのかどうかを一つ一つ検証していく手法は費用と時間がかかりました。一方で、RNA構造の機能解析に特化した大規模解析手法は未だ確立されておらず、一度にたくさんのRNA構造を用いて実験することはできませんでした。ひいては、RNAの構造が果たす細胞の役割は多くがわかっていませんでした。
この課題を克服すべく、本研究グループは新たな解析システム・FOREST(Folded RNA Element Profiling with Structure Library) を開発しました。FORESTは、任意の分子を利用した多重化結合アッセイへと展開可能で、1つのチューブで短時間のうちに数千〜数万種類の相互作用を定量することができます。
3. 研究結果
1) FOREST(Folded RNA Element Profiling with Structure Library) の開発
FORESTによる大規模解析を実現するために、まずRNA構造の機能的な単位を大量に収集する必要がありました。そこで、解析したいモチーフを定義し、抽出するためのプログラムを作成しました。その後プログラムによってコンピュータ内で天然に存在するRNA構造のデータセットからモチーフ抽出が自動で行われ、任意のデータからRNA構造を大量に収集できるようになりました。そして生化学実験に使用するために、試験管内転写反応を行い、一本のチューブ内で数千から数万種のRNA構造を含む「RNA構造ライブラリ」を生合成させました(図2)。本研究では実証のためにヒトのマイクロRNA前駆体、メッセンジャーRNA、HIV-1ウィルスのRNA構造を収集し、幅広い種やゲノムのRNAに適用可能であることを示しました。
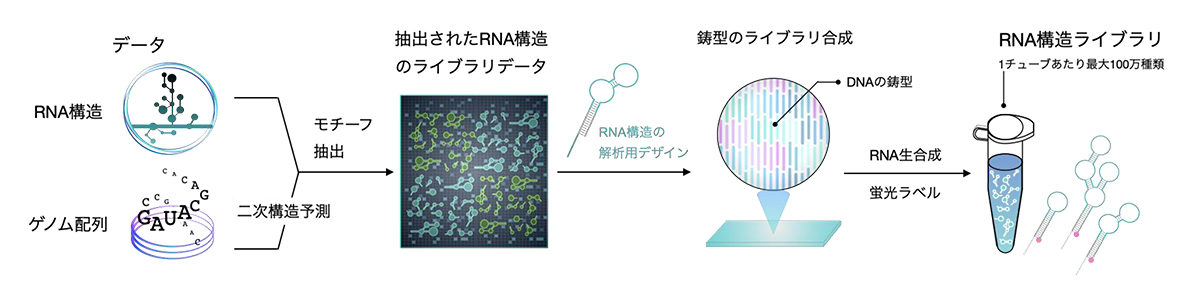
図2:RNA構造ライブラリの開発
生命が持ち得るRNA構造群のデータセットから各構造のモチーフ単位をコンピュータ上で収集し、
それらをライブラリとして生合成する。
あわせて、RNA構造の定量に特化したDNAバーコードマイクロアレイ注4)を開発し、RNA構造ライブラリ中の各RNA構造の量をそれぞれ定量することに成功しました (図3)。ここで、1本のチューブで実験しても1回の実験で数千、数万種類のRNA構造の機能を評価することができる大規模解析を実現できました。くわえて、並列シーケンサー解析に必要なPCR反応や逆転写反応を阻害するため従来の方法では検出が困難であったRNA高次構造 (RG4構造)や疾患に関連するトリプレットリピート注5)を通常のRNA構造と同様に検出することができ、多様な種類のRNA構造の機能を解き明かす基盤が整いました (図4)。

図3:RNA構造ライブラリの大規模定量を実現したDNAバーコードマイクロアレイの開発
RNAバーコードは相補的なDNAバーコードとしか結合しないことを利用し、各RNA構造を指定箇所に配置、その後RNAに付与した蛍光色素を通じて、ライブラリ中の全RNA構造の量を大規模に測定する。
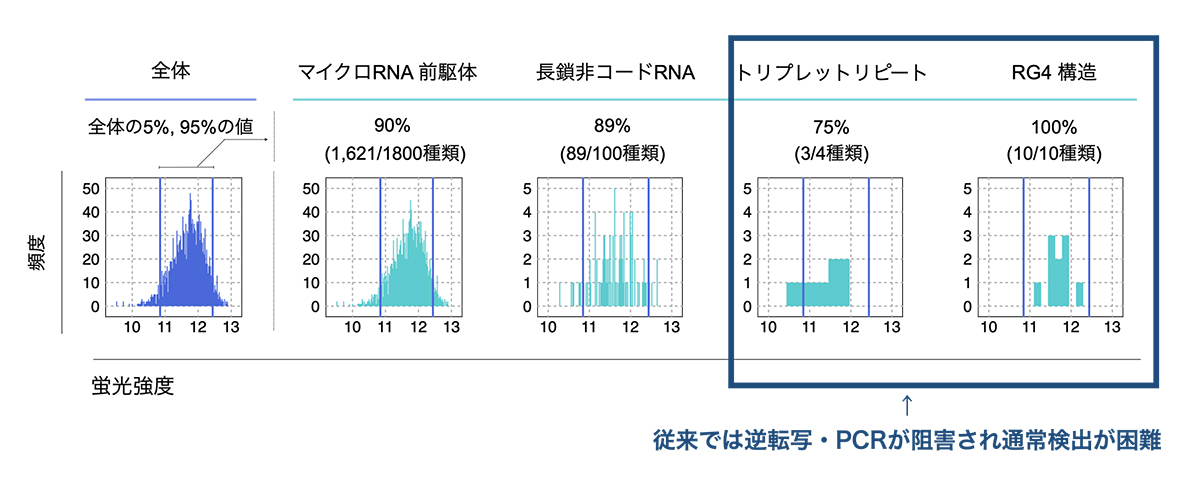
図4:DNAバーコードマイクロアレイによるRNA構造ライブラリの検出・定量
横軸: 蛍光強度 (RNAの量を反映)、縦軸: 頻度を表す。紫の2つの線は全体の5%-95%の範囲を表す。
検出が困難なRG4構造は、他のクラスのRNAと同等に検出可能であった。
2) RNA-タンパク質相互作用解析への展開
RNAの代表的な機能的な役割として、タンパク質と結合し、両者で細胞の運命を制御することがあげられます。しかしその結合に必要なRNA構造が不明であるケースや、RNA構造との結合特異性注6)が不明である種類のタンパク質もありました。そこで、本研究グループはタンパク質に結合したライブラリ中の各RNA構造を検出し、かつその量を測定することで、FORESTでタンパク質とRNA構造の結合を測定、結合特異性の違いを評価できると考えました。まず、このコンセプトを実証するために、ヒトのタンパク質を複数種類用いて、既知の文献や実験データと一致することを調べ、FORESTはRNA構造-タンパク質の相互作用に適用可能ということを証明しました。例えば、U1Aタンパク質は、既知のコントロールであるsnRNA由来のステムループがもっともよく結合すること、そしてその変異体が結合しないこと、GCACという配列が重要であることを確かめました。またLIN28Aタンパク質においても既知の結合群である let-7 ファミリーに属するマイクロRNA 前駆体が結合することを確認しました。同時にEMSA法を用いてU1AとLIN28Aの結合スコアと実際の結合親和性が相関することを実験的に示しました。他にも細胞由来のRoquinやEIF3タンパク質複合体に適用し、様々な実験条件や分子でFORESTが機能したことを確認し、FORESTの有用性と汎用性を確認しました。
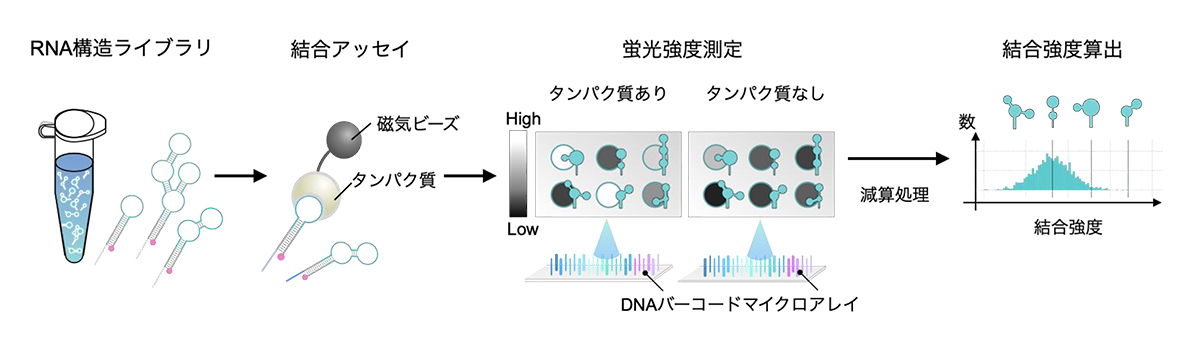
図5:ライブラリ規模でのRNA-タンパク質結合アッセイ
タンパク質は磁気を帯びたビーズに結合しているため、RNAはそれらを介して磁石に引き寄せられる。
その後、磁石に引き寄せられたタンパク質結合群をDNAバーコードマイクロアレイで定量する。
任意のタンパク質1つにつき、数千-数万種類のRNAに対する結合強度の数値が算出される。
3)RG4構造-タンパク質相互作用解析とその応用
次に本研究グループはRG4構造とそれに結合するタンパク質に着目しました (図6a)。これまでの大規模解析手法ではRG4の定量に課題があったため、RG4結合タンパク質の相互作用の詳細は不明でした。まずFORESTを使って、RG4と相互作用する報告がある3つの結合タンパク質 (BG4、CIRBP、DHX36) とRNA構造の相互作用を調べました (図6b)。結果、3つの結合タンパク質すべてにおいて、RG4構造のコントロールがマイクロRNA前駆体のループ構造よりも平均的に結合強度が高いことが分かりました (図6c左)。これは、3つともRG4と結合することを意味します。しかし、その相互作用の詳細は違っており、DHX36が最もRG4に対するRNA-タンパク質結合が強く、BG4が最も結合が弱いことが分かりました。さらに、驚くことに、BG4はRG4でなくても特定のRNAに結合すること、CIRBPはRG4らしさの指標と高い相関を示すがGAAの繰り返し(リピート)配列に強く結合すること、DHX36はRG4に対して極めて高い特異性で結合していることがわかりました (図6c-d)。このように同じRG4構造を認識するとされていたタンパク質でも、その相互作用の程度や好みに違いがあることを突き止めました。
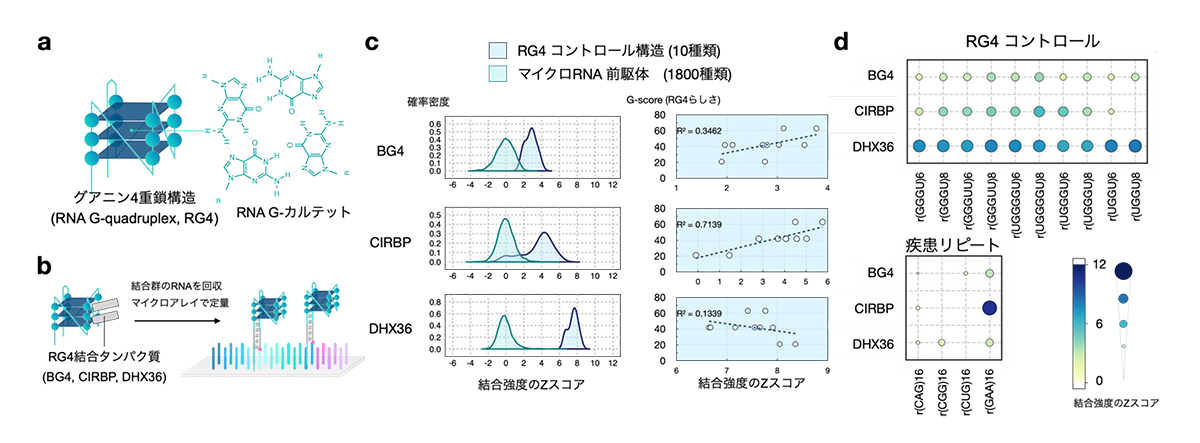
図6:FORESTによって明らかになったRG4結合タンパク質の結合特性
(a) RG4構造の図 (b) RG4結合タンパク質の解析 (c) FORESTで測定した結合強度のヒストグラム (緑: マイクロRNA 前駆体, 青: RG4コントロール)とRG4コントロールとRG4らしさの指標(G-score)の相関プロット
(d) 結合強度のバブルヒートマップ。円が大きくなるほど結合強度スコアが高い。

図7: RG4指示薬であるNMMアッセイによるRG4候補の検証実験
FORESTのデータからRG4候補として選ばれた4つのマイクロRNA 前駆体 (赤枠)。
配列中の青字はRG4を形成に重要なGの連続を示す。
最後に、RG4の探索にFORESTのデータを応用しました。RG4を形成できる領域の探索手法には長らく課題があり、RG4が存在するゲノムの領域はその多くがはっきりとわかっていませんでした。さらに、どこからどこまでがRG4なのかどうかという明確なRG4の構造的境界の情報を伴った大規模解析は未だ実現できていません。しかし、FORESTによる解析でCIRBP結合スコアがRG4の指標と相関したこととDHX36がRG4に対して高い特異性を持つと判明したことから、本研究グループはFORESTの特異的な結合データを統合することでRNA構造ライブラリからRG4構造を見つけられると着想しました。この仮説を実証するために、ヒトのマイクロRNA前駆体のループ構造のうち、RG4を形成できるものをCIRBPとDHX36の結合スコアを頼りに探索しました。結果的に、FORESTで見出したRG4候補である4つのマイクロRNA前駆体ループ構造は、NMMというRG4の指示薬で高い蛍光を示しました (図7)。この結果はこれら4つのRNAはRG4構造を有することを意味しています。これにより、FORESTは通常では判別できない特定の構造に対して高い特異性を持つ分子を明らかにし、それらデータの解析で特定のRNA構造を新しく発見できることを証明しました。
4. 本研究の意義と今後の課題
本研究では、RNA構造-タンパク質相互作用の解析を大規模に実行するためのシステム・FORESTを新たに開発しました。いくつかのタンパク質をモデルとして用いて、FORESTは相互作用を定量化できること、結合を行う新しいRNA構造を発見すること、RNA構造への結合特異性の違いを調べられること、そしてRG4の定量と発見をできることを示しました。今後、FORESTは、タンパク質の結合に重要なRNA構造をゲノムの中から自在に探索できる幅広いプラットホームとなり、RNAに関連する基礎研究や創薬開発の促進など、さまざまな領域で活用が期待されます。
5. 論文名と著者
- 論文名
RNA structure-wide discovery of functional interactions with multiplexed RNA motif library. - ジャーナル名
Nature Communications - 著者
Kaoru R. Komatsu1*, Toshiki Taya2, Sora Matsumoto1, Emi Miyashita1, Shunnichi Kashida1**
and Hirohide Saito1**
*:筆頭著者、**:責任著者 - 著者の所属機関
- 京都大学iPS細胞研究所(CiRA)
- ツイストバイオサイエンス社 (サンフランシスコ、米国)
6. 本研究への支援
本研究は、下記機関より支援を受けて実施されました。
- 日本学術振興会(JSPS)科研費[15H05722][17H05601][17J09014][19K22387]
- 公益財団法人 三菱財団
- 公益財団法人 内藤記念科学振興財団
7. 用語説明
注1)RNA
リボ核酸のこと。細胞内で様々な役割を担っており、DNAの塩基情報がRNA(メッセンジャーRNA)に転写され、その情報をもとにタンパク質が合成される。
注2) RG4 (RNA G-quadruplex, グアニン四重鎖)
塩基の1つであるグアニンは隣接した位置に連続して存在するとき、フーグスティーン塩基対を形成し、四重鎖構造を形成できる。この構造では、グアニン塩基が水素結合により平面構造(G-カルテット)をなし、それが積み重なっている形をした状態で安定している。RG4は遺伝子の発現や細胞の機能に大きく関わっていることが知られている。
注3)モチーフ
偶然起こったとは考えにくい確率で自然界、生物の中で頻出する単位のこと。本研究ではRNAの配列と構造、またはその両方から定義されるものを指す。モチーフの発見をして、その役割を調べることはRNAが行う細胞内の現象の発見に繋がる。
注4)DNAバーコードマイクロアレイ
スライドガラスの一面にわたって多種類のDNAを極小のスポット状に無数に生やしたものを通常DNAマイクロアレイと呼ぶ。そのDNAをRNA構造ライブラリ中のRNAが有するバーコード配列と相補的になるように本研究グループが開発・設計したもの。これにより、RNA構造ライブラリ中の各RNA構造を指定したスポットに配置し、定量する大規模解析が可能になる。
注5)トリプレットリピート
特定の3つの塩基を一つの単位とした繰り返し配列。一部の疾患においてこれらの繰り返し回数の伸長が認められる。本研究ではGAA, CUG, CGG, CAGリピートを実際に解析に使用した。
注6)特異性
ここでは複数の種類の物質が混在する中で、ある特徴をもつ標的分子だけに結合できるかどうかの指標を意味する。