2024-10-08 京都大学
新型コロナウイルス(SARS-CoV-2)「オミクロン株」は次々と変異を繰り返して流行の波を形成しています。橋口隆生 医生物学研究所教授と矢島久乃 薬学研究科博士課程学生、前仲勝実 北海道大学教授と安楽佑樹 同博士課程学生、佐藤佳 東京大学教授らの研究グループは、昨年流行したXBB系統から30箇所以上の変異をSpike蛋白質に獲得し、新たな変異株として今年流行の大きな波を形成したBA.2.86とJN.1のSpike蛋白質の構造を、クライオ電子顕微鏡を用いて解明しました。現在流行中の変異株であるKP.3などはこの子孫系統になります。
構造的な特徴として、ウイルスが細胞に感染する際に最初に細胞の入口として利用する受容体ACE2に対して、これまでに観測されたことのない様式でもウイルスが受容体に結合できる様子(中間体構造)が観察されました。さらに、獲得した変異のうち、K356Tという変異が中和抗体からの逃避能を高めていることが分かりました。
新しい変異株が感染力を高めたり、抗体から逃避する変異を獲得した理由を説明できる構造情報が得られたことで、病原性の理解と変異株対応ワクチン開発などの創薬研究への進展が期待されます。一連の構造情報は、国際的な構造データベースであるProtein Data Bank(PDB)とElectron Microscopy Data Bank(EMDB)を通じて世界中の研究者が無償で利活用することができます。
本研究成果は、2024年10月7日に、国際学術誌「Nature Communications」にオンライン掲載されました。
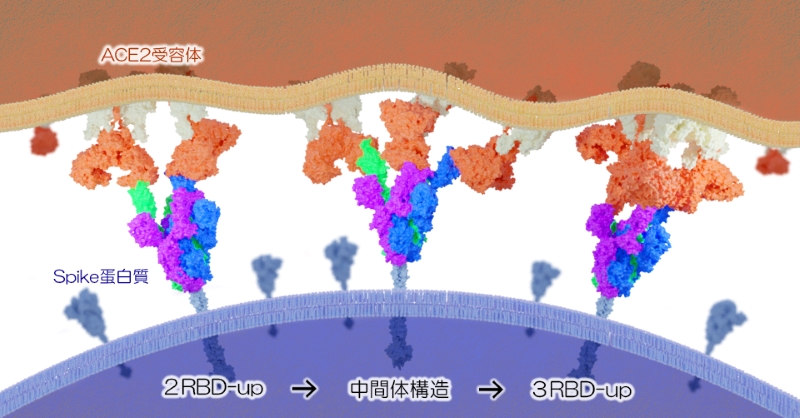 受容体結合に伴うウイルスSpike蛋白質の構造変化
受容体結合に伴うウイルスSpike蛋白質の構造変化
詳しい研究内容について
新型コロナウイルス変異株の構造特性を解明―流行株の変化を原子レベルで可視化―
研究者情報
研究者名:橋口 隆生
研究者名:矢島久乃
書誌情報
【DOI】
https://doi.org/10.1038/s41467-024-52808-2
【書誌情報】
Hisano Yajima, Yuki Anraku, Yu Kaku, Kanako Terakado Kimura, Arnon Plianchaisuk, Kaho Okumura, Yoshiko Nakada-Nakura, Yusuke Atarashi, Takuya Hemmi, Daisuke Kuroda, Yoshimasa Takahashi, Shunsuke Kita, Jiei Sasaki, Hiromi Sumita, The Genotype to Phenotype Japan (G2P-Japan) Consortium, Jumpei Ito, Katsumi Maenaka, Kei Sato, Takao Hashiguchi (2024). Structural basis for receptor-binding domain mobility of the spike in SARS-CoV-2 BA.2.86 and JN.1. Nature Communications, 15, 8574(2024).