2022-08-19 理化学研究所,東京大学定量生命科学研究所
理化学研究所(理研)生命機能科学研究センター転写制御構造生物学研究チームの江原晴彦研究員、関根俊一チームリーダー、東京大学定量生命科学研究所クロマチン構造機能研究分野の鯨井智也助教、胡桃坂仁志教授らの共同研究グループは、真核細胞の遺伝子発現を担うRNAポリメラーゼII(RNAPII)[1]が、メッセンジャーRNA(mRNA)[2]の転写に際して、いったんほどいたヌクレオソーム[3]を転写直後に組み立て直すことで、クロマチン[3]構造を壊さずに転写[1]を行う仕組みを解明しました。
本研究成果は、真核細胞の核内では遺伝子発現とクロマチン構造の維持がどのように両立しているのかという生物学上の大きな謎に答えるものであり、今後、転写制御やその破綻による疾患メカニズムの理解への発展が期待できます。
真核生物のDNAはヒストンタンパク質[3]と結合してヌクレオソーム構造を形成し、さらに複数のヌクレオソームが数珠状に連なったクロマチンと呼ばれる高次構造をとって、細胞核内に収納されています。mRNAの転写を担う酵素であるRNAPIIは、DNAを転写する際にヌクレオソームをほどく必要がありますが、ほどかれたヌクレオソーム構造が再形成される仕組みは不明でした。
今回、共同研究グループは「クライオ電子顕微鏡[4]」を用いて、RNAPIIが複数のタンパク質(転写伸長因子[5]やヒストンシャペロン[6])の助けを借りてヌクレオソームを通過する様子を捉えました。その結果、RNAPIIが前方のヌクレオソームをいったん解体し、後方で組み立て直す仕組みが初めて明らかになりました。
本研究は、科学雑誌『Science』オンライン版(8月18日付:日本時間8月19日)に掲載されました。
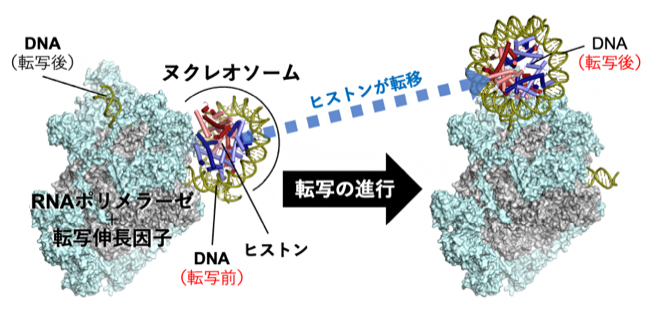
ヌクレオソームを通過する前後を捉えた転写伸長因子複合体の構造
背景
私たちヒトを含む真核生物の細胞では、生命の設計図であるゲノムDNAは「クロマチン」と呼ばれる構造をとって、核の中にコンパクトに収納されています。クロマチンはDNAとタンパク質の複合体であり、八つのヒストンタンパク質(ヒストン8量体)の周りにDNAがおよそ2回巻きついた糸巻きのような形の「ヌクレオソーム」が数珠状に多数連なった構造をしています。クロマチン構造は、ゲノムDNAを物理的に保護するだけではなく、DNAのどの部分をどのタイミングで読み出すか(=転写)を指定する重要な機能も担っています。
DNAの転写を行うのは、RNAポリメラーゼというタンパク質(酵素)です。転写の際には、RNAポリメラーゼがその通り道にあるヌクレオソームを解体してDNAをほどきながら進む必要があり、転写はクロマチン構造を壊す働きであるといえます。しかし実際の細胞内では、RNAポリメラーゼはクロマチン構造を維持したままDNAの転写を行っており、この矛盾を真核細胞がどのように解決しているのかは生物学における長年の謎でした。
本研究では、クライオ電子顕微鏡を用いて、タンパク質の設計図であるメッセンジャーRNA(mRNA)の転写を担当するRNAポリメラーゼII(RNAPII)がヌクレオソームを通過する際の構造をコマ撮りのように捉えることで、その謎の解明に挑みました。
研究手法と成果
共同研究グループはこれまでに、RNAPIIにヌクレオソームを転写させ、その途中で形成される複合体の構造をクライオ電子顕微鏡を用いて解析する手法を開発し、RNAPIIが段階的にヒストンからDNAを剥がしながら進んでいく様子を明らかにしてきました注1~3)。今回の研究では、RNAPIIがヌクレオソームDNAの中央部(dyad)を通過する際にヌクレオソームがどういった運命をたどるかに焦点を当て、以下の実験を行いました。
まず、RNAPIIがヌクレオソーム上のDNAの転写を開始してから4カ所の位置で停止するようにヌクレオソームDNAを設計しました。この設計では、RNAPIIがヌクレオソームに進入する入口から数えて42、49、58、115番目の塩基対(bp)の位置で転写を停止させることで、RNAPIIの先端がそれぞれdyadの直前(42bp)、dyad近傍(49bp)、dyadの直後(58bp)、ヌクレオソームの出口付近(115bp)に来ることが期待されます。これらのヌクレオソームとRNAPII、および細胞内での転写に必要な転写伸長因子とヒストンシャペロンFACT[6]の存在下で転写を行い、形成された複合体の構造をクライオ電子顕微鏡によって解析しました。その結果、DNA上の四つの位置で停止した6種類の複合体の構造が得られました(図1)。
これらの構造では、RNAPIIは転写伸長因子を結合して巨大な転写伸長複合体(EC)を形成していました。主要な転写伸長因子を全て結合した転写伸長複合体の構造解析はこれが初めてです。転写伸長因子はRNAPIIのほぼ全面を覆っており、DNAの入口や出口を補強してRNAPII内をDNAが通過する「トンネル」構造を再構築していました。
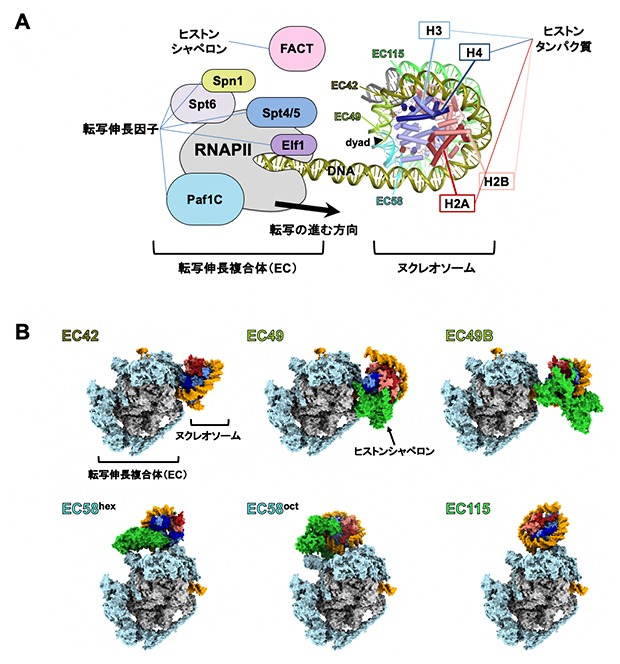
図1 ヌクレオソームを通過するRNAポリメラーゼと転写伸長因子の複合体
A)本実験で再構成したRNAポリメラーゼII(RNAPII)と転写伸長因子(左)、ヒストンシャペロンFACT、およびヌクレオソーム(右)の模式図。転写伸長複合体がヌクレオソームに進入し、B)で示した構造で停止するまでに転写されるDNAを色分けし、ヒストンから剥がれるDNAの先端を色矢印で示した。ヌクレオソームに入ったRNAPIIは、まずdyadの直前(42bp)までの濃緑色のDNAを転写し、次に緑色のDNAを転写してdyad近傍(49bp)で停止。その後、dyadの直後(58bp:水色)、ヌクレオソームの出口付近(115bp:明緑色)で停止する。
B)RNAPII+転写伸長因子(転写伸長複合体:EC)とヌクレオソームの複合体をクライオ電子顕微鏡で解析して得られた立体構造。灰色はRNAPII、水色は転写伸長因子、その他はヌクレオソーム(DNAとヒストンタンパク質)、ヒストンシャペロンを示す。42bp、115bp停止位置では1種類の構造(EC42、EC115)が得られ、49bpと58bpの停止位置ではヒストンシャペロン(緑色)も複合体に加わり、それぞれ2種類の構造が得られた(EC49とEC49BおよびEC58hexとEC58oct)。
次に、得られた複合体の構造を転写が進行していく順番に並べることで、転写伸長複合体がヌクレオソームを通過していく際のスナップショットを得ることに成功しました。この一連のスナップショットは、転写伸長複合体がその下流(これから転写するDNA)にあるヌクレオソームに侵入し、DNAをヒストンから徐々にほどいていき、dyadを通過した時点でヒストンを上流側(転写が終わったDNA)に転移させ、DNAの出口の部分でヌクレオソームを再形成していく様子を明確に捉えていました(図2)。
このように、RNAPIIの転写伸長複合体には、進行方向(下流)にあるDNAを転写する際に一度解体したヌクレオソームを、転写後速やかにRNAPIIの背後(上流)で再生させる仕組みが備わっていることが明らかになりました。特にDNAの出口の周囲では、転写伸長因子がヌクレオソームの「ゆりかご」を形成し、段階的なヌクレオソーム再形成のプロセスを支えていることも分かりました(図2①、⑦)。
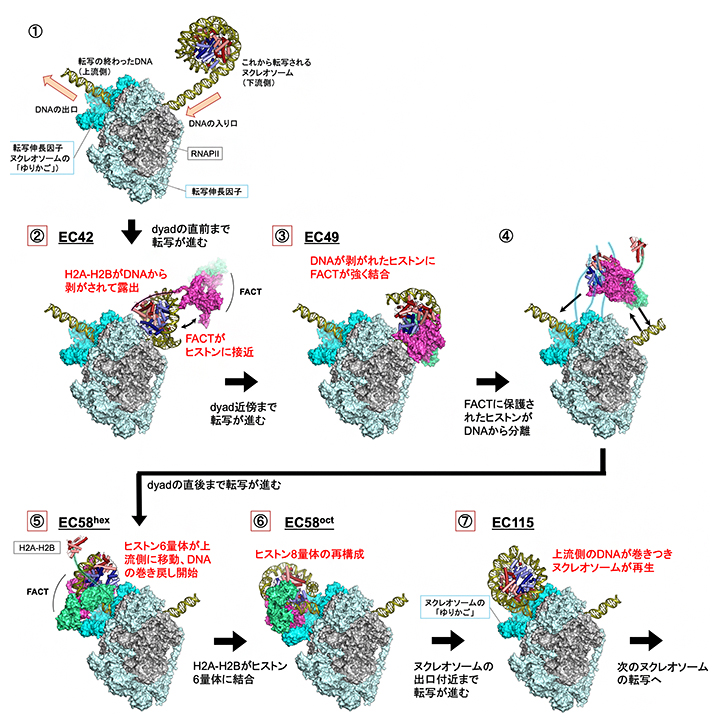
図2 転写伸長中に下流のヌクレオソームをいったん解体し、上流で再生させる仕組み
①ヌクレオソームDNAの転写が始まる前の状態。
②転写伸長複合体(EC)の前進に伴って、ヒストンの表面からDNAが引き剥がされていく。H2A-H2Bが露出すると、尾部を使ってヒストンシャペロンFACTがヒストンにアクセスを開始する(EC42)。
③ECがヌクレオソームの中央部(dyad)近傍にまで達すると、DNAが剥がれたヒストンにFACTが強く結合する(EC49)。
④FACTに保持されたヒストンがDNAから分離し、上流に転移する。FACTの本体はヒストン6量体(H2A-H2B-(H3-H4)2)の部分をしっかりとつかんでおり、残りのH2A-H2BはFACTの尾部につなぎ止められていると考えられる。
⑤FACTに保持されたヒストン6量体(H2A-H2B-(H3-H4)2)がDNAの出口付近でDNAに結合し(EC58hex)、ヌクレオソームの巻き直しが始まる。
⑥残るヒストン2量体(H2A-H2B)が加えられ、ヒストン8量体((H2A-H2B)2-(H3-H4)2)が完成する(EC58oct)。
⑦最後に、ECの前進に伴ってヒストン8量体の大部分がDNAで覆われ、ほぼ完全なヌクレオソームが再生する(EC115)。DNAの出口の周囲では、転写伸長因子がヌクレオソームと相互作用することで段階的なヌクレオソーム再形成のプロセスを支える「ゆりかご」の役割を果たしている。
以上のように、RNAPIIがヌクレオソームを通過する際に、ヒストンの下流から上流への受け渡しが起こり、結果としてRNAPIIは何事もなかったかのようにヌクレオソームを通過し、転写を継続することができます(図3)。転写の前後で同じヒストンがヌクレオソームの形成に参加することを示す今回の発見は、クロマチン構造やヒストン修飾などのエピジェネティック情報[7]を維持しつつ転写が行われる仕組みを明らかにするものです。
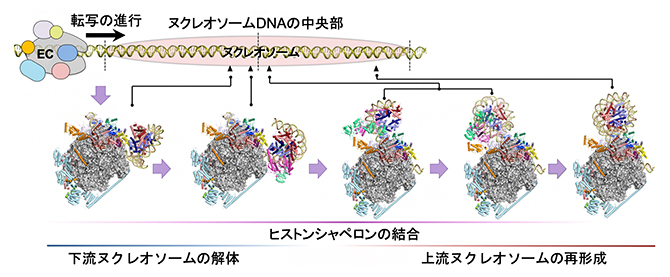
図3 本研究のまとめ
転写伸長因子複合体(EC)は、ヌクレオソームを通過する際、ヌクレオソームをいったんほどいてヒストンを下流から上流へ受け渡す。これにより、転写が終わると直ちにヌクレオソームが再形成される。
注1)2019年2月8日プレスリリース「コンパクトなDNAをスムーズに転写する仕組み」
注2)2018年10月5日プレスリリース「真核生物での遺伝子読み取りの仕組みを解明」
注3)2017年8月4日プレスリリース「転写中のRNAポリメラーゼIIの構造を解明」
今後の期待
クロマチン構造は、細胞が特定の遺伝子セットを発現するための情報(エピジェネティック情報)を備えており、同じ遺伝子を転写し続けるためには転写の前後でクロマチンの基本構造であるヌクレオソームが保持される必要があります。本研究は、RNAPIIが他の複数の因子の助けを借りて前方にあるヌクレオソームをいったん解体し、その背後で組み立て直すことを初めて明らかにしました。これは、転写によるクロマチン構造への影響を回避する仕組みと考えられますが、一方で、転写がクロマチン構造に影響を与える場合があることも知られています。
細胞内では、今回の解析で用いた転写伸長因子やヒストンシャペロンとともに、エピジェネティック情報の書き換えを行うリモデリング因子[8]やヒストン修飾酵素[9]などが連携して働き、クロマチン構造を制御しています。今回明らかになったヌクレオソームを通過中の転写伸長複合体の構造は、これらのクロマチン制御因子が作用する標的構造となっている可能性があり、今後のエピジェネティクス[7]や転写制御の研究を飛躍させる重要な基礎となると期待できます。
また、本研究で転写伸長における役割が明らかになったヒストンシャペロンFACTは、RNAの転写だけでなく、DNAの複製や修復、組換えなどにも関与していることが知られています。今回の観察結果は、ヌクレオソームからDNAをほどく反応を伴う核内機能の普遍的な理解につながる可能性があります。
ヌクレオソームを転写する転写伸長複合体のような細胞内の状態を反映した巨大複合体は、構造解析が非常に難しく、これまで取り残されていた創薬対象の一つです。本研究で得られた知見から、今後、転写制御やクロマチン構造の破綻による疾患や老化のメカニズムについての研究が発展するものと期待できます。
補足説明
1.RNAポリメラーゼII(RNAPII)、転写
DNA配列を鋳型にRNAが合成されることを転写と呼び、細胞内ではDNA依存性RNAポリメラーゼ(RNAポリメラーゼ)により行われる。真核生物は3種のRNAポリメラーゼを持ち、RNAポリメラーゼII(RNAPII)はmRNAの合成を担当する。複数のタンパク質(サブユニット)が集合してできた巨大な複合体で、細菌からヒトまで共通した”カニのはさみ”のような形をしている。中央にできた溝にDNAを挟み込み、およそ10塩基対のDNAをほどいて「転写バブル」を形成し、一方の鎖を鋳型にしてmRNA前駆体を合成する。
2.メッセンジャーRNA(mRNA)
細胞が合成するタンパク質のアミノ酸の並び方の情報(コドン)を持つRNA。真核生物では、DNAから転写された直後のmRNA前駆体に含まれるタンパク質情報を持たない配列(イントロン)を除去して、成熟したmRNAが作られる。
3.ヌクレオソーム、クロマチン、ヒストンタンパク質
DNAを巻き付けることで、長大なDNAを核内に納めるタンパク質をヒストンという。H2A、H2B、H3、H4の4種類のコアヒストンが存在し、それらが二つずつ集まってヒストン8量体を形成する。ヒストン8量体の周りにDNAが巻き付いた構造をヌクレオソームと呼び、真核生物においてヌクレオソームを基本単位とするゲノムDNAとタンパク質の高次複合体をクロマチンと呼ぶ。
4.クライオ電子顕微鏡
タンパク質などの生体試料を観察するために開発された電子顕微鏡。タンパク質などの試料を含んだ溶液を薄く展開し、液体エタン(-183~-160℃)中で急速凍結して試料をごく薄い氷の層に閉じ込めた上、液体窒素温度(-196℃)で電子顕微鏡により観察する。タンパク質試料を生理的な(細胞の状態に近い)条件で観察できる、低温のため電子線による試料の損傷が軽減される、といった利点がある。近年著しい発展を見せている構造解析技術であり、2017年のノーベル化学賞の対象ともなった。本研究では、理化学研究所(横浜地区)と東京大学(定量生命科学研究所)に設置されているクライオ電子顕微鏡を用いて解析を行った。
5.転写伸長因子
RNAポリメラーゼに結合し、転写伸長反応を制御するタンパク質。本研究では、主要な転写伸長因子であるSpn1、Spt6、Spt4/5、Elf1、Paf1C、TFIISを全て用いて構造解析を行った。
6.ヒストンシャペロン、FACT
シャペロンは、新生タンパク質が正常な立体構造をとるのを補助したり、変性したタンパク質を認識して正常な構造に戻す、あるいは除去する機能を持つ因子の総称。ヒストンシャペロンは、特にヒストンとDNAの結合や解離に関わる因子を指す。本研究では、ヒストンシャペロンであるFACTを構成するSpt16とPob3を用いて構造解析を行った。FACTは、facilitates chromatin transcriptionの略。
7.エピジェネティック情報、エピジェネティクス
DNAの塩基配列に依存しない遺伝子の調節機構をエピジェネティクスと呼ぶ。エピジェネティクスの基本的な分子基盤は、DNAのメチル化やヒストンのメチル化/アセチル化といったゲノムの特定領域に可逆的につけられた「目印」であり、このメチル化やアセチル化の情報をエピジェネティック情報と呼ぶ。
8.リモデリング因子
クロマチンの構造変化を引き起こす因子の総称。エピジェネティクスの制御に関わる因子の一つ。
9.ヒストン修飾酵素
ヒストンタンパク質の化学修飾を触媒する酵素の総称。可逆的なアセチル化修飾を担うアセチル基転移酵素と脱アセチル化酵素、可逆的なメチル化修飾を担うメチル基転移酵素と脱メチル化酵素などがある。
共同研究グループ
理化学研究所 生命機能科学研究センター
転写制御構造生物学研究チーム
チームリーダー 関根 俊一(セキネ・シュンイチ)
研究員 江原 晴彦(エハラ・ハルヒコ)
タンパク質機能・構造研究チーム
チームリーダー 白水 美香子(シロウズ・ミカコ)
東京大学 定量生命科学研究所 クロマチン構造機能研究分野
教授 胡桃坂 仁志(クルミザカ・ヒトシ)
助教 鯨井 智也(クジライ・トモヤ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「転写と共役したヌクレオソーム再構築の分子メカニズムの解明(研究代表者:江原晴彦)」、同基盤研究(S)「転写と中核的な生命機能を結びつける高次複合体の構造基盤(研究代表者:関根俊一)」、同基盤研究(A)「クロマチン上で起こる転写と共役した二重鎖切断修復の分子機構の解明(研究代表者:胡桃坂仁志)」、同新学術領域研究(研究領域提案型)「ヌクレオソーム高次構造とダイナミクスの解析によるクロマチン潜在能の解明(研究代表者:胡桃坂仁志)」、科学技術振興機構(JST)戦略的創造研究推進事業(ERATO)「胡桃坂クロマチンアトラスプロジェクト(研究総括:胡桃坂仁志)」、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)「統合的構造解析に向けた高難度複合体の生産支援と高度化(代表者:白水美香子)」「エピジェネティクス研究と創薬のための再構成クロマチンの生産と性状解析(代表者:胡桃坂仁志)」、生命科学・創薬研究支援基盤事業(BINDS)「エピジェネティクスの基盤原理解明と創薬のためのヒストンおよび再構成クロマチンの生産(代表者:胡桃坂仁志)」、および公益財団法人住友財団基礎科学研究助成「リモデリング因子SMARCAD1によるクロマチン構造変換と転写活性化機構の解明(助成対象:鯨井智也)」の支援を受けました。
原論文情報
Haruhiko Ehara†, Tomoya Kujirai†, Mikako Shirouzu, Hitoshi Kurumizaka* and Shun-ichi Sekine*(†共同筆頭著者、*責任著者), “Structural basis of nucleosome disassembly and reassembly by RNAPII elongation complex with FACT”, Science, 10.1126/science.abp9466
発表者
理化学研究所
生命機能科学研究センター 転写制御構造生物学研究チーム
チームリーダー 関根 俊一(セキネ・シュンイチ)
研究員 江原 晴彦(エハラ・ハルヒコ)
東京大学 定量生命科学研究所 クロマチン構造機能研究分野
教授 胡桃坂 仁志(クルミザカ・ヒトシ)
助教 鯨井 智也(クジライ・トモヤ)
報道担当
理化学研究所 広報室 報道担当
東京大学 定量生命科学研究所 総務チーム