2023-01-16 理化学研究所
理化学研究所(理研)環境資源科学研究センター 合成ゲノミクス研究グループの栗原 志夫 客員研究員、蒔田 由布子 客員研究員、松井 南 グループディレクターらの研究チームは、植物における遺伝子間スプライシング[1]による「ポリシストロニックなリードスルー転写物(複数の遺伝子から転写された異常なmRNA)」の蓄積が抑制される仕組みを明らかにしました。
本研究成果は、異常転写物の蓄積による毒性の除去に貢献するだけではなく、植物バイオマスに有用な技術の創出に寄与すると期待できます。
真核生物では、通常、一つの遺伝子から一つのタンパク質をコードする「モノシストロニックな転写物」が生成されますが、まれに隣接する複数の遺伝子にまたがり、複数のタンパク質をコードするポリシストロニックなリードスルー転写物が生成されることがあります。
今回、研究チームはロングリードシークエンサー[2]を用いて、シロイヌナズナ芽生えのポリシストロニックなリードスルー転写物が「ナンセンスコドン介在的mRNA分解(NMD)[3]」というRNA品質管理機構によって、選択的に細胞から除去されることを明らかにすることに成功しました。
本研究は、オンライン科学雑誌『Communications Biology』(12月20日付)に掲載されました。
背景
「ナンセンスコドン介在的mRNA分解(NMD)」は、未成熟終止コドン[4]を持つ転写物を選択的に分解するRNA品質管理機構の一つです。転写物の品質管理がおろそかになり、異常な転写物が細胞内に過剰蓄積してしまうと、形態異常の原因となったり、生命を維持できなくなったりします。従って、NMDがどのような転写物を分解の標的とするのかを知ることは、植物の遺伝子発現制御を理解するために重要です。
真核生物では、通常、一つの遺伝子から一つのタンパク質をコードする「モノシストロニックな転写物(mRNA)」が得られます。まれに、隣接する複数の遺伝子にまたがり、複数のタンパク質をコードする「ポリシストロニックなリードスルー転写物」が生成されることがあります。つまり、何らかの理由によって第一遺伝子の転写終結[5]が回避され、下流の第二遺伝子以降にまで転写が続いてしまうリードスルー転写が起こり得ます。これまで、そのようなリードスルー転写物がNMDの標的になると予測されてきました。
しかし、リードスルー転写物の量が極めて少ないことや転写物全長を網羅的に検出する方法が確立されていなかったことから、リードスルー転写物の発現制御のメカニズムは不明でした。
研究手法と成果
シロイヌナズナの変異体upf1-1は、NMDの必須因子であるUPF1タンパク質をノックダウン[6]させた変異体です。研究チームはまず、パックバイオ社のロングリードシークエンンサーを用いて、野生型と変異体upf1-1をそれぞれ暗所で3日間育成した芽生えと、暗所で3日間育成後に青色光を3時間照射した芽生えに蓄積するリードスルー転写物のゲノム全域にわたる探索を試みました。青色光は植物の生長に関わる主要な光波長であり、これにより青色光の受容によって転写される転写物を検出することができます。解析の結果、リードスルー転写物の生成に関連する271カ所の遺伝子座を同定することができました(図1左)。
さらに、NMDのもう一つの必須因子であるUPF3タンパク質をノックダウンさせた変異体upf3-1も研究に用いました。野生型、変異体upf1-1、変異体upf3-1でリードスルー転写物の蓄積を比べたところ、変異体upf1-1と変異体upf3-1で多くのリードスルー転写物が蓄積されていることが分かりました(図1右)。この結果は、リードスルー転写物がNMDの標的分子であることを示しています。
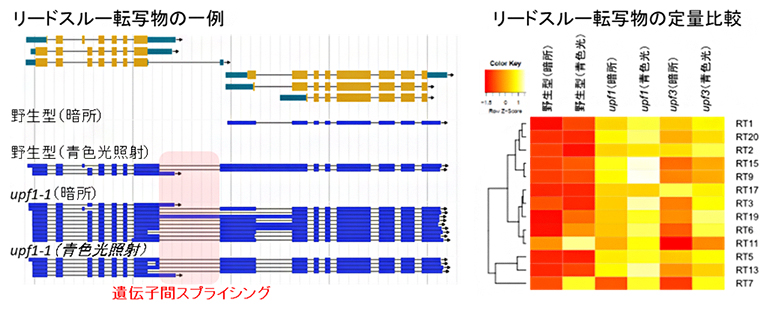
図1 ポリシストロニックリードスルー転写物はNMDの分解標的となる
(左)ロングリードシークエンサーを用いて、シロイヌナズナにおいてポリシストロニックなリードスルー転写物の生成に関連する271カ所の遺伝子座を同定した。
(右)野生型とNMDの必須因子の変異体upf1-1とupf3-1で、リードスルー転写物(RT1、RT20…)の定量比較を行ったところ、ほとんどのリードスルー転写物の蓄積がNMD変異体で上昇していた。つまり、リードスルー転写物がNMDの分解標的となることが分かった。
また、同定した遺伝子座271カ所のうち158カ所で、第一遺伝子と第二遺伝子の間で起こる遺伝子間スプライシングを経たリードスルー転写物が検出されました。そこで、変異により遺伝子間スプライシングが起こらない改変型のリードスルー転写物を一過的に発現させる実験を行いました。すると、第一遺伝子下流の転写終結を経た転写物が蓄積し、リードスルー転写物が著しく減少することが分かりました。この結果は、遺伝子間スプライシングが第一遺伝子の転写終結に必要な因子(ポリアデニル化シグナル[5]やポリアデニル配列付加サイト[5])を除くことで、転写のリードスルーが促進されることを示しています(図2)。
本研究では結論として、遺伝子間スプライシングによって第一遺伝子下流での転写終結が阻害され、リードスルー転写物が生成されるが、そのリードスルー転写物はNMDによって選択的に分解されることが明らかになりました(図2)。
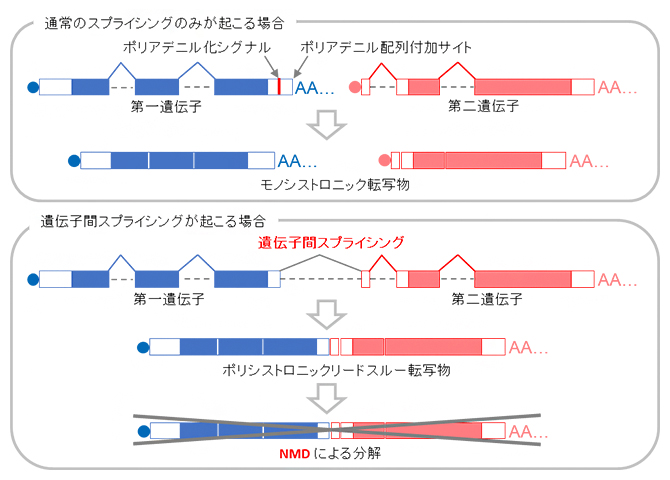
図2 遺伝子間スプライシングによるポリシストロニックリードスルー転写物の制御
(上)通常、転写終結に必要なポリアデニル化シグナルやポリアデニル配列付加サイトの存在により、各遺伝子から一つのタンパク質をコードするモノシストロニックな転写物が生成される。
(下)隣接する遺伝子間で遺伝子間スプライシングが起こると、ポリアデニル化シグナルやポリアデニル配列付加サイトが取り除かれてしまい、複数の遺伝子にまたがるポリシストロニックなリードスルー転写物が生成される。それらのリードスルー転写物はRNA品質管理機構であるNMDによって選択的に分解される。
今後の期待
植物は、不要なRNAや副産物が蓄積すると形態異常が生じたり、生命を維持できなくなったりします。そのような事態を回避するために、不要なRNAを選択的に分解するRNA品質管理機構が働きます。今回、その一つであるNMDがポリシストロニックなリードスルー転写物を分解標的とすることを明らかにしました。今後、リードスルー転写物以外のRNA品質管理機構の標的分子を同定していくことが、植物の遺伝子発現制御とそれに裏打ちされた恒常性維持の理解につながると期待できます。
遺伝子間スプライシングはリードスルー転写を促進しますが、ケースによっては第一遺伝子と第二遺伝子の読み枠がつながり、2種類のタンパク質が結合したキメラタンパク質が生成されることが予想されます。従って、この研究は今後、リードスルー転写が生み出すキメラタンパク質の同定とそれらの機能解析へ広がっていくものと期待できます。
本研究成果は、国際連合が2016年に定めた17項目の「持続可能な開発目標(SDGs)[7]」のうち「2. 飢餓をゼロに」と「12.つくる責任つかう責任」に大きく貢献するものです。
補足説明
1.遺伝子間スプライシング
スプライシングは、未成熟な転写物から不要な配列を取り除いてつなぎ合わせることで、タンパク質への正しい読み枠を持つ転写物を作る仕組み。遺伝子間スプライシングは隣接した二つの遺伝子から転写されたリードスルー転写物において、第一遺伝子領域と第二遺伝子領域の間で起こる。
2.ロングリードシークエンサー
イルミナ社に代表されるショートリードシークエンサーは、150bp長ほどのDNA断片を読むのに対して、パックバイオ社やナノポア社のロングリードシークエンサーは一般的な転写物の全長を読むことができる。
3.ナンセンスコドン介在的mRNA分解(NMD)
真核生物のRNA品質管理機構の一つ。未成熟終止コドンを持つ転写物や非常に長い3’非翻訳領域を持つ転写物を選択的に分解する。NMDが働かなくなると不要なRNAが蓄積され、形態異常や致死性の原因となる。NMDはNonsense-mediated mRNA decayの略。
4.終止コドン
タンパク質の中のアミノ酸の並び方は、そのタンパク質の遺伝子(DNA)の中の塩基配列に対応している。三つの塩基がひとまとまりになって一つのアミノ酸に対応しており、この3塩基の並びをコドンと呼ぶ。64通りのコドンが存在するが、そのうちの61種類のコドンがアミノ酸を意味している。例えばTTTはフェニルアラニンである。残り三つのコドン(TAA、TGA、TAG)はタンパク質合成終了を意味する「終止コドン」であり、遺伝子の最後に現れる。
5.転写終結、ポリアデニル化シグナル、ポリアデニル配列付加サイト
転写は、3’非翻訳領域に存在するポリアデニル化シグナルのすぐ下流のポリアデニル配列付加サイトで新生RNAは切断されることで終結する(転写終結)。その後、切断された3’末端にポリアデニル配列(ポリA鎖)が付加される。ポリA鎖は多数のアデノシン一リン酸(AMP)から構成されており、RNAをアデニンで伸長することに相当する。
6.ノックダウン
遺伝子の機能を大きく減衰させるが、完全には欠失させない状態にすること。
7.持続可能な開発目標(SDGs)
持続可能な開発目標(SDGs)とは、2015年9月の国連サミットで採択された「持続可能な開発のための2030アジェンダ」にて記載された2016年から2030年までの国際目標である。持続可能な世界を実現するための17のゴールから構成され、地球上の誰ひとりとして取り残さないことを誓っている。SDGsは発展途上国のみならず、先進国自身が取り組むユニバーサル(普遍的)なものであり、日本としても積極的に取り組んでいる。(外務省のホームページから一部改変して転載)
研究チーム
理化学研究所 環境資源科学研究センター 合成ゲノミクス研究グループ
客員研究員 栗原 志夫(クリハラ・ユキオ)
(東京大学大学院 総合文化研究科 助教)
客員研究員 蒔田 由布子(マキタ・ユウコ)
(前橋工科大学 工学部 生命情報学科 教授)
テクニカルスタッフⅠ 栗山 朋子(クリヤマ・トモコ)
研究パートタイマーⅡ 河内 正治(カワウチ・マサハル)
研修生(研究当時)蔭山 杏実(カゲヤマ・アミ)
グループディレクター 松井 南(マツイ・ミナミ)
研究支援
本研究は、RIKEN Pioneering Projects「Biology of Intracellular Environments(研究分担者:松井南)」の支援を受けて実施し、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「植物の成長段階における転写・翻訳開始点に依存したmRNAの運命決定の解明(研究代表者:栗原志夫)」による助成を受けて行われました。
原論文情報
Kurihara, Y.*, Makita, Y.*, Kawauchi, M., Kageyama, A., Kuriyama, T., Matsui, M. *These authors are equal contributions., “Intergenic splicing-stimulated transcriptional readthrough is suppressed by nonsense-mediated mRNA decay in Arabidopsis.”, Communications Biology, 10.1038/s42003-022-04348-y
発表者
理化学研究所
環境資源科学研究センター 合成ゲノミクス研究グループ
客員研究員 栗原 志夫(クリハラ・ユキオ)
客員研究員 蒔田 由布子(マキタ・ユウコ)
グループディレクター 松井 南(マツイ・ミナミ)
報道担当
理化学研究所 広報室 報道担当