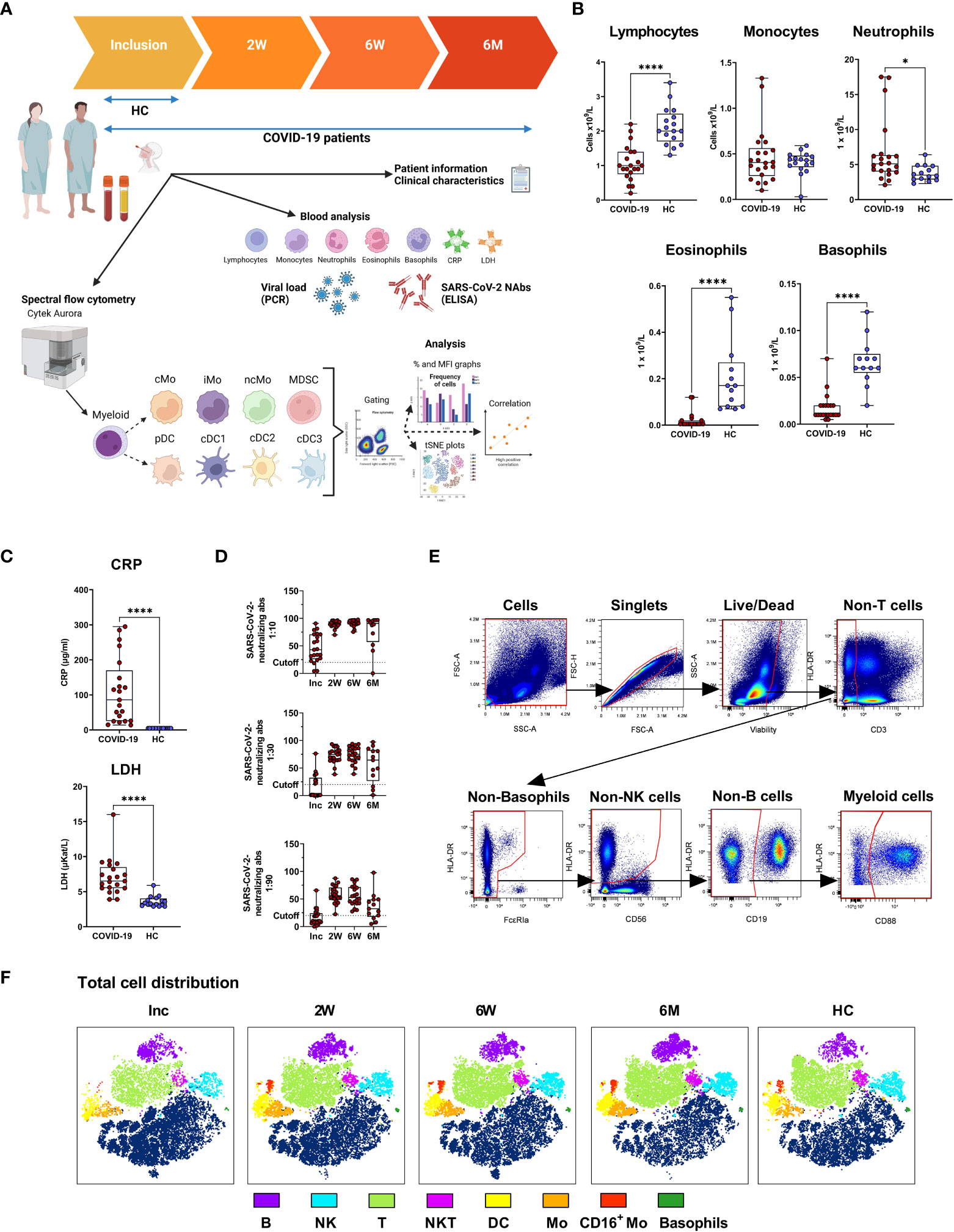
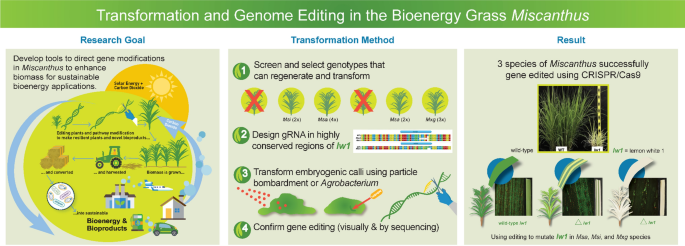
2023-01-19 先進バイオエネルギー・バイオ製品イノベーションセンター(CABBI)
◆この成果は、生産性は高いが遺伝的に複雑なこの草が持つ、バイオ燃料、再生可能なバイオ製品、炭素隔離の源としての大きな可能性を引き出す努力を加速させるものです。この研究はBiotechnology for Biofuels and Bioproducts誌に掲載された。ススキはバイオ燃料とバイオ製品の原料となる可能性があります。
◆これまで、ススキの遺伝子改良は、特定の部位を狙ったり、既存の遺伝子に手を加えたりするのではなく、外部遺伝子をゲノムのランダムな位置に導入することで植物を形質転換することに重点が置かれてきました。
◆CABBIチームは、CRISPR/Cas9を用いた遺伝子編集法を開発し、ススキの植物体内にある既存の遺伝子を選択的に標的として、その機能をノックアウトしたり、新しい遺伝子を正確な位置に導入したりすることを可能にした。このターゲティング能力は、この重要なバイオマス作物の遺伝子改良のための新しい道を示すものである。
◆今回の研究では、バイオエネルギー用に商業栽培されている生産性の高いススキ(Miscanthus x giganteus)と、その親であるサッカリフロラス(M. sacchariflorus)とシネンシス(M. sinensis)の3種のススキで遺伝子編集を実証している。これらの植物は古代のモロコシのようなDNAと複数の染色体が重複している古倍数体であるため、遺伝子を編集するためのガイドRNAの設計には、遺伝子のすべてのコピーを標的として、冗長性を考慮し、完全に「ノックアウト」することが必要だったのである。
<関連情報>
- https://cabbi.bio/cabbi-team-achieves-first-precision-gene-editing-in-miscanthus/
- https://biotechnologyforbiofuels.biomedcentral.com/articles/10.1186/s13068-022-02241-8
バイオエネルギーイネ科ススキの形質転換と遺伝子編集 Transformation and gene editing in the bioenergy grass Miscanthus
Anthony Trieu,Mohammad B. Belaffif,Pradeepa Hirannaiah,Shilpa Manjunatha,Rebekah Wood,Yokshitha Bathula,Rebecca L. Billingsley,Anjali Arpan,Erik J. Sacks,Thomas E. Clemente,Stephen P. Moose,Nancy A. Reichert & Kankshita Swaminathan
Biotechnology for Biofuels and Bioproducts Published:28 December 2022
DOI:https://doi.org/10.1186/s13068-022-02241-8
Abstract
Background
Miscanthus, a C4 member of Poaceae, is a promising perennial crop for bioenergy, renewable bioproducts, and carbon sequestration. Species of interest include nothospecies M. x giganteus and its parental species M. sacchariflorus and M. sinensis. Use of biotechnology-based procedures to genetically improve Miscanthus, to date, have only included plant transformation procedures for introduction of exogenous genes into the host genome at random, non-targeted sites.
Results
We developed gene editing procedures for Miscanthus using CRISPR/Cas9 that enabled the mutation of a specific (targeted) endogenous gene to knock out its function. Classified as paleo-allopolyploids (duplicated ancient Sorghum-like DNA plus chromosome fusion event), design of guide RNAs (gRNAs) for Miscanthus needed to target both homeologs and their alleles to account for functional redundancy. Prior research in Zea mays demonstrated that editing the lemon white1 (lw1) gene, involved in chlorophyll and carotenoid biosynthesis, via CRISPR/Cas9 yielded pale green/yellow, striped or white leaf phenotypes making lw1 a promising target for visual confirmation of editing in other species. Using sequence information from both Miscanthus and sorghum, orthologs of maize lw1 were identified; a multi-step screening approach was used to select three gRNAs that could target homeologs of lw1. Embryogenic calli of M. sacchariflorus, M. sinensis and M. x giganteus were transformed via particle bombardment (biolistics) or Agrobacterium tumefaciens introducing the Cas9 gene and three gRNAs to edit lw1. Leaves on edited Miscanthus plants displayed the same phenotypes noted in maize. Sanger sequencing confirmed editing; deletions in lw1 ranged from 1 to 26 bp in length, and one deletion (433 bp) encompassed two target sites. Confocal microscopy verified lack of autofluorescence (chlorophyll) in edited leaves/sectors.
Conclusions
We developed procedures for gene editing via CRISPR/Cas9 in Miscanthus and, to the best of our knowledge, are the first to do so. This included five genotypes representing three Miscanthus species. Designed gRNAs targeted all copies of lw1 (homeologous copies and their alleles); results also confirmed lw1 made a good editing target in species other than Z. mays. The ability to target specific loci to enable endogenous gene editing presents a new avenue for genetic improvement of this important biomass crop.