2023-01-20 信州大学,筑波大学,基礎生物学研究所,京都大学
【研究成果のポイント】
- 近年,川や池の水を汲むだけで,そこに生息する生物相を網羅的に究明する環境DNAからのメタゲノム(群集)解析は,世界的にも大きな脚光を浴び,需要が高まっている。
- 魚類や鳥類,哺乳類,甲殻類などでは汎用性の高い遺伝子マーカー(PCRプライマー)が開発されており,環境DNA解析などに多用されている。
- 地球上で最も種多様性の高い生物群である昆虫では,網羅的な群集解析の需要こそ大きいものの,種多様性が高い分だけ遺伝的多様性も高いことから,昆虫全般に適用可能な汎用性の高い遺伝子マーカー開発は困難視され,他の動物群に比べて遅れをとってきた。しかし,今回発表した論文のなかで公開した遺伝子マーカーは,論文内の試行においても高い汎用性が示されており,水生昆虫だけでなく昆虫類における群集解析が急激に加速することが期待される。
- 当該論文の投稿時には,プレプリント形式で新規開発したマーカー情報を公開しており,論文公表前から先行して国内外の環境DNA解析にも使用され,マーカーの汎用性についても好評を得ている。環境DNA解析の業務を請負う企業のなかには,既に当該論文の日本語解説をHPに掲載しているところもある。

図1 DNAバーコーディング・メタバーコーディングのイメージ
【研究成果の具体的ポイント】
- ① 地球上で最も種多様性の高い昆虫における新規DNAバーコーディング領域の提案・汎用PCRプライマーを設計した。
- ② 主要分類群をカバーする昆虫を対象に,新規開発プライマーでのPCR増幅を試行し,汎用性を実証した。
- 従来,昆虫類のDNAバーコーディングに用いられているミトコンドリアDNA(mtDNA) COI領域での解析における問題点を指摘し,その課題を改善する新規DNAバーコーディング領域をmtDNA内の16S rRNA領域と12S rRNA領域内に提案し,新規DNAバーコーディング領域を増幅するPCRプライマーを設計した。
- 昆虫の主要分類群をカバーする,無翅昆虫類,有翅昆虫類(旧翅類,多新翅類,準新翅類,完全変態類)の合計13目43科68種を対象に,設計したプライマーを用いたPCRを実施し,これらの(陸生昆虫も含めた)幅広い昆虫類に適用できることを実証した。
- 従来のDNAバーコーディングに用いられてきたmtDNA COI領域(658塩基)は長い配列であり,種内多型が多いことなどから,環境中に漂っているような分解(断片化)が進んだDNAを解析するような環境DNA研究への応用には不向きであった。しかし,この研究で新規に開発したPCRプライマーは200塩基ほどの短いDNA(短鎖DNA)断片を増幅するもので,なおかつ種識別が可能であることが特長である。
- さらに,これらの短鎖DNAを含み,より長鎖断片を解析するためのPCRプライマーも併せて設計しているため,分子系統解析などを実施しながら,自ずと各種のDNAデータベースの充実化を促進するような仕掛けにもなっている。
【背景と概要】
地球上には数多くの生物が生息しているが,その中で昆虫類は最大の種数となる約100万種が記載されている。一方で,数多くの未記載種(学名が付けられていない種)も生息しており,地球上の種多様性を把握することは重要な課題となっている。そのような背景下,絶滅に瀕している生物種も多く,環境保全や希少種の保全などが急務の課題となっている。効果的な保全対策を講じるためには,対象種や生息地の生物群集を把握する必要があるが,多大な労力と費用がかかる上に,採集した標本試料から種を識別・同定するためには,高い専門知識が必要である。
こうしたなかで,近年,注目されているのが「DNAバーコーディング」である(図1)。DNAバーコーディングは,「標準化」された遺伝子領域DNA断片を使用してデータベースの登録配列を参照することで,迅速,正確,そして自動的に種を同定するような手法である。スーパーマーケットの製品コードである黒い縞模様(バーコード)をスキャナーで読み取る方法と似ていることから名付けられたもので,高い専門知識がなくても種同定が可能になるだけでなく,卵や幼虫,不完全な標本(体の一部分など)からも同定することができる。また,未記載種や隠蔽種(外見上,一つの種として扱われてきたものの,生物学的には種分化した独立種であることが判明するような関係にある複数種)の発見にも繋がり,分類学的にも強力な補助ツールとなり得る。さらに,葉の食痕に残されたDNAから葉を摂食した動物種を特定したり,脱皮殻や排泄物,環境中からのDNA解析であれ希少種に対する非侵襲的な遺伝子解析をも可能とするなど,非侵襲的な遺伝的多様性の評価をも可能とする。
昆虫類における,従来の一般的なDNAバーコーディングでは,mtDNAのCOI領域が対象とされてきた。しかし,この遺伝子領域をDNAバーコーディング領域として用いる場合には,いくつかの問題点が挙げられる。第一に,ミトコンドリア遺伝子COI領域は,種内多型が多すぎるため,種内変異と種間変異の区別が困難である。また,環境DNA解析などにおける短鎖DNAをCOI領域内に設計するには,昆虫全般に適用できるような汎用性の高いPCRプライマーを設計する必要があるが,そのための保存領域を探索するのが困難である。実際に,今回の論文においてもその困難さを示すことができた。
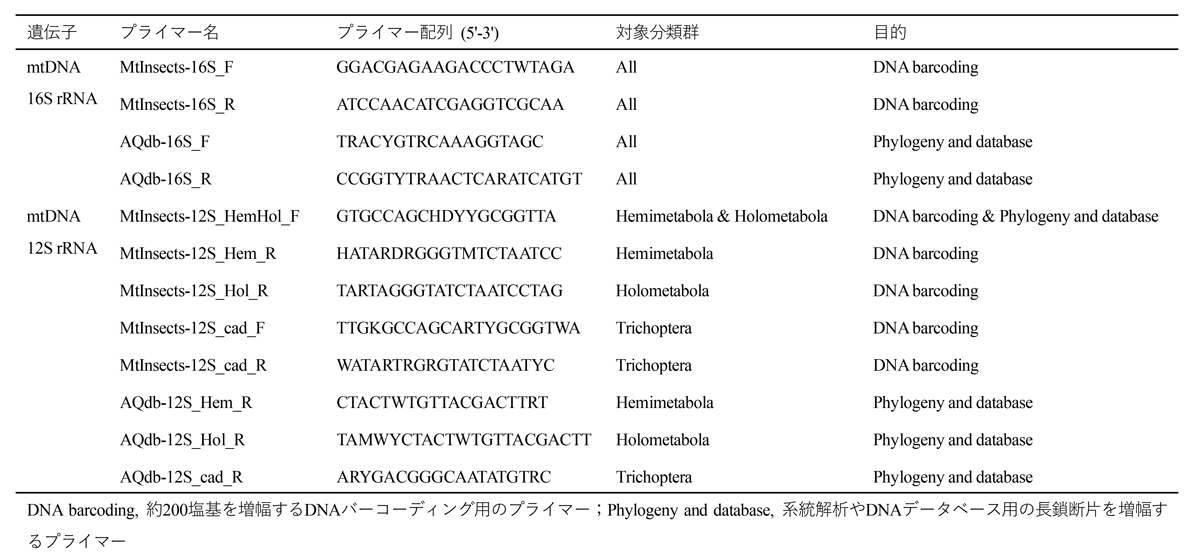
表1. 本研究で開発したプライマー情報
DNAバーコーディング,特に環境DNAを利用した群集解析における昆虫以外の先行研究では,mtDNAのリボソームRNA(rRNA)領域内に設計されたPCRプライマーが用いられてきた。mtDNA 内の16S rRNAや12S rRNAはCOI 領域よりも種内多型が少なく,塩基配列が高く保存された領域が存在し,mtDNA COI領域よりも非特異的にPCR増幅される可能性は低い。また,16S rRNAや12S rRNAは,種間変異の検出精度の観点でCOI領域にも匹敵することが報告されている。
DNA バーコーディング領域としての理想的な特性は,種間変異が大きく全ての種を区別できる一方で、種内変異は最小限であること,そして相同な特定領域をすべての対象種(この場合は全ての昆虫グループ)においてPCR増幅することができることがあげられる。本研究では,これらの条件を満たすことに加えて,環境DNA解析にも応用できるよう,200塩基程度の短鎖断片をPCR増幅するように設計している。
さらに,DNAバーコーディングには各種の参照データが必要となるため,DNA配列情報(データベース)の充実化が不可欠であり,この作業には多大な労力と経費が必要となる。そこで,データベースの充実化を促進するため,バーコディング時に参照するための短鎖断片をPCR増幅する汎用プライマーだけでなく,バーコーディング領域を含み,さらに系統解析にも適用できる長鎖断片をPCR増幅するための汎用プライマーについても併せて設計した。こうした長鎖断片をPCR増幅するプライマーを用いることで分子系統解析を目的とした研究での利用を促進しつつ,自ずとDNAバーコーディングのための参照データの蓄積にも寄与する仕掛けである。様々な目的の研究者にとっての利便性を高めることで,積極的な利用と得られた遺伝情報のデータベース登録を促進させることにより,データベースの充実につながるものと考えている。
最後に,新規に探索したDNAバーコーディング領域の遺伝情報を用いることで,近縁種間での種識別が可能であるかどうか?隠蔽種を検出できるのか?の2点についての検証を行った。水生昆虫では,近縁種間でのニッチ分化が生じ,それぞれがやや異なる微生息環境(マイクロハビタット)に適応しているような事例が知られている。ヒラタカゲロウ科 Heptageniidae のカゲロウ類は,近縁種間でニッチ分化を示す典型的なグループであるため,この研究で新しく開発されたプライマーのテストにおいて好適である。分子マーカーを用いたこれまでの研究では,広範囲や多様な環境に生息する種の系統解析の結果,未記載種や隠蔽種を発見した事例が報告されている。そこで,比較的広い範囲の河川流域に生息しており,かつ隠蔽種の存在も示唆されるキイロヒラタカゲロウ Epeorus aesculusを対象に,新規開発したDNAバーコーディング・プライマーセットの検出能力と感度の評価・検証を試みた。
【結果・考察】
研究グループは ,新規DNAバーコーディング領域をミトコンドリア遺伝子16S rRNA領域に提案し,昆虫の全分類群に汎用できるPCRプライマー「MtInsects-16S」を設計した。また,このDNAバーコーディング領域を含み,より長い断片を増幅する「AQdb-16S」を設計した。ミトコンドリア遺伝子12S rRNAに関しては,全ての分離群に汎用できるプライマーを設計することは困難であったため,不完全変態類,トビケラ目を除く完全変態類,そしてトビケラ目の3つの分類群に分けて,それぞれ設計した(表1, 図2)。
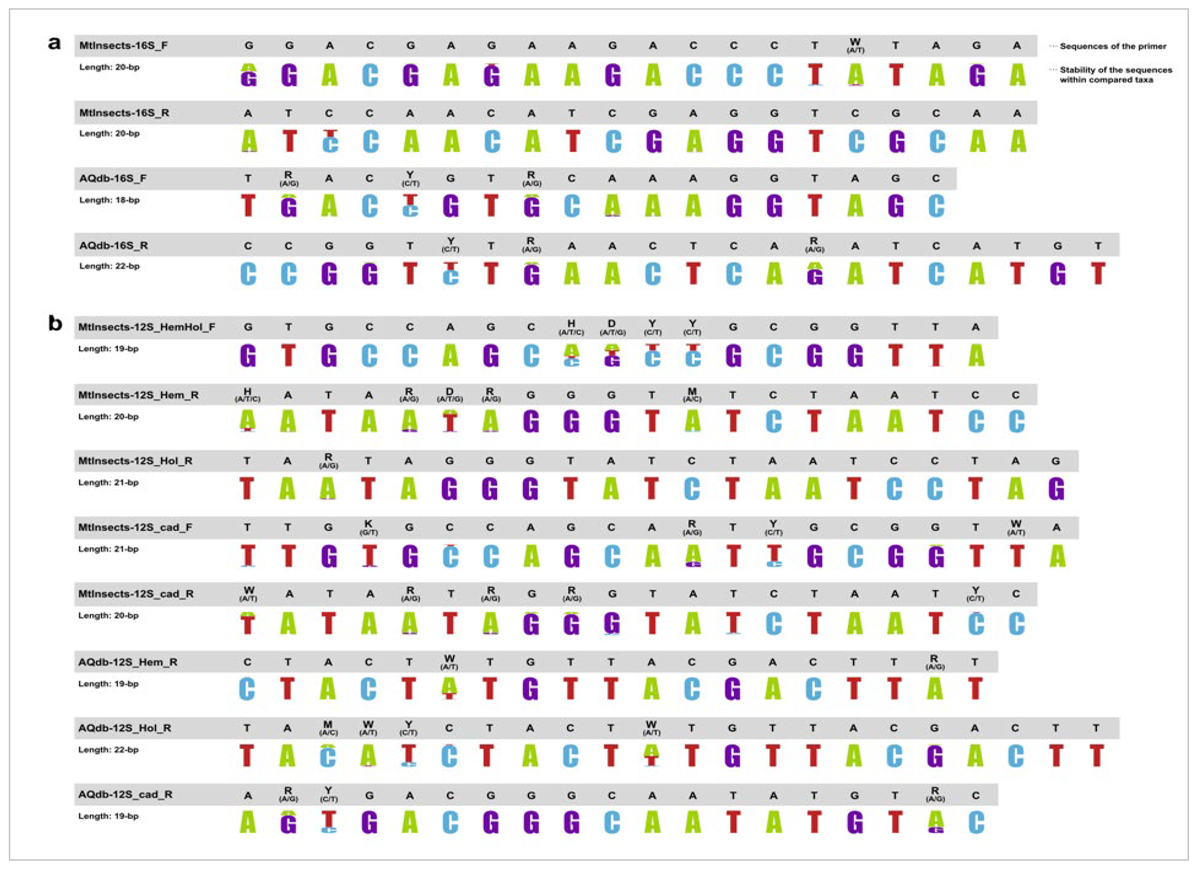
図2 本研究で開発したプライマーマー情報(a: 16S rRNA 領域, b: 12S rRNA 領域)
汎用性の確認
設計したプライマーの汎用性を確認するため,多様な昆虫群を対象にPCRを実施した結果,mtDNA 16S rRNA領域でも12S rRNA領域でも広くPCRでの増幅確認に成功した(図3)。これらの分類群には,無翅昆虫類(シミ類)から有翅昆虫類の旧翅類(カゲロウ類,トンボ類),新翅類(カワゲラ類,カメムシ類,バッタ類,カマキリ類など),そして完全変態類(トビケラ類,チョウ類,コウチュウ類,ハエ類など)が含まれている。すなわち,昆虫類の主要な系統を網羅した分類群において汎用性が高いことを確認した。
新規DNAバーコーディング領域の多型
本研究で開発した16S rRNA領域(短鎖断片)と12S rRNA領域(短鎖断片)のプライマーセットで増幅された塩基配列による種識別の成否については,従来のバーコーディング領域であるmtDNA COI領域を用いて,既に姉妹種や近縁種の解析を実施していた種群を対象に確認した。
対象種は限られるが,昆虫の主要系統の全てにおいてPCR増幅することができ,かつ種を識別することができることを確認した。対象種は限られるが,昆虫の主要系統の全てにおいてPCR増幅することができ,かつ種を識別することができることを確認した。水生昆虫の DNA バーコーディング用のプ ライマーの設計を試みたいくつかの先行研究でも,本研究と同様に mtDNA 16S rRNA 領域内にプライマーを設計しているが, COI領域のプライマーセットよりも増幅効率が良好ではないことが報告されている。そのようななかで,本研究で設計したPCRプライマーは,幅広い分類群において増幅可能であることを確認した(図3)。
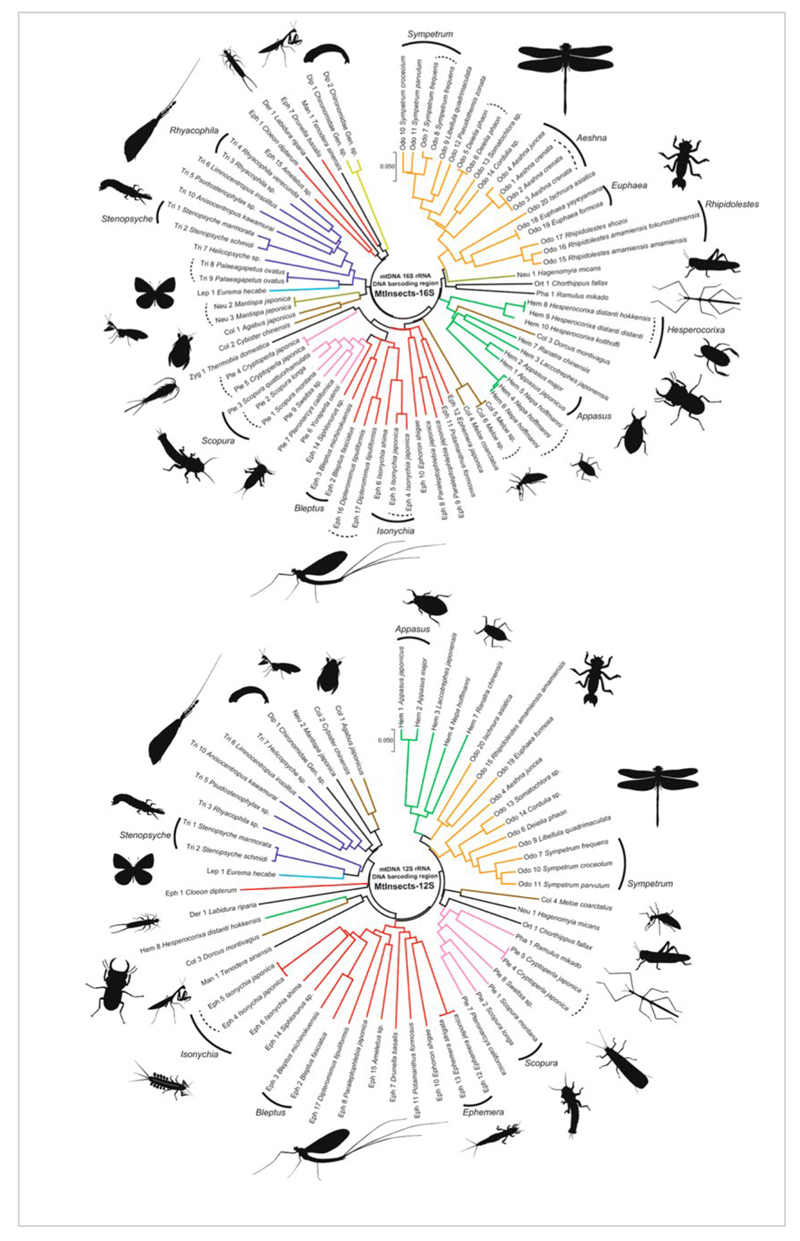 図3 本研究で開発したプライマー情報: 16S rRNA 領域, 12S rRNA 領域.mtDNA 16S rRNA領域のDNAバーコーディング領域に関しては14目43科68種で(上段),ミトコンドリア遺伝子12S rRNA領域のDNAバーコーディング領域に関しては13目42科66種で(下段)PCRの成功を確認した
図3 本研究で開発したプライマー情報: 16S rRNA 領域, 12S rRNA 領域.mtDNA 16S rRNA領域のDNAバーコーディング領域に関しては14目43科68種で(上段),ミトコンドリア遺伝子12S rRNA領域のDNAバーコーディング領域に関しては13目42科66種で(下段)PCRの成功を確認した
隠蔽種の検出
隠蔽種が含まれている可能性が示唆されるキイロヒラタカゲロウについて,長野県内の複数河川を対象に採集し,新規開発したPCRプライマーでの遺伝子解析を実施した。その結果,キイロヒラタカゲロウの新種記載におけるタイプ産地(北アルプス・黒部五郎岳)から採集したサンプル(トポタイプ)とは大きく遺伝的に分化した系統が検出された。つまり,従来,一種として扱ってきたキイロヒラタカゲロウ種内に,別種レベルに相当するほど大きく遺伝的に分化した系統が検出された。これは,本研究で新規開発した汎用プライマーが,こうした隠蔽種も検出し得る程に高い感度であることも明らかとなった。
【波及効果・今後の予定】
本研究で,広汎な昆虫類に適用可能であるDNAバーコーディング用のプライマーセットが開発されたことで,特定の環境下での昆虫相調査が大きく進展することとなる。生物多様性を評価するための長期的なモニタリングや,広範囲における同質的な手法による調査の簡便化が可能となる。これまで見落としていた,あるいは形態では識別が困難である未記載種や隠蔽種の新たな発見も期待される。加えて,直接的に人間が踏み入って調査を実施することが困難であるような地下水内の昆虫相の把握なども,採水さえ可能であれば実現可能となる。また,本研究で開発した汎用プラーマーは陸生昆虫類にも適用できることから,土壌や空気中の環境DNA解析などへの展開も期待される。
さらに本研究では,バーコーディング領域を探索し,PCRプライマーを設計しただけでなく,バーコーディング領域の参照データを効率よく蓄積していくための仕組みづくりも視野に入れている。分子系統解析など,多様な用途での利用が,結果的には参照データベースの充実化に向けて大きく寄与することが可能である。今後,これらのデータベースの充実化が進むほど,環境DNA解析の需要も加速的に高まると予想される。そして環境DNAからの群集解析は,生物の種多様性や遺伝的多様性を評価するための強力な手法となるものと考えている。
【用語解説】
PCR ポリメラーゼ連鎖反応(Polymerase Chain Reaction)といい,対象とする遺伝子配列を増幅する手法。わずかなDNA量のサンプルであっても,特定の配列だけを様々な解析に使用できるように増幅させる。新型コロナウイルスにおいても同じメカニズムを用いて,ウイルスのDNAが含まれていた際に,PCRによって特定の配列が増幅するかを調べることができる。
PCRプライマー PCRにより特定のDNA配列を増幅する際に,増幅する遺伝子配列の両端に結合し,DNAの増幅の開始点として機能する20塩基程度の短い遺伝子断片。DNAに結合した2つのプライマーが結合した箇所から,DNAが伸長することで,2つのプライマーの内側の配列が増幅されていく。新型コロナウイルスのPCR検査においては,ウイルス特異的なプライマーを設計することで,体内にウイルスが存在している時のみPCRによる特定遺伝子の増幅を確認する。
隠蔽種 実態としては複数種が含まれているにも関わらず(複数種の間で生殖的隔離が成立しているにも関わらず),形態学的な区別がつかないために同一種として扱われてきた種のことをいう。
メタゲノム解析 ある環境中に生息する生物群集を,環境中から検出されるゲノム(遺伝子情報)を網羅的に解析することで把握しようとする解析。
【論文タイトルと著者等】
タイトル:Development of novel PCR primer sets for DNA barcoding of aquatic insects, and the discovery of some cryptic species
著者:Masaki TAKENAKA, Koki YANO, Tomoya SUZIKI, Koji TOJO
掲載誌:Limnology
掲載日:2023年1月3日
URL:https://link.springer.com/article/10.1007/s10201-022-00710-5#Sec11
DOI:10.1007/s10201-022-00710-5
Masaki TAKENAKA 竹中 將起(信州大学,筑波大学) 筆頭著者・責任著者
Koki YANO 谷野 宏樹(自然科学研究機構 基礎生物学研究所)
Tomoya SUZUKI 鈴木 智也(京都大学)
Koji TOJO 東城 幸治(信州大学)責任著者
【参考情報】
本研究で開発された昆虫類のDNAバーコーディングにおける汎用性マーカーを用いたDNAメタバーコード解析などの成果は、2023年1月21日(土)に信州大学松本キャンパスで開催される公開シンポジウム「水生昆虫における環境DNAのいま」において講演予定である(オンライン配信のあるハブリッド形式のシンポジウム)。
https://www.shinshu-u.ac.jp/faculty/science/event/general/dna.html
【問い合わせ先】
〈研究内容に関する問い合わせ先〉
信州大学理学部理学科生物学コース
特任助教 竹中將起
教授 東城幸治
〈報道に関する問い合わせ先〉
国立大学法人信州大学総務部総務課広報室
国立大学法人筑波大学広報局
大学共同利用機関法人自然科学研究機構基礎生物学研究所広報室
国立大学法人京都大学総務部広報課国際広報室