ゲノムの基本構造単位の発見
2019-01-18 理化学研究所,科学技術振興機構
理化学研究所(理研) 生命機能科学研究センター 細胞システム制御学研究ユニットの大野(城村) 雅恵 研究員と谷口 雄一 ユニットリーダーらの研究チーム※1)は、ゲノム注1)DNA(以下ゲノム)の全領域にわたる3次元構造を、その最小構成単位である「ヌクレオソーム注2)」レベルの分解能で決定することに成功しました。
本研究成果は、細胞内の遺伝子の制御状態を分子レベルで理解し、人為的に制御するための基盤になるものであり、遺伝子制御に関わる新しいドラッグデザインや、iPS細胞注3)や培養臓器の効率的な作製など、医学・生物学の幅広い研究に貢献することが期待できます。
近年、生体内における遺伝子制御が適切に行われているかを診断するために、ゲノム全領域の3次元構造を調べる技術の開発が全世界的に進められています。しかし、従来の方法では、それぞれの遺伝子がどのような構造的特徴を持つのか、詳細に調べられるだけの分解能はありませんでした。
今回、研究チームは、単一ヌクレオソームレベルの分解能で、ゲノムの3次元構造を決定する手法を開発しました。酵母ゲノムの解析を行った結果、ゲノムに結合する各種タンパク質因子や化学修飾に応じたヌクレオソームの配置構造の変化を捉えることに成功しました。さらに、4つの隣り合ったヌクレオソーム同士が単位構造を形成しており、開構造と閉構造の2種類を切り替えることで、ゲノムの反応性が制御されることが分かりました。
本研究は、米国の科学雑誌「Cell」(2019年1月24日号)の掲載に先立ち、オンライン版(1月17日付け:日本時間1月18日)に掲載されます。また、本研究成果を基に制作されたイラストが、同号の表紙を飾ります。
本研究は、科学技術振興機構(JST) 戦略的創造研究推進事業 さきがけ「統合1細胞解析のための革新的技術基盤(研究総括:浜地 格)」における「1細胞内多階層オミックス動態の連関性(研究者:谷口 雄一)」の支援を受けて行われました。
<背景>
細胞内のゲノムDNA(以下ゲノム)には、生命を構成するために必要なさまざまなタンパク質のコード情報(遺伝子)が書き込まれています。このゲノムは、ヒストンと呼ばれるタンパク質に1周半ずつ巻き付き、それらが多数つながる形で、細胞内に格納されています。この巻き付いた構造は「ヌクレオソーム」と呼ばれており、ゲノム構造の最小単位となっています。
生体内では、ゲノム上のさまざまな遺伝子のスイッチのオンとオフが切り替わることで、生理状態の調節が行われます。ゲノムは、さまざまな構造状態を取ることで、遺伝子発現のスイッチを調節し、細胞の機能(分化・恒常性の維持など)を制御していると考えられています。例えば、密な凝集構造を取る領域では遺伝子の発現が抑えられることや、異なるゲノム領域が集合した領域では複数の遺伝子が同時に制御されることなどが知られています。
こうした理由から、近年、細胞がどのような制御状態にあるかを探るための手法として、ゲノムの3次元構造を解析する技術が注目を集めています。しかし、従来の方法では、それぞれの遺伝子がどのような構造的特徴を持つのか、詳細に調べられるだけの分解能はありませんでした。そこで、研究チームは、ゲノムの3次元構造の解析を世界最高の分解能で行うための技術開発を試みました。
<研究手法と成果>
研究チームは、「染色体立体配座捕捉法(Hi-C法)注4)」と呼ばれるゲノム構造解析法に注目しました。この手法では、空間的に近い距離にあるゲノム同士を連結させ、連結されたDNA領域の周辺を次世代シーケンサー注5)を用いて解読します。これによって、ゲノム上のどの部分が空間的に近接関係にあるかを、連結が起こった頻度から網羅的に解析でき、その情報からゲノムの3次元構造を導くことが可能です。ただし、従来のHi-C法では、数十~数千ヌクレオソームの分解能でしか3次元構造が得られませんでした※2)。
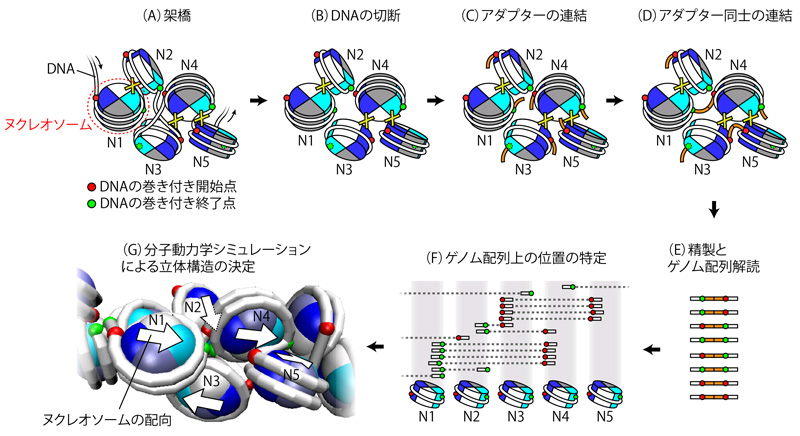
今回、研究チームは、単一ヌクレオソームの分解能で、さらにそれぞれの配向(orientation)を含めた形で3次元構造解析を行う「"Hi-C" with nucleosome "O"rientation法(Hi-CO法)」と名付けた方法を開発しました(図1)。
Hi-CO法では、まずゲノムを細やかに切断、連結した後に不純物を取り除き、得られたゲノム断片を次世代シーケンサーで網羅的に解読します。そして、得られた各ヌクレオソーム間連結頻度のデータから3次元構造を得るための計算的手法として、従来の数学的な手法ではなく、分子動力学シミュレーション注6)を新しく活用しました。計算機上で多数のヌクレオソームをDNAでつないだゲノムのモデルを構築し、それぞれのヌクレオソームを物理法則に従って揺らがせながら、連結頻度のデータを最も良く満たす各ヌクレオソームの配置と配向を探索することで、ヌクレオソームレベルの高分解能の3次元構造を得ることに成功しました。
さらに、スーパーコンピューターを用いて、構造計算を大規模に行うことで、全ゲノムスケールの構造を一度で得ることができました(図3)。得られた構造から、ヌクレオソームが不規則に配列しながら、約20~30ナノメートル(nm、1nmは10億分の1メートル)の太さのファイバーを形成していることが分かりました。このように、ゲノムの3次元構造を、単一ヌクレオソームから全ゲノムまでのスケールで表現したのは世界初となります。
ゲノム上では、遺伝子発現の制御のために、さまざまなタンパク質が結合し、さらにヒストンにはさまざまな化学修飾が起こることが知られています。統計的に解析を行った結果、これらの因子(エピゲノム因子注7))の種類に応じて、ヌクレオソームの配置構造が有意に変化していることが分かりました(図4)。つまり、分化や発生などの生命現象においては、遺伝子発現の制御状態の変化と連動して、ヌクレオソーム配置構造の変化が起こることが示されました。
また、不規則なヌクレオソームの3次元配置構造の中に一定の法則性があることを発見しました。ゲノム上で隣り合う4つのヌクレオソームの位置座標に注目したところ、正四面体で配置するグループ(閉構造)と、ひし形で配置するグループ(開構造)の2タイプに大別されることが分かりました(図5)。つまり、ヌクレオソームの配置構造には2種類の基本構造が存在しており、これらが切り替わることによって、ゲノム構造による細胞状態が制御されていることが分かりました。
<今後の期待>
今回の研究により、ゲノムの3次元構造を世界最高分解能であるヌクレオソームレベルで決定する方法を開発し、さらにその構造が細胞の制御状態と連動していることを示しました。
近年、ゲノムの配列解読技術が進んだことで先天的な病気を予測できるようになりましたが、今回、遺伝子制御の仕組みが詳しく分かるようになったことから、後天的な病気も鋭敏に発見されることが期待できます。また、ゲノム構造を土台とした、遺伝子制御を行うための新たな薬剤を設計できるようになると考えられます。
※1)研究チーム
理化学研究所 生命機能科学研究センター 細胞システム制御学研究ユニット
- 大野(城村) 雅恵(オオノ(ジョウムラ) マサエ) 研究員
- デイビッド・プリースト(David G. Priest) 国際特別研究員(研究当時)
- ビピン・クマ―(Vipin Kumar) 研修生(研究当時)
- 吉田 大和(ヨシダ ヤマト) 研究員(研究当時)
- 谷口 雄一(タニグチ ユウイチ) ユニットリーダー(JST さきがけ研究者)
分子機能シミュレーション研究チーム
- 安藤 格士(アンドウ タダシ) 研究員(研究当時)
※2)Duan, Z., Andronescu, M., Schutz, K., Mcllwain, S., Kim, Y.J., Lee, C., Shendure, J., Fields, S., Blau, C.A., and Noble, W.S. (2010). A three-dimensional model of the yeast genome. Nature 465, 363–367.
<参考図>
![]()
図1 本研究で開発した「Hi-CO法」の概要
(A)ヌクレオソームを構成するヒストンタンパク質(青・水色・白・灰の部分)やDNA(白のひも)の一部を架橋する(Xは架橋部分)。N1、N2、N3、N4、N5は、それぞれヌクレオソームを示す。
(B)ヒストンに巻き付いていない部分のDNAを切断する。
(C)ヒストンに巻き付いている部分のDNAの末端に、アダプターDNA(のりしろDNA、橙)を連結させる。
(D)空間的に近接したアダプターDNA同士を連結させる。
(E)連結したアダプターDNAの周辺を切り出して、精製し、次世代シーケンサーを用いて、それぞれのDNA配列を解読する。
(F)ゲノム上の各ヌクレオソームが連結された場所を特定する。
(G)連結情報に基づいて、各ヌクレオソームのDNAの巻き付き開始点(赤)と終了点(緑)の位置を求めることで、ヌクレオソームの配向(白矢印)を含めた立体構造を構築する。
![]()
図2 分子動力学シミュレーションによる3次元構造の導出
次世代シーケンサーにより得られたデータ(左)から3次元構造を導出するため、分子動力学シミュレーション(右)を用いた。まず、ゲノム上の各ヌクレオソーム間の連結頻度のデータを基に、各ヌクレオソームのDNA巻き付き開始点と終了点の間に引力・斥力を設定した。そして、分子動力学シミュレーションにおいて、最初は高い温度を設定して各ヌクレオソームを散らばらせた後、温度を徐々に下げていき、最適なヌクレオソームの位置と配向を見つけた。
![]()
図3 導出された出芽酵母ゲノムの3次元構造
DNAは白いひもで、ヌクレオソーム内のヒストンは青・水・灰・白色の4つのビーズの塊で表現した。ヌクレオソーム・1遺伝子レベルの図から、各ヌクレオソームが不規則に配列していることが、また1遺伝子・遺伝子群レベルの図からは、各遺伝子がひと固まりになっていることが分かる。
![]()
図4 エピゲノム因子によるヌクレオソームの配置構造の変化
それぞれのエピゲノム因子が存在するゲノム領域での、平均的なヌクレオソーム間距離(nm)を下図の棒グラフ、ヌクレオソームの配向性を上図のドット、開構造(ひし形)の取りやすさを下図の赤いドットで表した。
![]()
図5 ゲノム内におけるヌクレオソームの基本配置構造
導出した3次元ゲノム構造の中に高頻度で存在した、2種類のヌクレオソームの配置構造を示した。一方は正四面体状であり、もう一方はひし形状である。研究チームは、タンパク質の折り畳みの中に発見されたαへリックス構造、βシート構造にちなんで、それぞれαテトラへドロン構造、βロンバス構造と命名した。
<用語解説>
- 注1)ゲノム
- 生命の遺伝情報を全て含んだデオキシリボヌクレオチド(塩基と糖が結合したもの)の重合体(DNA)。アデニン、チミン、グアニン、シトシンの4種類の塩基により構成されている。さまざまな遺伝子をコードした領域が並んでいる。
- 注2)ヌクレオソーム
- 細胞内におけるゲノムDNAの最小構造単位。ヒストンと呼ばれるタンパク質に、約150~200塩基対のDNAがおよそ1周半巻き付くことで形成される。
- 注3)iPS細胞
- 脊椎動物の初期胚が持つ、全ての種類の体細胞へ分化する能力を多能性という。多能性を持ち、試験管内で培養して無限に増やすことができる細胞を多能性幹細胞という。iPS細胞(人工多能性幹細胞)は、皮膚や血液などから採取した細胞に少数の遺伝子などを導入して作製された多能性幹細胞である。
- 注4)染色体立体配座捕捉法(Hi-C法)
- ゲノムの3次元構造の解析法の1つで、2009年に開発された。次世代シーケンサーを用いて空間的に近接したゲノム領域を網羅的に検出することで、ゲノムの3次元構造の解析を行える。Hi-Cはhigh-throughput chromosome conformation captureの略。
- 注5)次世代シーケンサー
- 数百万から数億にわたる数のDNA断片の配列を並列して解読する技術。さまざまな生物種のゲノムを解読したり、RNA発現量を解析したりするのに用いられる。今日では生物学のみならず、医療・診断の分野にも幅広く普及しつつある。
- 注6)分子動力学シミュレーション
- 計算機の中でモデルの原子間に働く力を計算し、ニュートンの運動方程式を繰り返し解くことで、分子の動きを追跡する方法。
- 注7)エピゲノム因子
- DNAの配列を変えることなく、遺伝子の発現に影響を与える因子の総称。ヒストンの化学修飾や、DNAへの細胞内タンパク質の結合などが挙げられる。
<論文情報>
タイトル:“Sub-nucleosomal genome structure reveals distinct nucleosome folding motifs”
著者名:M. Ohno, T. Ando, D. G. Priest, V. Kumar, Y. Yoshida, Y. Taniguchi
DOI:10.1016/j.cell.2018.12.014
<お問い合わせ先>
<研究に関すること>
大野(城村)雅恵(オオノ(ジョウムラ) マサエ)
理化学研究所 生命機能科学研究センター 細胞システム制御学研究ユニット 研究員
谷口 雄一(タニグチ ユウイチ)
理化学研究所 生命機能科学研究センター 細胞システム制御学研究ユニット ユニットリーダー
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部
<報道担当>
理化学研究所 広報室 報道担当
科学技術振興機構 広報課