2020-11-12 京都大学iPS細胞研究所(CiRA)
ポイント
- 難治性疾患に対する新たな治療薬の開発が期待されているが、数百万個の化合物スクリーニングを実施するには多くの時間と費用がかかる。
- 化合物スクリーニングにおいてヒット化合物注1)を予測する機械学習注2)である熱拡散方程式(HDE)注3)モデルを開発し、当該研究に応用した。
- 筋萎縮性側索硬化症(ALS)注4)患者さんのiPS細胞を用いてHDEモデルを用いた200万個の化合物スクリーニングを実装し、ヒット化合物を予測抽出した。
- さらにALS患者さん由来のiPS細胞パネルを用いて、有効性が強く多くのALS患者さん由来iPS細胞に有効な化合物を同定した。
- HDEモデルは、次世代のAI創薬ツールとして期待される。
1. 要旨
日高 中(武田薬品工業)、今村恵子(京都大学CiRA増殖分化機構研究部門特定拠点講師、T-CiRA井上プロジェクト)、日置剛司(武田薬品工業、元T-CiRA井上プロジェクト)、高木輝文(武田薬品工業)、伏見真(武田薬品工業)、井上治久(京都大学CiRA増殖分化機構研究部門教授、T-CiRA井上プロジェクト、理化学研究所革新知能統合研究センター(AIP)iPS細胞連携医学的リスク回避チーム(上田修功チームリーダー)客員主管研究員、理化学研究所バイオリソース研究センターiPS創薬基盤開発チームチームリーダー)、河原吉伸(理化学研究所AIP構造的学習チームチームリーダー、九州大学 マス・フォア・インダストリ研究所 教授)および儀我美一(東京大学大学院数理科学研究科 教授)らの研究グループは、化合物スクリーニングにおけるヒット化合物を予測する機械学習のアルゴリズムである熱拡散方程式(HDE)モデルを開発し、当該研究に応用しました。HDEモデルは、熱が拡散していく様子を計算式に表して化合物の有効性の高さをスコア化することでヒット化合物を予測する人工知能のひとつで、偏微分方程式論という数学解析からの知見に基づいています。一般公開されている化合物スクリーニングのデータを用いて、HDEが高い予測精度を示すことを明らかにしました。さらに、ALS患者さんから作製したiPS細胞を用いて運動神経の細胞死を抑制する化合物を探索するための5万個の化合物スクリーニングを実施し、その結果から、HDEモデルを用いて約200万個の化合物の有効性を予測しました。HDEで有効性が高いと予測抽出された化合物について30株のALS患者さんの iPS細胞から運動神経細胞を作製し、このALS iPSパネルを用いて効果を評価しました。結果として、多くのALS患者さんの細胞に強い効果を示す化合物の同定に成功しました。これらの化合物は、これまでに知られていなかった化合物の特徴や骨格(ケモタイプ)を有しており、ALSに対する全く新規の薬剤開発のシーズ(種)として、利用されることになりました。本研究で開発されたHDEモデルは、今後の人工知能を用いたAI創薬に貢献することが期待されます。
この研究成果は2020年11月12日午前1時 (日本時間)に米国科学誌「Patterns」でオンライン公開されました。
2. 研究の背景
これまで、難病治療のために多くの化合物が開発されてきましたが、まだ治療法のない病気が多くあり、早期の治療薬開発が望まれています。化合物スクリーニング、特にヒトiPS細胞を用いた表現型スクリーニング注5)は、新規候補薬や疾患経路を発見するための有用なツールです。しかし、数100万個もの化合物を評価するには非常に多くの時間と費用がかかるため、大きな課題となっています。この課題を解決するために機械学習の創薬への利用が進められています。しかしながら、表現型スクリーニングの固有の複雑さのために、従来の機械学習での予測にはいくつかの課題がありました。例えば、化合物スクリーニングのデータは、ヒット数と非ヒット数の比率が不均衡(一般的には数%以下)で、かつ活性と物性値群が非線形な対応関係になっており、従来の方法では正確な予測が困難でした。さらに、入力するデータセットの中で特定されなかった新しいケモタイプの予測は困難でした。これらの課題を克服するために、新たな機械学習のアルゴリズムであるHDEモデルを、数学解析の知見に基づいて開発しました。
3. 研究結果
1) 化合物スクリーニングにおいてヒット化合物を予測するHDEモデルの構築
まず、研究グループは、熱拡散方程式を用いて、化合物スクリーニングでヒット化合物を予測するモデルを構築しました。このモデルは、熱が拡散していく様子を計算式で表し、化合物の効果をスコア化してヒット化合物を予測します(図1)。
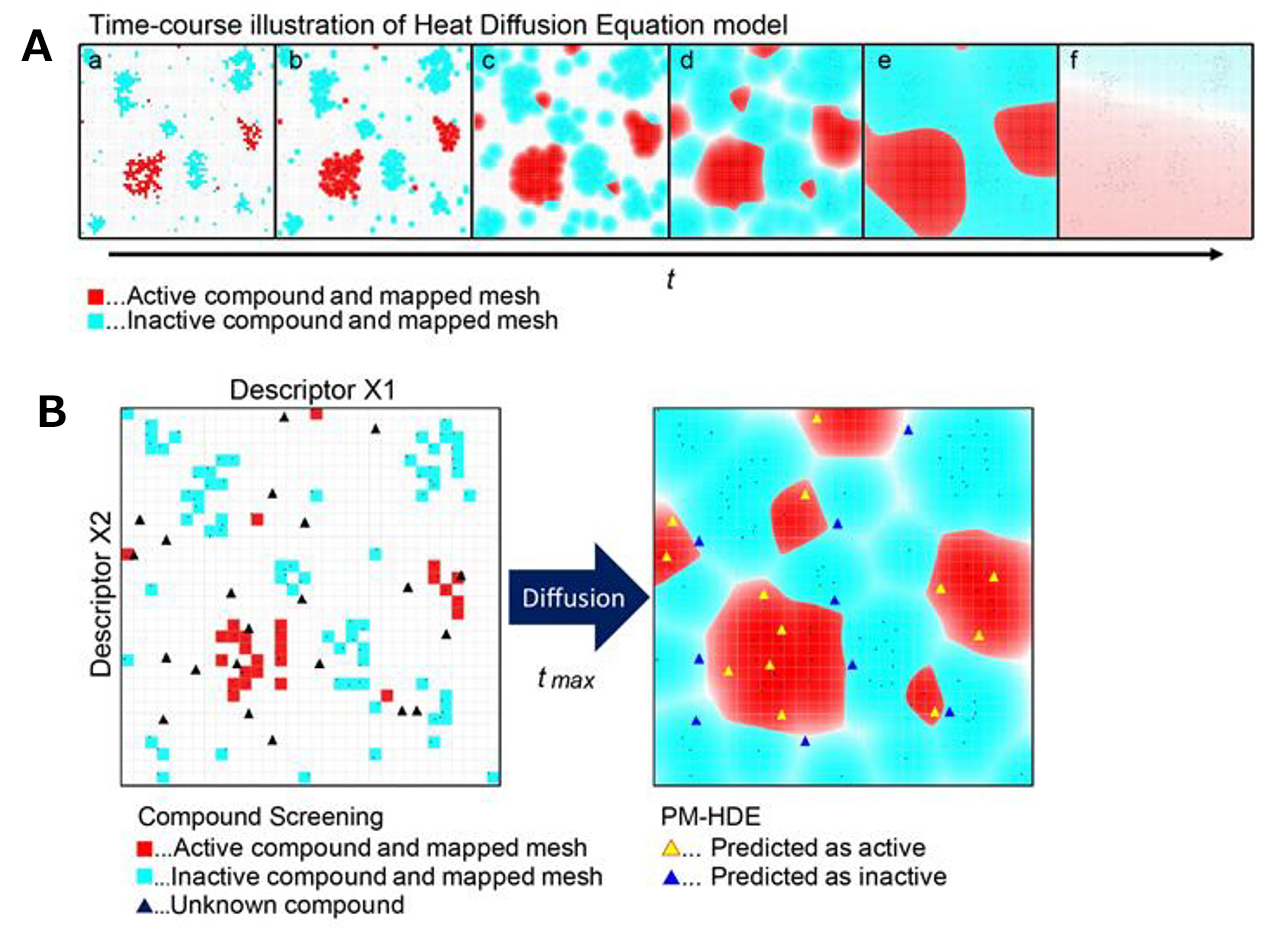
図1. HDEモデル
熱拡散の様子と予測イメージ。縦横軸は記述子(HDEモデルの説明変数)。
赤色は化合物の活性を示し、青色は非活性を示す。
A: 時間経過により、化合物の活性状態が拡散されていく様子。
B: 時間経過により化合物の活性状態が強いとされる部分をスコア化して予測する(右側黄色部分)。
2) 公開データベースを用いたHDEモデルの予測精度の評価
次に、一般に公開されている化合物スクリーニングのデータベース(PubChem)を用いて、HDEモデルの予測精度を評価しました。HDEモデルは高い予測精度を示しました(図2)。
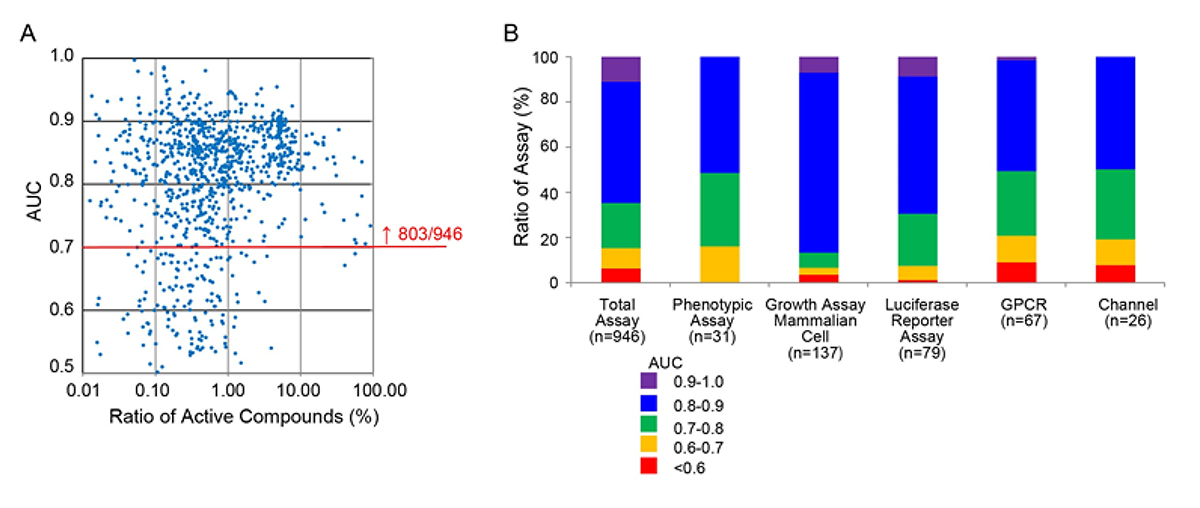
図2. データベースを用いたHDEモデルの精度評価
熱拡散の様子と予測イメージ。縦横軸は記述子(HDEモデルの説明変数)。
赤色は化合物の活性を示し、青色は非活性を示す。
A: 946セットのデータベースを評価したところ、803セットでAUC注6)0.7以上を示した。
B: 様々な評価法を用いたデータセットでHDEモデルは高い精度を示した。
3)ALS患者さんのiPS細胞を用いた化合物スクリーニング
ALS患者さんのiPS細胞から作製した運動神経細胞の細胞死を抑制する化合物のスクリーニングを実施しました(図3)。約50,000個の化合物スクリーニングを実施し、その結果をHDEモデルの学習に用いました。
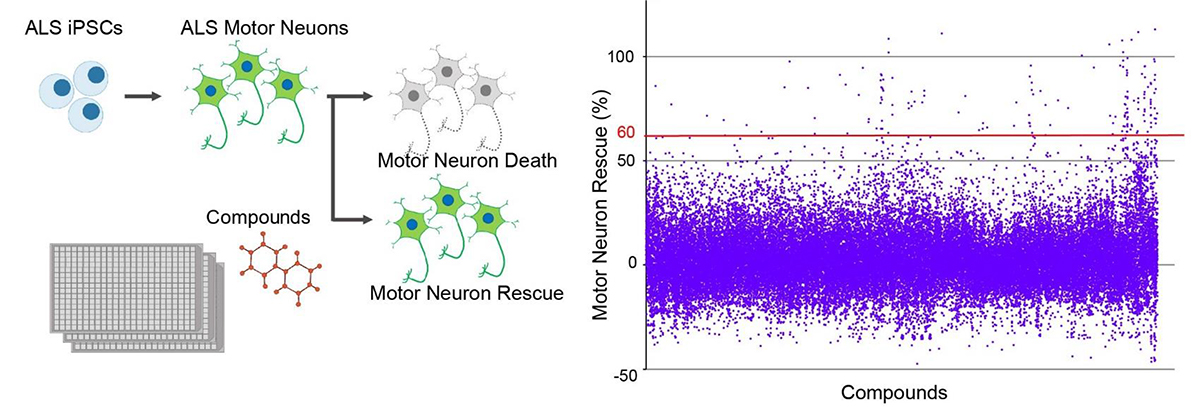
図3. ALS患者さんのiPS細胞を用いた化合物スクリーニング
ALS患者さんのiPS細胞から運動神経細胞を作製し、約50,000個の化合物スクリーニングを行い運動神経細胞の細胞死抑制率を観察した。右グラフの細胞死抑制率60%を閾値として、ヒット化合物を求めた。
4) ALS患者iPS細胞の化合物スクリーニングにおけるHDEモデルの実装
ALS患者さんのiPS細胞の化合物スクリーニングの結果を用いてHDEモデルを学習させ、200万個の化合物のシミュレーションを行いました。HDEモデルは、200万個の化合物の中から5,875個の化合物を抽出しました(図4)。
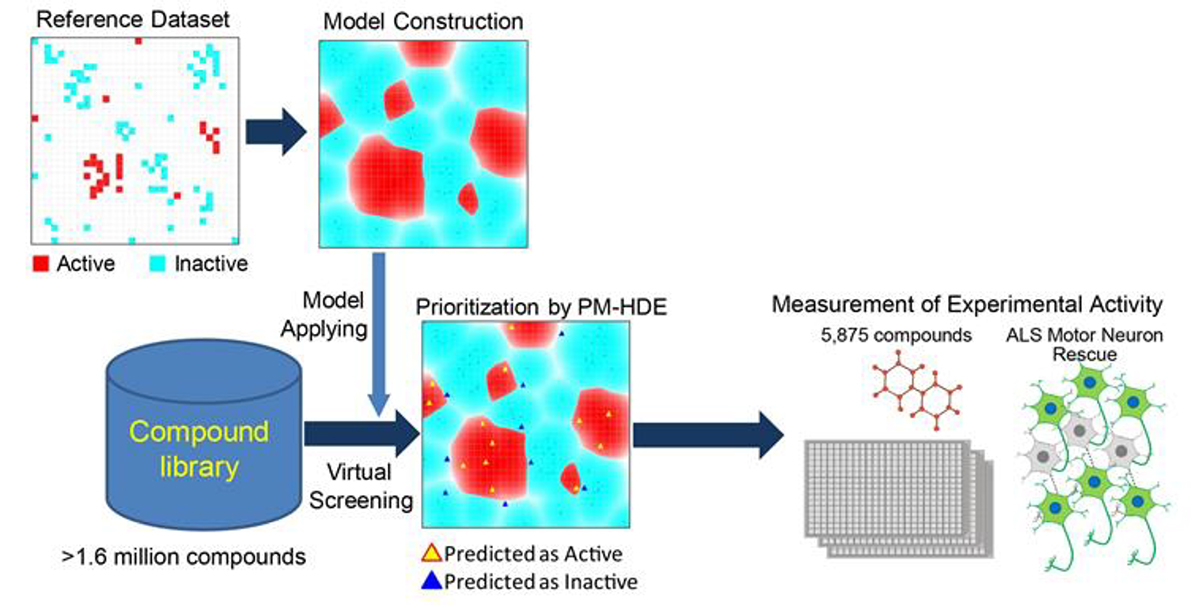
図4. ALS化合物スクリーニングにおけるHDEモデルの実装
HDEモデルを用いてALS運動神経細胞に有効な化合物の予測を実施した。
5)ALS iPS細胞パネルを用いた化合物有効性評価
最後に、約30株のALS患者さんの iPS細胞から運動神経細胞を作製し、このALS iPSパネルを用いてHDEモデルで抽出した化合物を評価しました(図5)。その結果、既にALSの治療薬として認可されているリルゾールやエダラボンよりも細胞死抑制効果が強く、多くのALS患者運動神経細胞に効果を示す化合物が同定されました。これらの化合物は、これまでに知られていなかった化合物の特徴(ケモタイプ)を保有しており、ALSに対する全く新規の薬剤開発のシーズとして、利用されることになりました。
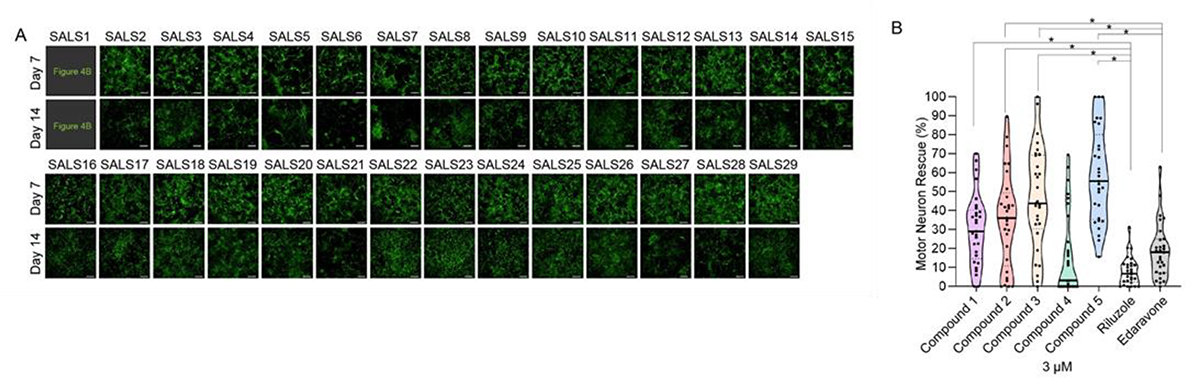
図5. ALS iPS細胞パネルを用いた化合物の評価
A: ALS患者iPS細胞から作製した運動神経細胞。
B: 抽出された化合物の有効性評価。縦軸は運動神経細胞の細胞死抑制率を示す。
リルゾールやエダラボン(右側)よりも細胞死抑制効果が高い複数の化合物が同定された。
4. まとめ
化合物スクリーニングにおいて有効な化合物を予測する機械学習のアルゴリズムとして、数学解析の知見に基づいてHDEモデルを開発し、当該研究に活用しました。これは、熱拡散方程式の創薬へのはじめての応用で、HDEモデルはこれまでの機械学習方法よりもヒット化合物予測の柔軟性を有している利点があると考えられます。
HDEモデルを用いて、ALS治療薬のための化合物スクリーニングを実装し、有望なヒット化合物の同定に成功しました。これらの化合物は、ALSに対する全く新規の薬剤開発のシーズとして、利用されることになりました。本研究成果は、今後の人工知能を用いたAI創薬に貢献することが期待されます。
5. 論文名と著者
- 論文名
Prediction of Compound Bioactivities Using Heat-Diffusion Equation - ジャーナル名
Patterns - 著者
Tadashi Hidaka1)10), Keiko Imamura2)3)4)5)10), Takeshi Hioki1)3)10), Terufumi Takagi1), Yoshikazu Giga6)7), Mi-Ho Giga6)7), Yoshiteru Nishimura8), Yoshinobu Kawahara8)9), Satoru Hayashi1)3), Takeshi Niki2)3), Makoto Fushimi1)*, Haruhisa Inoue2)3)4)5)1)*
*Correspondence: Haruhisa Inoue, Makoto Fushimi - 著者の所属機関
- Research, Takeda Pharmaceutical Company Limited, Tokyo, Japan
- Center for iPS Cell Research and Application (CiRA), Kyoto University, Kyoto, Japan
- Takeda-CiRA Joint Program (T-CiRA), Fujisawa, Japan
- iPSC-based Drug Discovery and Development Team, RIKEN BioResource Research Center (BRC), Kyoto, Japan
- Medical-risk Avoidance based on iPS Cells Team, RIKEN Center for Advanced Intelligence Project (AIP), Kyoto, Japan
- Graduate School of Mathematical Sciences, University of Tokyo, Tokyo, Japan
- Institute for Mathematics in Advanced Interdisciplinary Study, Sapporo, Japan
- Structured Learning Team, RIKEN Center for Advanced Intelligence Project (AIP), Fukuoka, Japan
- Institute of Mathematics for Industry, Kyushu University, Fukuoka, Japan
- These authors contributed equally
6. 本研究への支援
本研究は、下記機関より支援を受けて実施されました。
- AMED 再生医療実現拠点ネットワークプログラム(iPS細胞研究中核拠点)
- AMED 再生医療実現拠点ネットワークプログラム(疾患特異的iPS細胞を活用した難病研究)
- 科研費(18K18452)
7. 用語説明
注1) ヒット化合物
スクリーニングにおいて評価された化合物のうち、期待される効果を示した化合物。
注2) 機械学習(MI: Machine Intelligence)
人工知能(AI:Artificial Intelligence)の一つで、人間の学習能力のような機能をコンピュータで実現しようとする技術・手法のこと
注3) 熱拡散方程式(HDE: Heat Diffusion Equation)
熱の拡散を記述する偏微分方程式。熱拡散方程式 (HDE) モデルは、武田薬品工業株式会社が東京大学と共同でHDEを応用して開発したもの「特開2018-92575(P2018-92575A)」。
注4) 筋萎縮性側索硬化症(ALS)
運動神経細胞が選択的に変性・消失していく原因不明の疾患。筋萎縮と筋力低下が主な症状で、進行すると上肢の機能障害、歩行障害、嚥下障害、呼吸障害などを生じる。病気を起こすメカニズムは詳しく知られておらず、まだ十分な治療法ない。約5%が家族性で、そのうちの約20%にSOD1遺伝子変異を伴う。その他、TDP-43遺伝子変異やC9orf72遺伝子内のくり返し配列の伸長などの遺伝要因が知られている。
注5) 表現型スクリーニング
疾患の特異的な病態(表現型)を再現し、それを抑えるための化合物スクリーニングのこと。
注6) AUC(Area Under Curve)
機械学習の評価指標として用いられる。0から1までの値をとり、機械学習の能力が高いほど1に近づく。