ゲノム解析と遺伝子発現量の変化の統合解析法を開発
2018-06-21 理化学研究所,リエージュ大学
理化学研究所(理研)生命医科学研究センター基盤技術開発研究チームの桃沢幸秀チームリーダー、久保充明副センター長(研究当時)、リエージュ大学GIGAセンターのミシェル・ジョージズディレクター、動物ゲノミクス研究室のジュリア・ドミトリーバ博士らをはじめ、7カ国22大学・研究所からなる国際共同研究グループ※は、ゲノム解析と遺伝子発現量の変化を組み合わせた新たな解析手法を開発し、「炎症性腸疾患[1]」の発症には非常に複雑な遺伝子発現制御機構が存在することを明らかにしました。本研究成果は今後、炎症性腸疾患の発症機構や新たな診断法・治療法の開発につながると期待できます。
炎症性腸疾患は大腸や小腸などに慢性の炎症や潰瘍を起こす難病の一つで、日本においても患者数は年々増加しており、新薬開発が望まれています。これまでゲノムワイド関連解析(GWAS)[2]によって、200以上の疾患発症に関わる因子である遺伝子バリアント[3]が明らかになりました。しかし、ゲノム解析から発症機構の解明に至ったのは一部のみでした。今回、国際共同研究グループは、これまでのゲノム解析に加え、独自に構築したeQTL[4](遺伝子発現量の個人差に関与するゲノム領域)データを新たな手法で統合解析しました。その結果、約100の新たな原因遺伝子候補を同定するとともに、予想に反して近傍の複数遺伝子の発現量が同時に変化することで発症につながっている可能性が示されました。また、今回開発した統合解析手法は他の疾患にも応用できると考えられます。
本研究は、英国のオンライン科学雑誌『Nature Communications』(6月21日付け)に掲載されます。
※国際共同研究グループ
理化学研究所
生命医科学研究センター 基盤技術開発研究チーム
チームリーダー 桃沢 幸秀(ももざわ ゆきひで)
生命医科学研究センター
副センター長(研究当時) 久保 充明(くぼ みちあき)
リエージュ大学(ベルギー) GIGAセンター
ディレクター ミシェル・ジョージズ(Michel Georges)
動物ゲノミクス研究室
博士研究員 ジュリア・ドミトリーバ(Julia Dmitrieva)
背景
「炎症性腸疾患」は、大腸や小腸などに慢性の炎症や潰瘍を起こす疾患の総称で、クローン病[5]と潰瘍性大腸炎[6]に分類されます。10代~20代の若者が発症しやすい傾向にありますが完治させることは難しく、継続的な経過観察および治療が必要となる難病の一つです。欧米諸国において患者数が多いことが知られていますが、日本でも患者数は年々増加しています。発症には衛生環境や食生活などに加えて、遺伝的な要因も影響を強く及ぼしていることが知られています。
その遺伝的影響を明らかにするために、桃沢チームリーダーらの国際共同研究グループも参加する「国際炎症性腸疾患ジェネティクス・コンソーシアム[7]」が中心となり、これまでゲノムワイド関連解析(GWAS)を用いて、疾患発症に関わるゲノム領域が200以上明らかになりました注1)。それらのゲノム領域に存在すると想定される疾患発症の原因遺伝子同定は、疾患発症機構の解明や新しい治療薬の開発につながります。
これまで、低頻度の遺伝子バリアントの解析が可能なイムノチップ解析[8]注2)、低頻度の遺伝子バリアントの集積を解明するシークエンス解析注3)、高度な統計的解析手法注4)などを用いて解析が行われましたが、原因遺伝子を同定できたのは一部のゲノム領域のみでした。
注1)Uniken Venema WT et al., The genetic background of inflammatory bowel disease: from correlation to causality. J Pathol. 2017 Jan;241(2):146-158.
注2)2015年7月21日プレスリリース「炎症性腸疾患の発症に関わる38カ所のゲノム領域を発見」
注3)Momozawa Y et al., Resequencing of positional candidates identifies low frequency IL23R coding variants protecting against inflammatory bowel disease. Nat Genet. 2011 Jan;43(1):43-7.
注4)Huang H et al., Fine-mapping inflammatory bowel disease loci to single-variant resolution. Nature. 2017 Jul 13;547(7662):173-178
研究手法と成果
国際共同研究グループはまず、GWASにより明らかになった遺伝子バリアントの多くは遺伝子の翻訳領域外に存在することから、「それらの遺伝子バリアントが疾患発症に重要な細胞内で、近傍に存在する遺伝子の発現量を変化させることで発症に至る」という仮説を立てました。この仮説に基づいた解析を行うためには、ゲノム全体にわたり、遺伝子バリアントと遺伝子発現量の関係を明らかにしたeQTL(遺伝子発現量の個人差に関与するゲノム領域)データが必要ですが、そのようなeQTLデータは細胞の種類によって異なるため、炎症性腸疾患の発症に重要だと考えられる免疫細胞や腸管の上皮細胞におけるeQTLデータが不可欠だと考えられました。
そこで、リエージュ大学付属病院において、健康な約350人の血液から6種類の免疫細胞と、腸管の3カ所の上皮細胞をそれぞれ収集しました。続いて、各遺伝子の発現量を測定するとともに、ゲノム全体の遺伝子バリアントを解析し、合計23,650個の遺伝子バリアントと遺伝子発現量の関係を示したeQTLデータを構築しました。
次に、疾患発症の原因遺伝子の同定を目指し、遺伝子バリアントと疾患の関係を示すGWASデータとeQTLデータを統合解析するための新たな手法を開発しました。
まず、図1のように、遺伝子バリアントが近傍遺伝子の発現量の変化を引き起こすことが発症につながる場合は、最も疾患への影響が大きい遺伝子バリアントは遺伝子の発現量も最も大きく変え、疾患への影響が弱いバリアントになるにつれ発現量の変化も小さくなると予想されることから、GWASデータとeQTLデータの関連(P値:観察した事象が偶然に起こる確率)のパターンが類似していると考えられます。
図1左には、GWASデータとeQTLデータに共通して、同じゲノム位置に遺伝子バリアントの関連の強さを示すピークが見られます。また、図1右のように、それぞれの遺伝子バリアントの関係を発現量の増減も考慮した散布図で表わすと、強い負の相関があることが分かります。このことから、遺伝子バリアントは対象遺伝子の発現量を低下させることで、疾患発症に関与すると考えられます。
逆に、図2のように、遺伝子バリアントが近傍遺伝子の発現量変化に関わっているが、疾患発症には関与していないと考えられる場合は、異なるゲノム位置に遺伝子バリアントの関連の強さを示すピークが存在し、かつ互いに相関が認められません。
この解析手法を用いて、国際炎症性腸疾患ジェネティクス・コンソーシアムから入手したGWASデータと、200以上の疾患発症に関わるゲノム領域について統合解析を行った結果、約100個の遺伝子を原因遺伝子の有力な候補として同定しました。この中には、炎症性腸疾患の原因遺伝子としてすでに知られるATG16L1、CARD9、FUT2などや、免疫関連遺伝子のIL18R1、IL6ST、THEMISに加えて、APEH、ANKRD55、CISD1、CPEB4、DOCK7、ERAP2、GNA12、GPX1、GSDMB、ORMDL3、SKAP2、UBE2L3、ZMIZ1など、現状では疾患発症との関連が不明な遺伝子も含まれていました。今回、示した各ゲノム領域についてのGWASデータとeQTLデータの関係については、本研究のウェブサイト注5)において確認することができます。
また、各ゲノム領域からGWASデータとeQTLデータの両方に類似のパターンを示す遺伝子は一つだけと予想しましたが、図3に示すように、一つのゲノム領域から複数の遺伝子のeQTLとGWASデータが同じパターンを示しているものが25カ所も同定されました。これは、これまでの想定と異なり、複数の近傍遺伝子が同時に疾患発症に寄与するという複雑な制御機構を持つ可能性を示しています。
最後に、得られた原因遺伝子候補の中に真の原因遺伝子が実際に含まれているかを検証するために、低頻度の遺伝子バリアントの集積という概念を用いました。つまり、目的の遺伝子について疾患群と対照群のシークエンス解析を行って低頻度の遺伝子バリアントを同定し、それが疾患群または対照群の一方に偏りがあれば、それは原因遺伝子としてのさらなる証拠になるという考え方です。
約6,600人の疾患群、約5,500人の対照群について、約100個の原因遺伝子候補のうち45遺伝子の翻訳領域を、基盤技術開発研究チームで開発したターゲットシークエンス手法注6)を用いて解析しました。その結果、疾患群と対照群の間に強い偏り(P<6.9×10-4)が認められ、今回同定した遺伝子群の中に多く真の原因遺伝子が含まれていることが示されました。
注5)CEDAR のウェブサイト
注6)2016年11月11日プレスリリース「加齢黄斑変性発症に関わる新たな遺伝子型を発見」
今後の期待
今後、同定された100個の各遺伝子について炎症性腸疾患にどのようにつながるか機能や動物モデルの解析を行うことで、炎症性腸疾患の発症機構や、新たな診断法・治療法の開発につながると期待できます。また、今回開発したeQTLデータとGWASデータの統合解析手法は、他の疾患にも応用できると考えられます。
なお、この解析に使用した遺伝子バリアント(登録番号 E-MTAB-6666)および遺伝子の発現量のデータ(E-MTAB-6667)は、欧州バイオインフォマティクス研究所(EBI)で公開しています。
原論文情報
Yukihide Momozawa, Julia Dmitrieva, Emilie Théâtre, Valérie Deffontaine, Souad Rahmouni, Benoît Charloteaux, François Crins, Elisa Docampo, Mahmoud Elansary, Ann-Stephan Gori, Christelle Lecut, Rob Mariman, Myriam Mni, Cécile Oury, Ilya Altukhov, Dmitry Alexeev, Yuri Aulchenko, Leila Amininejad, G. Bouma, F. Hoentjen, M. Löwenberg, B. Oldenburg, M.J. Pierik, A.E. vander Meulen-de Jong, C.J. vander Woude, Marijn C. Visschedijk, The International IBD Genetics Consortium, Mark Lathrop, Jean-Pierre Hugot, Rinse K. Weersma, Martine De Vos, Denis Franchimont, Severine Vermeire, Michiaki Kubo, Edouard Louis & Michel Georges, “IBD risk loci are enriched in multigenic regulatory modules encompassing causative genes”, Nature Communications, 10.1038/s41467-018-04365-8
発表者
理化学研究所
生命医科学研究センター 基盤技術開発研究チーム
チームリーダー 桃沢 幸秀(ももざわ ゆきひで)
生命医科学研究センター
副センター長(研究当時) 久保 充明(くぼ みちあき)
リエージュ大学(ベルギー) GIGAセンター
ディレクター ミシェル・ジョージズ(Michel Georges)
動物ゲノミクス研究室
博士研究員 ジュリア・ドミトリーバ(Julia Dmitrieva)
報道担当
理化学研究所 広報室 報道担当
補足説明
-
- 炎症性腸疾患
- 消化管に炎症や潰瘍を起こす難病。主にクローン病と潰瘍性大腸炎に分類される。10~20歳代の若者が発症しやすく、下痢や血便、体重減少が見られる。完治が難しく生涯にわたって経過観察・治療が必要なため、新薬開発が望まれている。
-
- ゲノムワイド関連解析(GWAS)
- 疾患の関連遺伝子を見つける代表的な方法。ヒトゲノムを網羅した300~1,000万の遺伝子バリアントを対象に、患者群と対照群の間のアレル頻度の違いを見る統計学的解析手法。患者群・対照群の数が多いほど検出力が上がるため、近年はコンソーシアムを結成し、大規模検体を用いた解析が行われている。GWASはGenome-Wide Association Studyの略。
-
- 遺伝子バリアント
- ヒトのDNA配列は30億の塩基対からなるが、その配列の個人間の違いを遺伝子バリアントと呼ぶ。
-
- eQTL
- 遺伝子の発現量の個人差に関与するゲノム領域。eQTLはexpression Quantitative Trait Locusの略。
-
- クローン病
- 炎症性腸疾患の一つ。口腔から肛門までの全ての消化管に炎症や潰瘍ができることが知られているが、小腸と大腸に発症することが多い。
-
- 潰瘍性大腸炎
- クローン病と並ぶ炎症性腸疾患の一つ。大腸の粘膜にびらんや潰瘍(かいよう)を形成する疾患。
-
- 国際炎症性腸疾患ジェネティクス・コンソーシアム
- 炎症性腸疾患の遺伝的背景を解明するために結成された国際共同研究グループ。結成当初は欧米諸国が中心だったが、その後、東アジア・インド・イランなどが加わった。
-
- イムノチップ解析
- 自己免疫疾患や炎症性疾患についてさらに詳細にゲノム解析するため、GWASにおいて関連が示された遺伝子を中心に約20万の遺伝子バリアントを調べられるように開発されたチップ。頻度が低い遺伝子バリアントを多く含むため、GWASでは検出できない遺伝子バリアントについても解析が可能である。
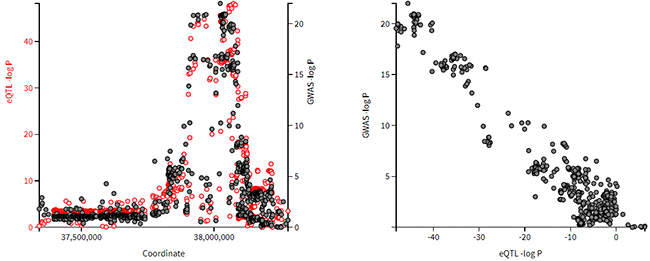
図1 遺伝子バリアントが対象遺伝子の発現量を低下させることで発症に関わっている例
左:横軸はゲノム地図上の位置、縦軸は関連の強さを示す。赤丸がeQTLデータにおける遺伝子バリアントの関連の強さ、黒丸がGWASデータにおける遺伝子バリアントの関連の強さを示している。赤丸と黒丸は共通して同じゲノム位置にピークが見られる。
右:各遺伝子バリアントにおける、eQTLとGWASの関連の強さの散布図。横軸のeQTLの関連の強さは、発現量への影響に応じて正負を付加している。この例では、強い負の相関が見られるため、遺伝子バリアントは対象遺伝子の発現量を低下させることで、発症に関与すると考えられる。
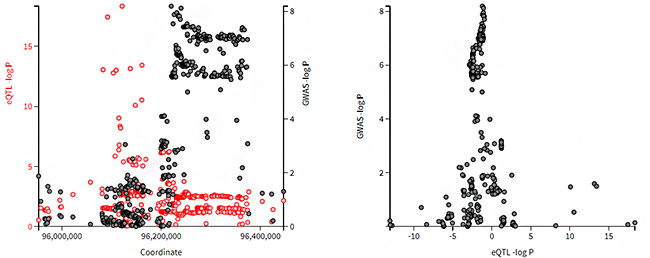
図2 遺伝子バリアントが近傍遺伝子の発現量変化に関わっているが発症に関与しない場合
左: 横軸はゲノム地図上の位置、縦軸は関連の強さを示す。赤丸がeQTLデータにおける遺伝子バリアントの関連の強さ、黒丸がGWASデータにおける遺伝子バリアントの関連の強さを示している。赤丸と黒丸は異なるゲノム位置にピークが見られる。
右: 各遺伝子バリアントにおける、eQTLとGWASの関連の強さの散布図。両者の間に相関関係は認められないことから、遺伝子発現量の変化は疾患の発症に関与しないと考えられる。
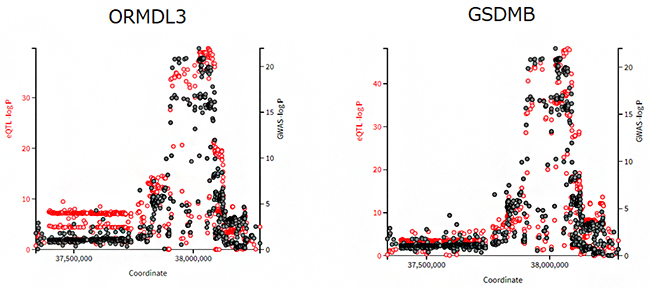
図3 遺伝子バリアントが近傍の二つの遺伝子の発現量変化に関わって発症に関与する場合
横軸はゲノム地図上の位置、縦軸は関連の強さを示す。赤丸がeQTLデータにおける遺伝子バリアントの関連の強さ、黒丸がGWASデータにおける遺伝子バリアントの関連の強さを示している。ORMDL3(左)とGSDMB(右)という遺伝子は同じゲノム領域に存在しているが、ともに赤丸と黒丸が共通して同じゲノム位置にピークが見られる。このことから、両方の遺伝子の発現量変化が疾患発症に関与していると考えられる。