2022-3-24 スウェーデン王・王立工科大学(KTH)
・KTH王立工科大学とカロリンスカ研究所の研究者らは、この病原体ハンターの移動が、飛び石のある小川で遊ぶ子供のランダムな動きに似ていることを示唆するモデルを作成した。
<関連情報>
- https://www.kth.se/en/aktuellt/nyheter/how-antibodies-navigate-pathogen-surfaces-like-a-child-at-play-1.1155230
- https://www.nature.com/articles/s43588-022-00218-z
抗体結合の確率的モデリングにより、抗原パターン上でのプログラマブルな移動を予測 Stochastic modeling of antibody binding predicts programmable migration on antigen patterns
Ian T. Hoffecker,Alan Shaw,Viktoria Sorokina,Ioanna Smyrlaki &Björn Högberg
Published: 24 March 2022 DOI:10.1038 / s43588-022-00218-z
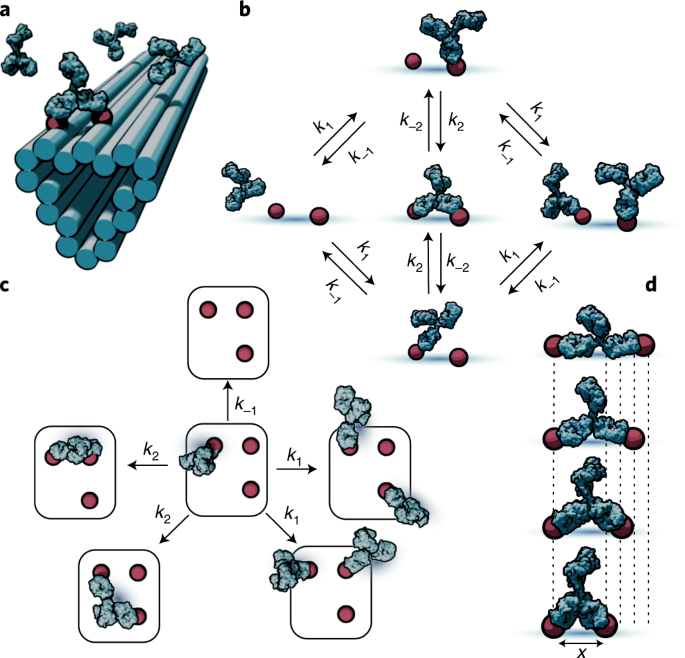
図1:多抗原基質への抗体の結合ダイナミクスをモデル化するためのスキーム。
Abstract
Viruses and bacteria commonly exhibit spatial repetition of the surface molecules that directly interface with the host immune system. However, the complex interaction of patterned surfaces with immune molecules containing multiple binding domains is poorly understood. We developed a pipeline for constructing mechanistic models of antibody interactions with patterned antigen substrates. Our framework relies on immobilized DNA origami nanostructures decorated with precisely placed antigens. The results revealed that antigen spacing is a spatial control parameter that can be tuned to influence the antibody residence time and migration speed. The model predicts that gradients in antigen spacing can drive persistent, directed antibody migration in the direction of more stable spacing. These results depict antibody–antigen interactions as a computational system where antigen geometry constrains and potentially directs the antibody movement. We propose that this form of molecular programmability could be exploited during the co-evolution of pathogens and immune systems or in the design of molecular machines.