2021-12-01 理化学研究所,福岡大学,九州大学
理化学研究所(理研)生命医科学研究センターゲノム解析応用研究チームの末次弘征客員研究員(九州大学医学部整形外科学教室)、寺尾知可史チームリーダー、骨関節疾患研究チームの池川志郎チームリーダー、福岡大学医学部整形外科学教室の山本卓明教授らの共同研究グループを中心とする特発性大腿骨頭壊死症調査研究班[1]は、日本人と韓国人合わせて約13万人からなるアジア人集団の遺伝情報を用いてゲノムワイド関連解析(GWAS)[2]を行い、「全身性エリテマトーデス(SLE)[3]患者に伴うステロイド関連大腿骨頭壊死症(S-ONFH)」の発生に関わる疾患感受性領域[4]を新たに3カ所同定しました。
本研究成果は、自己免疫疾患[5]の一つであるSLEの患者に伴うS-ONFHの病態の解明や治療法の開発につながると期待できます。
S-ONFHは、ステロイド[6]を服用している人に何らかの原因により大腿骨頭に虚血性の骨梗塞[7]が生じる疾患です。ステロイドを服用する基礎疾患としてはSLEが最も多いとされていますが、服用していても大腿骨頭壊死症が発生しない人もおり、その違いは遺伝因子によると考えられています。
今回、共同研究グループは韓国の研究グループと協力し、アジア人集団(SLE患者でS-ONFHを発生している人784人、対照群13万2603人)において大規模GWASのメタ解析[8]を行いました。この解析は単一の基礎疾患を背景としたS-ONFHの研究コホート(集団)としては世界最大です。解析の結果、SLE患者に伴うS-ONFH発生に関わる新たな三つの疾患感受性領域を同定し、そのうち二つはそれぞれ脂質代謝、血管での発現を介してS-ONFHの発生に関与することが分かりました。
本研究は、科学雑誌『Human Molecular Genetics』オンライン版(12月1日付:日本時間12月1日)に掲載される予定です。
背景
特発性大腿骨頭壊死症(Osteonecrosis of the femoral head; ONFH)は、何らかの原因により大腿骨頭に虚血性の骨梗塞が生じる疾患です。日本国内で年間2,000~3,000人が発症し、好発年齢は30~50代の働き盛りであることが分かっています注1)。発生の初期は無症状ですが、進行するに従って骨頭が圧壊し、股関節に疼痛が生じます。ひとたび疼痛が始まると、根本的な治療は手術しかないことから、病態の解明および予防法や治療薬の開発が待ち望まれています。
ONFHの発症原因は分かっていませんが、過去の疫学研究から、ステロイドの服用とアルコールの多飲が関連することが明らかになっています。そのうち、ステロイド服用に関連するONFHを「ステロイド関連大腿骨頭壊死症(Steroid-associated osteonecrosis of the femoral head; S-ONFH)」といいます。ステロイドと大腿骨頭壊死の因果関係は分かっていませんが、ステロイドを服用している人でも大腿骨頭壊死を発生する人としない人がおり、その違いは遺伝因子によると考えられています。
これまで、S-ONFHに関する多くの遺伝子研究が行われてきましたが、サンプル数の不足や、ステロイドを服用する基礎疾患の混在により、統一した見解の得られた疾患感受性遺伝子は同定されていませんでした。今回、共同研究グループは全ゲノムレベルでバイアスなく疾患感受性遺伝子を同定できるゲノムワイド関連解析(GWAS)を用いて、S-ONFHの疾患感受性遺伝子の同定を試みました。
研究手法と成果
S-ONFHはステロイド関連の大腿骨頭壊死症であるため、患者はステロイドを投与される何らかの基礎疾患を持っています。共同研究グループは、S-ONFHの疾患感受性遺伝子の同定を阻んでいるのは、S-ONFHに性質の異なるサブタイプが存在することが主な原因と考え、基礎疾患の定義が明確なS-ONFHに注目しました。基礎疾患によってS-ONFHの原因となる遺伝子多型[9]が異なる可能性も想定し、ステロイドを服用する基礎疾患では最も多いとされる、自己免疫疾患の一つである全身性エリテマトーデス(SLE)を対象としました。
SLEの患者でS-ONFHを発生している日本人636人に対して、バイオバンクジャパン[10]の9万5588人を対照群として、GWASを行った結果、ゲノムワイド有意水準(P<5×10-8)[11]を満たす領域が16カ所見つかりました(図1)。
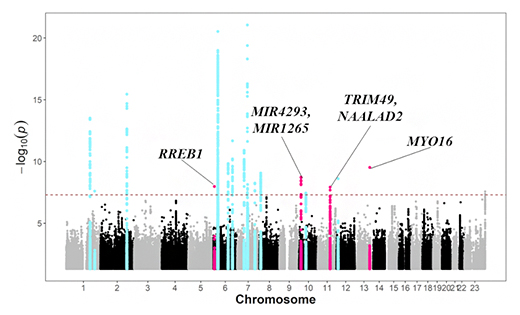
図1 日本人におけるSLE患者でS-ONFHを発生している人と対照群のGWASの結果
横軸は染色体の位置、縦軸は10を底としたP値の対数である。点線はゲノムワイド有意水準(P<5×10-8)を示す。各点は一塩基多型(SNP)を示しており、Y軸の値が高いほど、全身性エリテマトーデス(SLE)もしくはステロイド関連大腿骨頭壊死症(S-ONFH)に対する相関が強いことを意味する。赤がS-ONFHの疾患感受性領域の候補であり、青はSLEの既知の疾患感受性領域。
ただし、これら16領域がSLEとS-ONFHのどちらの疾患感受性領域かは分かりません。そこで、過去のSLEの遺伝子研究論文を当たったところ、16領域のうち12領域は既にSLEの疾患感受性領域として報告されていたため、残りの4領域がS-ONFHの疾患感受性領域の候補と考えられました。
この4領域がSLEと関連がないことを証明するために、4領域でそれぞれ最も関連が強い一塩基多型(SNP)[9]について、SLE患者でS-ONFHを発生していない日本人686人に対して、バイオバンクジャパンの9万5588人を対照群としてGWASを行いました。すると、4領域のSNPはSLEとは関連がないことが分かりました(図2)。過去最大の東アジア人研究コホート(集団)を対象とした過去の研究においてもSLEとの関連が見られなかったことから、4領域のSNPはSLEではなくS-ONFHに関連した領域であると考えられました(図2)。
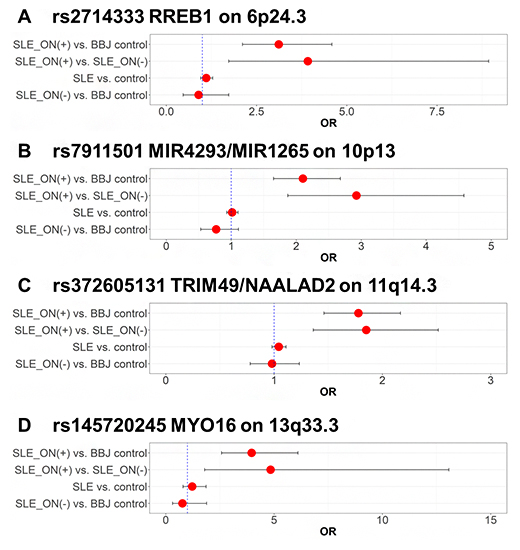
図2 4領域で最も相関が強かったSNPについて患者と対照群を入れ替えた関連解析の結果
横軸はオッズ比(Odds ratio; OR)、赤点は各々の関連解析におけるOR、エラーバーは95%信頼区間を示す。縦軸は、各々の関連解析におけるケースvs. 対照群である。SLE_ON(+)がSLE患者でS-ONFHを発生している人636人、SLE_ON(-)がSLE患者でS-ONFHを発生していない人686人、BBJ controlはバイオバンクジャパンの9万5588人。SLE vs. controlは、過去に共同研究グループが発表した、東アジア人のSLE患者1万3377人と対照群19万4993人のGWASの結果から引用したもの。A~DのSNPはSLE_ON(+)と相関しており、SLE_ON(-)や東アジア人のSLE相関解析では相関が見られなかったことから、これらのSNPはSLEではなくSLE_ON(+)と相関していることが分かる。
さらに、この4領域に関して再現性を確認するために、韓国人のSLE患者でS-ONFHを発生している148人に対して、韓国人一般集団3万7015人を対照群としたGWASを行い、さらに日本人とのメタ解析を行ったところ、3領域がゲノムワイド有意水準を満たしました。以上の結果から、この3領域がS-ONFHの疾患感受性遺伝子座[4](MIR4293、NAALAD2、MYO16)であると考えられました。
次に、同定した疾患感受性遺伝子座の機能を調べました。MIR4293はマイクロRNA(miRNA)[12]の一種です。miRNAは他の遺伝子の発現を制御することで、疾患に関連していると考えられています。MIR4293が発現を制御する遺伝子を調べたところ111個の遺伝子が見つかり、そのうちの多くは脂質代謝に関連することが分かりました。この結果、MIR4293は脂質代謝に関連する遺伝子群を制御することにより、S-ONFHに関連している可能性が示されました。また、NAALAD2は、有意水準を満たす他のSNP(rs372605131)に、脛骨(けいこつ)動脈において発現を制御されており、血管系での遺伝子発現を介してS-ONFHの発症に関与している可能性が示されました。
さらに、今回のGWASにより、S-ONFHとの関連の強さをどの程度持ったSNPが検出されるのかを検証しました。その結果、アレル頻度[13]10%のSNPにおいては、オッズ比[14](発症リスクの大きさの指標)1.87のリスクSNPが99%の確率で検出されました。アレル頻度10%以上のSNPは検出されなかったことから、ありふれたSNPにおいてはオッズ比が2を超えるようなハイリスクの疾患感受性SNP[4]は存在しないことが強く示唆されました。
また、今回のアプローチでは、SLE患者でS-ONFHを発生している人と発生していない人では、サンプル数の不足によりSNPの検出力が不十分であったため、対照群としてバイオバンクジャパンのサンプルを用いることで検体数を増やし、検出力を向上させました。さらに、SLE患者でS-ONFHを発生していない人のサンプルを用いてS-ONFHと相関がないことを示すことにより、検出された領域がSLEではなくS-ONFHと相関していることが証明されました。
今後の期待
本研究では、疾患感受性遺伝子を三つ同定しました。そのうちMIR4293は脂質代謝、NAALAD2は血管での発現を介してS-ONFHの発生に関与する可能性が示されました。今後、分子生物学的にこれらの遺伝子についてS-ONFHとの関連が明らかにされれば、病態解明や治療薬の開発につながると期待できます。
また、今回用いたGWASのデザインは、疾患内での合併症の有無を比較する関連解析において、検出力不足を解決できる非常に有用なアプローチであると考えられます。
補足説明
1.特発性大腿骨頭壊死症調査研究班
福岡大学医学部整形外科学教室の山本卓明教授らを中心とする大腿骨頭壊死症の専門医で構成された特発性大腿骨頭壊死症の研究グループ(Japanese Research Committee on Idiopathic Osteonecrosis of the Femoral Head)。
主なメンバーは以下の通り。所属は研究協力当時、敬称略。
九州大学整形外科(中島康晴、岩本幸英、本村悟朗、池村聡、山口亮介、烏山和之、園田和彦、久保祐介、宇都宮健、畑中敬之、馬場省次、河野紘一郎、末次弘征、山本典子)、福岡大学整形外科(山本卓明、坂本哲哉)、古賀病院21(坂本悠磨)、大阪大学整形外科(菅野伸彦、西井孝、坂井孝司、高尾正樹、安藤渉)、市立札幌病院(向井正也、片岡浩、近藤真)、北海道大学整形外科(岩崎倫政、高橋大介、入江徹、浅野毅)、北海道大学第2内科(渥美達也、狩野皓平)、九州医療センター(宮村知也)、千葉大学整形外科(岸田俊二、中村順一、萩原茂生)、新潟大学腎・膠原病内科(成田一衛、黒田毅)、九州大学第一内科(赤司浩一、塚本浩、新納宏昭、有信洋二郎、赤星光輝、三苫弘喜、綾野雅宏)、慶應大学整形外科(戸山芳昭、宮本健史、船山敦、藤江厚廣)、慶應義塾大学リウマチ内科(竹内勤、花岡洋成、山岡邦宏)、東京女子医科大学付属膠原病リウマチ痛風センター(桃原茂樹、山中寿、川口鎮司、猪狩勝則、細澤徹自)、埼玉医科大学総合医療センターリウマチ・膠原病内科(天野宏一)、佐賀大学医学部膠原病・リウマチ内科(多田芳史)、順天堂大学膠原病・リウマチ内科(山路健、関谷文男、松下雅和)、北海道内科リウマチ科病院(清水昌人、谷村一秀、松橋めぐみ)、昭和大学藤が丘病院整形外科(渥美敬、玉置聡、中西亮介)、名古屋大学整形外科(長谷川幸治、関泰輔)、産業医科大学第1内科(田中良哉、齋藤和義、中野和久)、京都府立医科大学整形外科(久保俊一、上島圭一郎)、帯広厚生病院(久田諒)、筑波大学整形外科(吉岡友和)、木戸病院(山﨑美穂子)、金沢大学整形外科(加畑多文、楫野良知)、斗南病院(天崎吉晴、楫野知道)、大阪市立大学整形外科(中村博亮、溝川滋一、大田陽一)、久留米大学医療センター整形外科・関節外科センター(大川孝浩、久米慎一郎)、三重大学整形外科(湏藤啓広、長谷川正裕、内藤陽平)、金沢医科大学整形外科(兼氏歩、市堰徹)、広島大学整形外科(安永裕司、山崎琢磨)、関西労災病院整形外科(大園健二)
2.ゲノムワイド関連解析(GWAS)
疾患の感受性遺伝子を見つける方法の一つ。ヒトのゲノム全体を網羅する遺伝子多型を用いて、疾患を持つ群と疾患を持たない群とで遺伝子多型の頻度に差があるかどうかを統計学的に検定する方法。検定の結果得られたP値(偶然にそのようなことが起こる確率)が低いほど相関が高いと判定できる。GWASは、Genome-Wide Association Studyの略。
3.全身性エリテマトーデス(SLE)
何らかの原因により自己抗体が産生され、皮膚・関節・脳・腎臓・肺・血管をはじめとする全身の臓器が障害され、多彩な臨床症状を呈する病気。
4.疾患感受性領域、疾患感受性遺伝子座、疾患感受性SNP
疾患の発症に関連している染色体上の領域、遺伝子座、SNPのこと。
5.自己免疫疾患
本来は、外来から侵入してくる全ての異物から生体を守るはずの免疫システムが異常を来し、誤って自分自身の細胞や組織を攻撃してしまう疾患。
6.ステロイド
副腎皮質ホルモンの一つで、免疫反応を抑えたり炎症を鎮めたりするのによく使用されている薬剤。
7.骨梗塞
骨の血流が途絶し、骨が壊死する病態。
8.メタ解析
独立して行われた複数の研究の統計解析結果を合算する統計学的手法。
9.遺伝子多型、一塩基多型(SNP)
ヒトゲノムは30億塩基対のDNAからなるが、個々人を比較するとそのうちの0.1%の塩基配列の違いがある。これを遺伝子多型という。遺伝子多型のうち一つの塩基が、他の塩基に変わるものを一塩基多型と呼ぶ。SNPは、Single Nucleotide Polymorphismの略。
10.バイオバンクジャパン
オーダーメード医療実現化プロジェクトの基盤となるDNAサンプルや血清サンプルを47疾患(延べ約20万人)から収集し、臨床情報とともに保管している世界でも有数の資源バンク。
11.ゲノムワイド有意水準(P<5×10-8)
GWASでは数十万から数百万のSNPを一度に調べるため、偶然疾患などと関連しているようにみえるSNPが多数検出される(偽陽性の問題)。そのため、「P値が5×10-8未満」という高い有意水準で関連を認めたSNPのみを真の陽性としており、このP値の基準をゲノムワイドな有意水準と呼ぶ。
12.マイクロRNA(miRNA)
20~25塩基長程の配列を持つ1本鎖RNA。他の遺伝子の発現を制御することでさまざまな疾患に関連しているといわれている。
13.アレル頻度
個々のヒトゲノムを比較すると、染色体上の場所が同一であっても、遺伝子や個々の塩基配列が異なる場合がある。これらの遺伝子や塩基配列をアレルという。例えば、ある染色体上の位置において、個人によりAA/AG/GGのどれかの塩基配列を持つ場合、集団の中でのA(アデニン)やG(グアニン)の頻度をアレル頻度という。
14.オッズ比
発症リスクの大きさの指標。基準とする集団のリスクに対して、発症するリスクが何倍になるかを表す。
共同研究グループ
理化学研究所 生命医科学研究センター
ゲノム解析応用研究チーム
客員研究員 末次 弘征(すえつぐ ひろゆき)
(九州大学 医学部 整形外科学教室)
チームリーダー 寺尾 知可史(てらお ちかし)
骨関節疾患研究チーム
チームリーダー 池川 志郎(いけがわ しろう)
福岡大学 医学部 整形外科学教室
教授 山本 卓明(やまもと たくあき)
九州大学大学院 医学研究院 整形外科学教室
教授 中島 康晴(なかしま やすはる)
准教授 本村 悟朗(もとむら ごろう)
原論文情報
Hiroyuki Suetsugu, Kwangwoo Kim, Takuaki Yamamoto, So-Young Bang, Yuma Sakamoto, Jung-Min Shin, Nobuhiko Sugano, Ji Soong Kim, Masaya Mukai, Yeon-Kyung Lee, Koichiro Ohmura, Dae Jin Park, Daisuke Takahashi, Ga-Young Ahn, Kohei Karino, Young-Chang Kwon, Tomoya Miyamura, Jihye Kim, Junichi Nakamura, Goro Motomura, Takeshi Kuroda, Hiroaki Niiro, Takeshi Miyamoto, Tsutomu Takeuchi, Katsunori Ikari, Koichi Amano, Yoshifumi Tada, Ken Yamaji, Masato Shimizu, Takashi Atsumi, Taisuke Seki, Yoshiya Tanaka, Toshikazu Kubo, Ryo Hisada, Tomokazu Yoshioka, Mihoko Yamazaki, Tamon Kabata, Tomomichi Kajino, Yoichi Ohta, Takahiro Okawa, Yohei Naito, Ayumi Kaneuji, Yuji Yasunaga, Kenji Ohzono, Kohei Tomizuka, Masaru Koido, Koichi Matsuda, Yukinori Okada, Akari Suzuki, Bong-Jo Kim, Yuta Kochi, Hye-Soon Lee, Shiro Ikegawa, Sang-Cheol Bae, Chikashi Terao, “Novel susceptibility loci for steroid-associated osteonecrosis of the femoral head in systemic lupus erythematosus”, Human Molecular Genetics, 10.1093/hmg/ddab306
発表者
理化学研究所
生命医科学研究センター ゲノム解析応用研究チーム
客員研究員 末次 弘征(すえつぐ ひろゆき)
(九州大学医学部 整形外科学教室)
チームリーダー 寺尾 知可史(てらお ちかし)
骨関節疾患研究チーム
チームリーダー 池川 志郎(いけがわ しろう)
福岡大学 医学部 整形外科学教室
教授 山本 卓明(やまもと たくあき)
報道担当
理化学研究所 広報室 報道担当
福岡大学 企画部広報課
九州大学 広報室