2023-02-22 理化学研究所
理化学研究所(理研)放射光科学研究センター 生命系放射光利用システム開発チームの中迫 雅由 客員主管研究員、佐藤 航地 研修生、大出 真央 研修生(研究当時)の研究チームは、タンパク質の水和構造[1]を予測する人工知能(AI)[2]の開発に成功しました。
本研究成果は、分子動力学計算[3]によるタンパク質の動態の解明や創薬の解析に貢献すると期待できます。
近年、クライオ電子顕微鏡[4]によって、数多くのタンパク質の立体構造が可視化できるようになってきました。しかし、タンパク質の内部に取り込まれたり、表面に吸着している水分子(水和水分子)は十分に可視化できないという弱点があります。そのため、クライオ電子顕微鏡で得られた立体構造モデルを基にして、分子認識[5]や構造変化についてさらに研究を進めるには、その内部や表面に適切な水和構造を付与する必要があります。
このような状況に対応するべく、今回研究チームはX線結晶構造解析[6]によって高い分解能で可視化された水和水分子を含む膨大な数のタンパク質立体構造について、水分子周辺の原子配置を学習し、その学習結果に基づいて任意のタンパク質の水和構造を予測できるAIを開発しました。
本研究は、科学雑誌『Scientific Reports』オンライン版(2月7日付)に掲載されました。
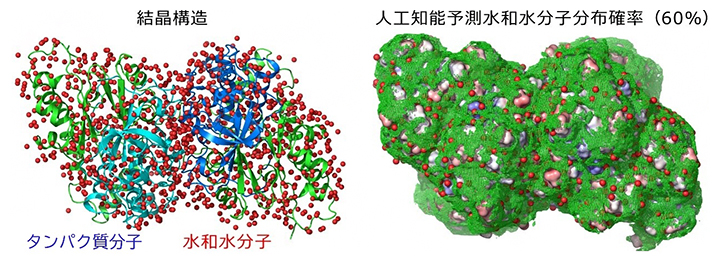
タンパク質の結晶構造解析(左)およびAIが予測した水和水分子の分布(右)
背景
生命現象の素過程を担う生体高分子は、水溶液中において折り畳まれた状態で機能しています。そのため、水溶液環境にある生体高分子の振る舞いを可視化することは、生命現象を原子レベルで読み解く上で極めて重要です。実際、タンパク質の内部に取り込まれたり、タンパク質の表面に吸着している水分子(水和水分子と呼ぶ)とタンパク質を構成する原子との相互作用が、タンパク質の機能発現を支配していることが多くの研究から示唆されてきました。
近年、クライオ電子顕微鏡が溶液中での生体高分子の立体構造を近原子分解能で可視化する強力な方法として成熟してきました。この観察方法では、X線結晶構造解析のような結晶化のための特殊な溶媒環境が不要であり、単に、急速凍結によって非晶質氷に包埋された生体高分子を観察し、単粒子解析法[7]によって構造解析を行うため、溶液中での構造を可視化できると考えられています。
しかし、低温下でのX線結晶構造解析と比べると、解像度が若干低く、また電子が酸素原子によって散乱されにくいことから、水分子の位置の検出が困難だという弱点があります。そのため、得られる分子構造モデルには、水分子がほとんど含まれていません。従って、その構造モデルを出発点として、分子動力学計算のような計算科学解析や、どのような分子が生体高分子の機能を抑制できるのかといった創薬解析を迅速に進めようとすると、簡便に水分子の位置を知る必要があります。
研究チームは長年タンパク質の水和構造を研究しており、2009~2010年にタンパク質の立体構造データベースから、水に溶けやすいタンパク質構成アミノ酸について水和構造形態を明らかにするとともに、それに基づいたタンパク質の水和構造予測法を開発しました。しかしその方法では、タンパク質表面に露出した水に溶けにくいアミノ酸で構成された部分の水和構造は予測できませんでした。
一方で近年、人工知能(AI)が急速に発達し、科学技術分野においてもその能力が注目されるようになっています。AIは、脳の神経ネットワークにおける情報制御を計算機上で模倣し、既に得られている情報を学習することによって最適化されます。その結果、与えられた新しい情報に対して、最適な対応を提案することができます。このことから、既にX線結晶構造解析によって蓄積されてきた水和水分子を含むタンパク質立体構造モデルから、水和水分子の周りにどのようなタンパク質原子が分布しているのかを学習するAIを構築できれば、与えられた水和構造が欠落したタンパク質構造モデルに対して、水和水分子の分布確率[8]やもっともらしい位置を予測することが期待できます。
研究手法と成果
AI分野では多くの場合、2次元画像データを対象とするのに対して、タンパク質の水和構造は3次元データとなります。今回、水和構造予測のためのAIを構築するにあたって、研究チームは3次元畳み込みニューラルネットワーク[9]によってAIの中心部分を構成し、水和水分子周辺のタンパク質原子分布を学習させることにしました。構築されたAIの学習データとして、0.16~0.18ナノメートル(nm、1nmは10億分の1メートル)の高い分解能で得られた2,145個のX線結晶構造解析モデルから5,310,762個のデータを作成し、そのうちの70%を学習用に、残り30%を学習成果を検証する試験に用いました。学習によって最適化されたAIは、タンパク質の内部や表面に存在する水和水分子がどのような場所に何%の確率で存在しうるかを示す3次元予測分布確率と、その分布確率の局所極大位置を水和水分子サイトの候補として回答します。
図1に、検証試験で予測されたタンパク質の水和水分子の3次元分布確率を示します。分布確率のレベル(%)を下げてみると、それは、水分子を近似する直径0.3nmの球をタンパク質表面に転がして得られる水分子アクセス表面と一致しました。また、レベルを上げていくと、水和水分子サイトの候補が現れ、それらはX線結晶構造解析で得られた水和水分子サイトとよく一致しました。
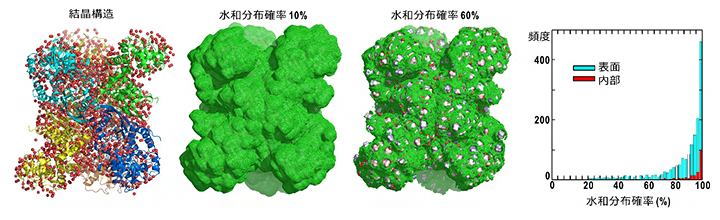
図1 人工知能(AI)が予測した水和水分子の3次元分布確
左: X線結晶構造解析で見いだされた水和水分子の位置。赤球が水分子を、リボンがタンパク質を示す。
中左・中右: AIで予測された分布確率。分布確率が10%と低い場合は、水分子を近似する直径0.3nmの球をタンパク質表面に転がして得られる水分子アクセス表面が現れる。分布確率を60%に上げると、タンパク質表面の局在候補位置が現れる。
右: 分布確率を80%以上にするとタンパク質表面に吸着した水分子の位置を、分布確率を90%以上にするとタンパク質表面だけでなく内部の水分子の位置も表すことができる。
さらに、低温X線結晶構造解析によって研究チームが解析した水和水分子を含むタンパク質構造モデルについて、AIによる予測結果とX線結晶構造解析で得られた水分子の電子密度を比較したところ、このAIが水和構造をうまく予測できることが明らかになりました(図2A~C)。また、以前に作成した経験的水和分布を用いた水和分布予測方法では、水に溶けにくいフェニルアラニン(F)などのアミノ酸で構成された表面の水和構造予測が不可能でしたが、このAIでは、それが可能であることも確認できました(図2D)。
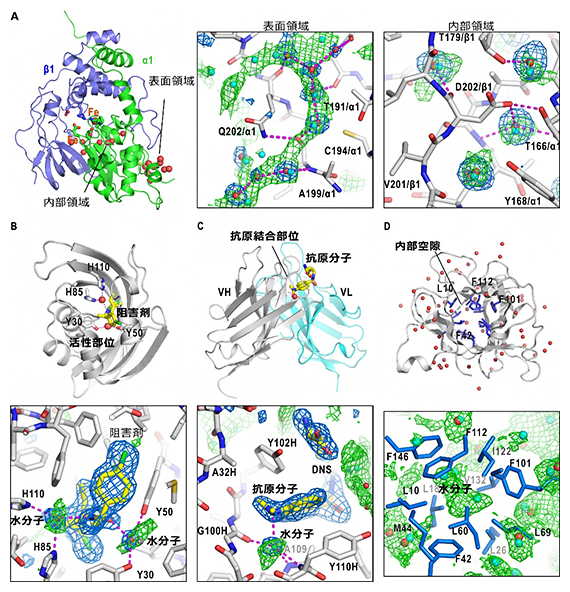
図2 水和水分子を含むタンパク質に対するAIによる水和構造予測の結果
さまざまなタンパク質構造モデルとそれらに対するAIによる水和構造予測の結果を示す。四角枠内の青メッシュは結晶構造で得られた水分子や薬剤分子の電子密度、緑メッシュはAIが予測した水和水分子分布確率を閾値80%で描いた予測結果。
A:理研が1998年に独自かつ初めて構造解析に成功した、ニトリルヒドラターゼ(ニトリルを水和し、アミドを生成する酵素)の立体構造注1)と予測結果
B:理研と日本バイエルアグロケム(当時)が2022年に可視化した、稲いもち病菌シタロン脱水酵素注2)とその阻害剤の相互作用に関わる水分子位置の予測結果。
C:抗体分子の抗原結合フラグメントにおける抗原とタンパク質の相互作用を媒介する水分子位置の予測結果。
D:インターロイキン-1β分子内部の水に溶けにくい青スティックで示すアミノ酸(L: ロイシン、I: イソロイシン、F: フェニルアラニン、M: メチオニン、V: バリン)で構成された空隙に存在する水分子分布確率予測の結果。
現在、米国や韓国で、AIを用いたタンパク質の水和構造予測が報告されています注3-4)が、それらとの性能比較では、タンパク質表面の予測水和水分子による被覆率や結晶解析で見いだされた水分子位置の再現度において、今回構築したAIが圧倒的に良好な結果を与えることも判明しました。例えば、タンパク質表面の予測水和サイトが結晶構造解析結果からどの程度ずれているかを評価すると、今回開発したAIでは、平均して0.06nm程度であったのに対して、他のAIでは、0.11nmでした。
注1)理研ニュース No.205(7)(1998)研究最前線「ニトリルヒドラターゼ:光と一酸化窒素でスイッチする有望酵素(PDF 7.7MB)」
注2)2022年3月11日プレスリリース「農薬耐性イネいもち病菌の防除化合物」
注3)Huang, P., Xing, H., Zou, X., Han, Q., Liu, K., Sun, X., Wu, J. & Fan, J. Accurate prediction of hydration sites of proteins using energy model with atom embedding. Front. Mol. Biosci. 8, 756075 (2021). doi:10.1021/acs.jcim.0c01434
注4)Park, S. & Seok, C. GalaxyWater-CNN: Prediction of water positions on the protein structure by a 3D-convolutional neural network. J. Chem. Inf. Model. 62, 3157-3168 (2022). doi:10.3389/fmolb.2021.756075
今後の期待
今後、構築したAIの予測精度をさらに高める工夫を施した後、公共データサーバーにて運用し、クライオ電子顕微鏡や分解能が十分でない結晶構造モデルの水和構造予測に供することを計画しています。また、水溶性タンパク質の結果を基に、以前に作成した親水性領域の経験的水和分布を用いた水和分布予測方法を組み合わせることで、膜タンパク質全体の水和構造予測を可能にする予定です。
このようなアプローチによって、タンパク質の立体構造モデルに水和構造を付与することにより、タンパク質の動態の解明や創薬解析に貢献することが期待できます。
補足説明
1.タンパク質の水和構造
タンパク質分子は、水溶液や脂質の中で特定の立体構造を形成する。また、その機能発現には、一定量以上の水分子がタンパク質に吸着している必要があることが知られてきた。低温X線結晶構造解析が開発されたことを契機に、タンパク質表面の水分子の吸着状態が原子レベルで可視化されるようになり、この表面やタンパク質内部の水分子の配列様態をタンパク質の水和構造と呼ぶ。水和構造は、タンパク質の構造安定化、分子認識、機能発現において不可欠な役割を担っていることが明らかにされている。
2.人工知能(AI)
ヒトの知能を研究する情報工学分野や、そこで創生される計算機上のシステム。創生されるシステムは、事例を基に訓練され、新たに与えられる問題の解決に向けた認識、推論、言語運用および創造などの知的行動を人間に代わって行う。情報工学分野で創生されてきた人工知能は、基礎および応用科学分野だけではなく、現在、日常的にも応用されるようになっている。AIはArtificial Intelligenceの略。
3.分子動力学計算
原子や分子の動きを計算機上で解析するための方法。生体高分子の場合には、水分子集団に内包された生体高分子がどのような時間変化を行うのかを、古典力学における運動方程式を数値的に解くことによって決定する。粒子間に働く力は、分子力学力場パラメーターで決められている。タンパク質のような巨大分子への分子動力学計算の適用に貢献した計算科学者には、2013年にノーベル化学賞が授与された。理研が所有するスーパーコンピュータ「富岳」では、新型コロナウイルスのスパイクタンパク質をはじめとして、創薬分野に関わる分子動力学研究が精力的に行われている。
4.クライオ電子顕微鏡
電子顕微鏡では電子線を当てて試料を観察する。電子線の波長は可視光よりもはるかに短いため、理論上0.1nm程度の分解能が得られる。ただし、電子と物質の相互作用が強く、常温下での電子線照射では生体高分子が壊れてしまう。クライオ電子顕微鏡では試料水溶液を急速凍結して、液体窒素温度付近で損傷を低減し、生体分子やその複合体の構造解析を行うことができる。この構造解析技術を開発した科学者は2017年のノーベル化学賞を受賞した。
5.分子認識
タンパク質は単独で機能する場合もあるが、他のタンパク質や小さな分子と結合して機能する場合も多い。後者では、タンパク質表面の原子配置が他の分子と弱く結合する必要がある。このような分子同士が互いの原子配置に基づいて弱く結合することを分子認識と呼ぶ。新型コロナウイルスが細胞の中に侵入する際には、ウイルスのスパイクタンパク質と、喉の細胞にあるタンパク質受容体の分子認識が鍵となっている。
6.X線結晶構造解析
対象となる分子を結晶化し、結晶にX線を照射して得られるX線回折現象から、結晶内分子の3次元構造を可視化する実験手法。理研が所有する兵庫県の播磨科学公園都市にある世界最高性能の放射光を生み出す大型放射光施設SPring-8(Super Photon ring-8 GeV)では、生体高分子のX線結晶構造解析が精力的に行われている。
7.単粒子解析法
ほぼ同一の構造を持つ粒子をさまざまな角度からランダムに撮影した多数の2次元画像から、信号/雑音比(S/N比)を向上させ、その3次元構造を再構成する方法。透過型電子顕微鏡における生体分子の構造解析において発展してきた。
8.水和水分子の分布確率
構築したAIは、タンパク質表面や内部の空隙を細かな箱に分け、各箱の中に水分子の酸素原子が存在する確率を計算する。全ての箱について予測を行えば、どのような場所に何%の確率で水分子が存在しうるかを分布として可視化できる。
9.3次元畳み込みニューラルネットワーク
畳み込みニューラルネットワークは、神経細胞(層)の連結を模した人工知能を構成するアルゴリズムの一つであり、層間を共通重みの局所結合でつないだもので、特に画像や動画認識に広く使われる。画像は2次元であるが、これを3次元データについても人工知能が対応できるように拡張されたのが3次元畳み込みニューラルネットワークである。
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「クライオ電顕画像から蛋白質の動的構造を描写するための新規計算科学手法の確立と応用(研究代表者:中迫雅由)」の助成を受けて実施されました。
原論文情報
Kochi Sato, Mao Oide and Masayoshi Nakasako, “Prediction of Hydrophilic and hydrophobic hydration structure of protein by neural network optimized using experimental data”, Scientific Reports, 10.1038/s41598-023-29442-x
発表者
理化学研究所
放射光科学研究センター 利用システム開発研究部門 生物系ビームライン基盤グループ 生命系放射光利用システム開発チーム
客員主管研究員 中迫 雅由(ナカサコ・マサヨシ)
研修生 佐藤 航地(サトウ・コウチ)
研修生(研究当時)大出 真央(オオイデ・マオ)
報道担当
理化学研究所 広報室 報道担当