2023-12-20 マサチューセッツ工科大学(MIT)
◆この研究により、低毒性でありながら抗菌活性を示す化合物が同定され、新しい抗生物質の設計に役立つ可能性があります。研究はNatureに掲載され、ディープラーニングモデルが抗生物質の強さを予測する際に使用している情報も明らかになりました。
◆これにより、モデルが予測を行う際の特徴が明らかになり、追加の抗生物質の同定や設計が容易になる可能性があります。これにより、7つの致死的な細菌に対する新しいクラスの抗生物質を7年かけて発見することを目指すMITのAntibiotics-AIプロジェクトの一環として進められています。
<関連情報>
- https://news.mit.edu/2023/using-ai-mit-researchers-identify-antibiotic-candidates-1220
- https://www.nature.com/articles/s41586-023-06887-8
説明可能なディープラーニングで抗生物質の構造クラスを発見 Discovery of a structural class of antibiotics with explainable deep learning
Felix Wong,Erica J. Zheng,Jacqueline A. Valeri,Nina M. Donghia,Melis N. Anahtar,Satotaka Omori,Alicia Li,Andres Cubillos-Ruiz,Aarti Krishnan,Wengong Jin,Abigail L. Manson,Jens Friedrichs,Ralf Helbig,Behnoush Hajian,Dawid K. Fiejtek,Florence F. Wagner,Holly H. Soutter,Ashlee M. Earl,Jonathan M. Stokes,Lars D. Renner & James J. Collins
Nature Published:20 December 2023
DOI:https://doi.org/10.1038/s41586-023-06887-8
Abstract
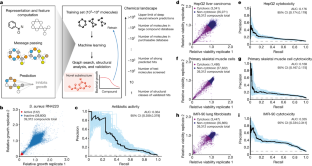
The discovery of novel structural classes of antibiotics is urgently needed to address the ongoing antibiotic resistance crisis1,2,3,4,5,6,7,8,9. Deep learning approaches have aided in exploring chemical spaces1,10,11,12,13,14,15; these typically use black box models and do not provide chemical insights. Here we reasoned that the chemical substructures associated with antibiotic activity learned by neural network models can be identified and used to predict structural classes of antibiotics. We tested this hypothesis by developing an explainable, substructure-based approach for the efficient, deep learning-guided exploration of chemical spaces. We determined the antibiotic activities and human cell cytotoxicity profiles of 39,312 compounds and applied ensembles of graph neural networks to predict antibiotic activity and cytotoxicity for 12,076,365 compounds. Using explainable graph algorithms, we identified substructure-based rationales for compounds with high predicted antibiotic activity and low predicted cytotoxicity. We empirically tested 283 compounds and found that compounds exhibiting antibiotic activity against Staphylococcus aureus were enriched in putative structural classes arising from rationales. Of these structural classes of compounds, one is selective against methicillin-resistant S. aureus (MRSA) and vancomycin-resistant enterococci, evades substantial resistance, and reduces bacterial titres in mouse models of MRSA skin and systemic thigh infection. Our approach enables the deep learning-guided discovery of structural classes of antibiotics and demonstrates that machine learning models in drug discovery can be explainable, providing insights into the chemical substructures that underlie selective antibiotic activity.