2020-04-18 東北大学,ミュンヘン大学,ケースウェスタンリザーブ大学,日本医療研究開発機構
発表のポイント
- 様々な疾患の原因となるメッセンジャー(伝令)RNA(mRNA)の安定性を決定するコドン(遺伝暗号)の最適度は、mRNAの分解に関与するCcr4-Not複合体によって監視されていることを、遺伝学・生化学的な機能解析によって証明しました。
- クライオ電子顕微鏡を用いた単粒子解析によって、Ccr4-Not複合体とリボソームの結合様式を明らかにしました。
- Ccr4-Not複合体は、発生・細胞分化や癌、炎症に寄与することが報告されており、今回の研究成果は、これまで原因不明とされてきた幅広い疾患の発症機構の解明につながることが期待されます。
概要
個々のmRNAがもつ固有の安定性は、コドンの最適度によって調節されていることが報告され、最適度が高いコドンを持つmRNAほど安定であり、最適度が低いコドンを持つmRNAは不安定であるという一般則が確立されています。mRNAの安定性制御は遺伝子発現の根幹であり、その破綻は様々な疾患の原因になります。しかし、コドンの最適度によって調節される翻訳の伸長速度を監視し、個々のmRNAがもつ固有の安定性を決定する機構は不明でした。東北大学大学院薬学研究科の稲田利文教授、松尾芳隆助教とドイツミュンヘン大学Roland Beckmann教授、ケースウェスタンリザーブ大学のJeff Coller教授らの研究グループは、mRNAの安定性を決定する新たな分子機構を発見しました。今回の研究成果は、これまで原因不明とされてきた幅広い疾患の発症機構の解明につながることが期待されます。
本研究成果は、2020年4月17日(金曜日)に米国科学誌『Science』に掲載されます。本研究は、文部科学省科学研究費補助金(基盤研究(A)、(C)、新学術領域研究「ケモテクノロジーが拓くユビキチンニューフロンティア」)、日本医療研究開発機構の革新的先端研究開発支援事業(AMED-CREST)「全ライフコースを対象とした個体の機能低下機構の解明」、(公財)武田科学振興財団、(公財)加藤記念バイオサイエンス振興財団の研究助成により実施しました。
論文名
“The Ccr4-Not complex monitors the translating ribosome for codon optimality”
(Ccr4-Not複合体は翻訳中のリボソームを介してコドンの最適度を監視する)
発表雑誌
Science
DOI : 10.1126/science.aay6912
URL:https://science.sciencemag.org/content/sci/368/6488/eaay6912.full.pdf
研究の背景
ゲノムDNA注1にコードされた遺伝情報は、mRNA注2に転写され、リボソーム注3によってアミノ酸(タンパク質)に変換されることで、遺伝子を発現します。細胞はストレスや環境変化などに対応するため、細胞内のmRNA量を調節することで遺伝子発現を制御しています。mRNAの量は合成(転写)と分解の割合で決まっており、発現量の多い遺伝子をコードするmRNAは、合成量が多いだけでなく、より安定であること(半減期が長い)が知られていました。
mRNAにコードされる遺伝情報は、リボソームによってタンパク質へと変換されます(翻訳)。この際、リボソームはmRNA上の3つの塩基配列を1つの読み枠(コドン注4)として遺伝情報を解読しますが、ほとんどのアミノ酸は複数種類のコドンによってコードされています。同じアミノ酸に対応するそれぞれのコドンは同義コドン注5とよばれ、対応するtRNA注6の細胞内存在量には偏りがあります。多く使われるコドンは最適コドンと呼ばれ、同義コドンが最適かどうかは、対応するtRNAの存在量に従って数値化され、tRNA量が高いほど最適度が高いと評価されます。従って、最適度が高いコドンが多いmRNAを翻訳するときの伸長速度は速くなり、合成されるタンパク質も多いことが知られています。翻訳の伸長速度は発現量の調節だけでなく、合成されるペプチド鎖のフォールディング注7やターゲティング注8などとも密接に連動しているため、コドンの選択は遺伝子発現において非常に重要な役割を担っています。
近年、個々のmRNAがもつ固有の安定性は、コドンの最適度によって調節されていることが報告され、最適度が高いコドンを持つmRNAほど安定であり、最適度が低いコドンを持つmRNAは不安定であるという一般則が確立されました。最適度が高いコドンを持つmRNAほど翻訳の伸長速度が早いため、コドンの最適度によってmRNAの半減期が決定されることになります。一方で、 コドンの最適度によって調節される翻訳の伸長速度を監視し、個々のmRNAがもつ固有の安定性を決定する機構は依然として不明なままでした。
研究の概要
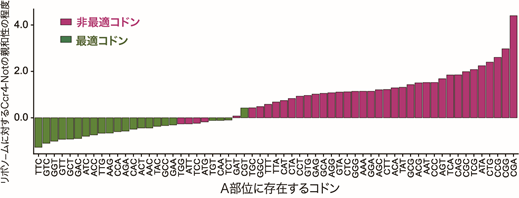
mRNAの転写・分解や翻訳抑制に関与するCcr4-Not複合体注9は、RNA結合タンパク質を介してmRNAに結合することが広く知られていますが、本研究グループは、はじめに生化学的手法を用いて、Ccr4-Not複合体がリボソームに直接結合することを見いだしました。続いて、Ccr4-Not複合体が特異的に結合するリボソームによって翻訳されるmRNAの特徴を調べるために、選択的リボソームプロファイリング注10による網羅的解析を行いました。その結果、コドンレベルの解析では、Ccr4-Not複合体とリボソームの親和性がコドンの最適度と非常に強い逆相関を示すことがわかりました(図1)。
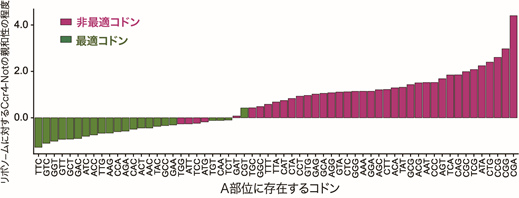
図1 Ccr4-Notとリボソームの親和性は最適コドンに強い逆相関を示す
縦軸はリボソームに対するCcr4-Not複合体の親和性を、横軸はリボソームのA部位のコドンを示す。非最適コドン(赤)を含むリボソームはCcr4-Not複合体に高い親和性を示し、最適コドン(緑)を含むリボソームは低い親和性を示した。従って、Ccr4-Not複合体が結合するリボソームのA部位には、より最適度の低いコドンが存在することが明らかになった。
また、研究グループはCcr4-Not複合体の機能欠損によって、コドンの最適化によるmRNAの安定性制御が失われることも示しました。つまり、コドンの最適度が高い(最適コドンが多い)mRNAでおこる安定化や、コドンの最適度が低い(最適コドンが少ない)mRNAでおこる不安定化がみられなくなりました。以上の結果より、Ccr4-Not複合体は、コドンの最適度が低いmRNAを翻訳するリボソームに対して強い親和性をもつことで、mRNAを分解に導くことが明らかになりました。
次に、研究グループは、リボソームに結合したCcr4-Not複合体の構造を決定しました。クライオ電子顕微鏡注12を用いた単粒子解析で、Ccr4-Not複合体の構成タンパク質の1つであるNot5が、A部位にtRNAを含まないリボソームのE部位に結合することを見いだしました(図2)。翻訳過程において、コドン-アンチコドン注11の認識はリボソームのA部位によって行われます。非最適コドンを翻訳しているリボソームでは、対応するtRNA の存在量が少ないため、A部位にtRNAが結合するまでに長い時間が必要となります。A部位にtRNAが結合しないため、E部位からtRNAが解離します。tRNAが解離し空になったE部位にCcr4-Not複合体が結合し、効率よくmRNAを分解に導くことがわかりました。 以上の結果より、Ccr4-Not複合体は、非最適コドンを翻訳中のリボソームのE部位に効率よく結合することで、コドンの最適度を監視し、mRNAの安定性を制御することが明らかになりました(図3)。
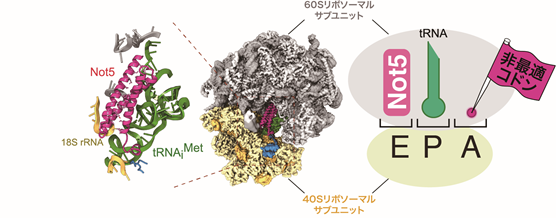
図2 Not5とリボソームの結合様式
左図:クライオ電子顕微鏡注12を用いたCcr4-Not複合体とA部位にtRNAを含まないリボソームの単粒子解析。Ccr4-Not複合体の構成タンパク質の1つであるNot5が、リボソームのE部位に結合している。ピンクはNot5のアミノ末端領域、緑はtRNA、黄色は40Sリボソーマルサブユニット、灰色は60Sリボソーマルサブユニットを示す。
右図:リボソームのA部位に非最適コドンが存在する場合、Not5がE部位に結合することで mRNAの分解を引き起こす。
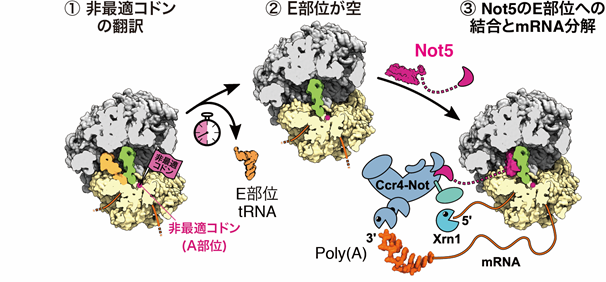
図3 コドンの最適度に依存したmRNAの分解制御機構モデル
① 非最適コドンがA部位に位置したリボソーム。tRNAの存在量が少ないため、A部位にtRNAが結合するまでの時間が長い。② A部位にtRNAが結合しないため、E部位からtRNAが解離する。③Ccr4-Not複合体のNot5 サブユニットのN末端領域がE部位に結合する。リボソームに結合したCcr4-Not複合体がポリAを短鎖化する。さらにキャップ構造の除去後にリボヌクレアーゼXrn1がmRNAを分解する。ピンクはNot5のアミノ末端領域、マジェンダはE部位に結合したtRNA、緑はP部位に結合したtRNA、黄色は40Sリボソーマルサブユニット、灰色は60Sリボソーマルサブユニット、オレンジは mRNAを示す。
社会的意義と今後の展望
遺伝子の発現制御は生命の根幹であり、その分子機構を理解することは、様々な生命現象、あるいは幅広い疾患の病態の理解につながります。細胞は、絶えずストレスや環境の変化に適応するため、細胞内のmRNA量を制御しています。その制御は、mRNAの合成(転写)と分解によって行われており、それぞれが協調的に機能することが重要です。本研究により、コドンの最適化によるmRNAの分解制御機構の実態が明らかになり、長年不明であった遺伝情報における同義コドンの存在意義がより明確になりました。
タンパク質合成途中の翻訳速度調節の異常は、タンパク質の機能に重大な欠陥を引き起こし、タンパク質恒常性の破綻につながります。タンパク質恒常性の破綻は、不良タンパク質の蓄積やオルガネラの損傷、シグナル伝達経路のかく乱など、広範な細胞機能の障害を引き起こすため、アルツハイマー病やパーキンソン病などの神経変性疾患や老化の原因になると考えられます。今回の研究成果は、翻訳異常によって機能欠損タンパク質が合成されることで発症する疾患の発症機構や老化のメカニズムを理解するための基盤になることが期待されます。
用語説明
注1)ゲノムDNA:
各種生物がもつ遺伝情報が全て含まれるDNAセット。
注2)mRNA:
メッセンジャーRNAの略で日本語では伝令RNA。タンパク質合成の設計図となる遺伝情報を持つRNA。
注3)リボソーム:
mRNAのもつ遺伝情報に従ってアミノ酸同士を結合させ、タンパク質を合成する装置。タンパク質とRNAから構成される巨大な複合体であり、tRNAと結合するA、P、E部位をもつ。
注4)コドン:
mRNAの塩基配列からタンパク質を構成するアミノ酸配列へ翻訳される際、リボソームのA部位によって認識される連続した3つの塩基配列。
注5)同義コドン:
同じアミノ酸に対応する複数のコドン。アミノ酸は20種類でコドンは64種類。コドンの数がアミノ酸より多いため、ほとんどのアミノ酸には対応するコドンが複数存在する。
注6)tRNA:
トランスファーRNAの略で日本語では運搬RNA。mRNAのコドンに対応するアンチコドンをもち、特定のアミノ酸と結合し、リボソームによる翻訳過程においてアミノ酸を供給する。
注7)ペプチド鎖のフォールディング:
翻訳反応によって合成されたアミノ酸が結合したポリペプチド鎖が立体構造に折りたたまれる現象。タンパク質が機能を獲得するには、正しい立体構造を形成する必要がある。
注8)ターゲティング:
細胞内でタンパク質が機能する特定の場所へ輸送されること。膜タンパク質などは、共翻訳的に小胞体へと輸送される。
注9)Ccr4-Not複合体:
酵母からヒトまで保存されたタンパク質複合体。遺伝子発現調節因子として転写調節やmRNAの分解に関与する。
注10)リボソームプロファイリング:
翻訳中のリボソームが結合するmRNA領域を次世代シークエンスによって網羅的に解析する方法。mRNA上でリボソームが翻訳する様子をスナップショットとして可視化することができる。
注11)コドン-アンチコドン:
mRNAがもつコドンとtRNAがもつアンチコドンによる塩基対形成。翻訳過程では、コドンとアンチコドンの塩基対形成によって正しいtRNA(アミノ酸)を認識する。
注12)クライオ電子顕微鏡:
試料を低温(液体窒素と同程度)のまま観察できる装置を備えた高性能な透過型電子顕微鏡。
お問い合わせ先
研究に関すること
東北大学大学院薬学研究科 稲田 利文 教授
東北大学に関すること
薬学研究科事務 星野 公太郎
AMEDの事業に関すること
国立研究開発法人日本医療研究開発機構
シーズ開発・研究基盤事業部 革新的先端研究開発課