2018-05-08 理化学研究所,京都大学
理化学研究所(理研)眞貝細胞記憶研究室の加藤雅紀協力研究員(研究当時)、開拓研究本部眞貝細胞記憶研究室の眞貝洋一主任研究員と京都大学ウイルス・再生医科学研究所竹本経緯子助教らの共同研究チームは、哺乳動物の体細胞で内在性レトロウイルス(ERV)[1]の発現を抑制する普遍的な仕組みを明らかにしました。
本研究成果は、ERVの発現が引き起こす自己免疫疾患[2]などの原因解明に役立つと期待できます。
哺乳動物のゲノム中、ERV由来の配列は約10%存在します。ERVの異常な発現は、近傍遺伝子の発現への影響や、ERV由来RNAに対する免疫応答反応を引き起こします。体細胞におけるERVの抑制には、DNAのメチル化[3]が重要な役割を果たしています。今回、共同研究チームは、ヒストンの9番目のリジンのトリメチル化(H3K9me3)修飾を担うヒストンメチル化酵素Setdb1遺伝子をさまざまな種類のマウス体細胞でノックアウト[4]し、ERVの脱抑制を網羅的に検証しました。その結果、Setdb1ノックアウト体細胞ではERV領域のH3K9me3修飾が一様に低下するが、それぞれの細胞に特徴的なERVの種類が脱抑制することが明らかになりました。また、DNAのメチル化はERVの抑制には限定的であること、組織特異的な転写因子[5]がERVの発現に重要であることが分かりました。本研究により、H3K9me3は従来の見解と異なり、どの細胞種においても普遍的な抑制エピゲノムとしてERVの抑制に寄与する可能性を提示しました。
本研究は、国際科学雑誌『Nature Communications』(4月27日付)に掲載されました。
※研究支援
本研究は、日本医療研究開発機構(AMED)国際科学技術共同研究推進事業(戦略的国際共同研究プログラム「細胞移植治療の実現に向けた細胞アイデンティティー制御」)による支援を受けて行われました。
背景
哺乳動物のゲノムの約半分は、動く遺伝子とも呼ばれるトランスポゾン[6]由来の配列で占められています。なかでも内在性レトロウイルス(ERV)由来の配列は、ゲノムの約10%存在しています。ERVの異常発現は、近傍遺伝子の発現への影響やゲノムの不安定性、ERV由来RNAに対する免疫応答反応を引き起こすことが知られています。このため、エピジェネティック修飾[7]と呼ばれるDNAのメチル化やヒストン修飾[8]によって、ERVの発現は抑制されています。
DNAのメチル化は酵素Dnmt1によって維持されており、Dnmt1遺伝子のノックアウトマウスの胎児では、ERVの一種であるIAP(intracisternal A particles)の転写が劇的に上昇します注1)。このことから、体細胞におけるERVの抑制にはDNAのメチル化が重要であると考えられてきました。
一方、胚の発生初期や始原生殖細胞の発生時期には、DNAのメチル化が大きく低下します。そのため、DNAのメチル化に代わるバックアップ機構として、ヒストンH3の9番目のリジンのトリメチル化(H3K9me3)およびその修飾を行うヒストンメチル化酵素Setdb1が重要な役割を果たすことが報告されています注2)。
しかし、これまで体細胞のERV領域におけるH3K9me3修飾の役割については明らかになっていませんでした。
注1)Walsh C P, Chaillet J R & Bestor T H. Transcription of IAP endogenous retroviruses is constrained by cytosine methylation. Nat Genet. 20, 116-7.(1998)
注2)Matsui T, Leung D, Miyashita H, Maksakova I A, Miyachi H, Kimura H, Tachibana M, Lorincz M C & Shinkai Y. Proviral silencing in embryonic stem cells requires the histone methyltransferase ESET. Nature 464, 927-31.(2010)
研究手法と成果
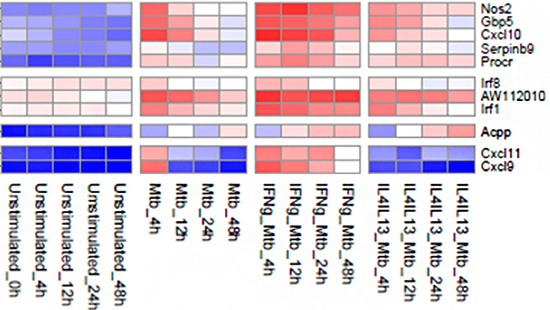
共同研究チームは、H3K9me3修飾を担うヒストンメチル化酵素の一つであるSetdb1遺伝子をマウスES細胞[9]およびさまざまな体細胞でノックアウトし、ERVが脱抑制するか、次世代シーケンサー[10]を用いて網羅的に検証しました。その結果、ES細胞のみならず、体細胞においても、それぞれの細胞に特異的なERVの種類が脱抑制することが明らかになりました(図1)。
さらに、マウス線維芽細胞[11]でSetdb1ノックアウト細胞を作製し、それぞれのERV領域におけるH3K9me3の量を網羅的に検証したところ、ほぼ全てのERV領域でH3K9me3の減少が見られました。ただし、マウス線維芽細胞では主にVL30ファミリー[12]に属するERVのみが脱抑制することから、H3K9me3はほぼ全てのERVの発現を抑制しうるが、それに加えて発現を調節する仕組みが存在する可能性が考えられます。
共同研究チームは、このVL30ファミリーに注目し、Setdb1ノックアウトマウス線維芽細胞では、LTR[13]のクラスによって4種類ある中でもクラス1のみしか脱抑制しないことを見いだしました。マウスゲノム中に存在するVL30クラス1は71コピーですが、コピーごとに脱抑制度合いが異なります。脱抑制の弱いコピーのDNA配列に注目すると、MAPK経路[14]の下流に位置する転写因子の結合配列が破壊されていることから、MAPK経路がVL30クラス1の発現に重要であることが明らかになりました。
また、DNAのメチル化を担う酵素であるDnmt1をマウス線維芽細胞でノックダウン[15]したところ、予想に反してほとんどのERVは脱抑制せず、IAPファミリーのみ脱抑制することが明らかとなりました。またSetdb1のノックアウトによってもDNAのメチル化は減少しませんでした。これらの結果から、DNAのメチル化はERVの抑制に対して限定的であること、ヒストンH3K9me3がどの細胞種においても普遍的にERVの抑制に寄与しうること、しかしERVが脱抑制するためにはそれぞれの細胞種においてそれぞれのERVに必須な転写因子の活性化が必要であることを示しています(図2)。
今後の期待
これまで、DNAのメチル化が体細胞においては一般的にERVの発現抑制に重要であると考えられていましたが、本研究の結果から、ヒストンH3K9me3修飾がERVの抑制に普遍的な役割を持つ可能性が明らかとなりました。
ERVの異常な発現は自己免疫疾患を引き起こすことが示されており、ERVの抑制機構についての研究は、将来的には自己免疫疾患の発症メカニズムの解明につながると期待できます。
原論文情報
Masaki Kato, Keiko Takemoto, Yoichi Shinkai, “A somatic role for the histone methyltransferase Setdb1 in endogenous retrovirus silencing”, Nature Communications, 10.1038/s41467-018-04132-9
発表者
理化学研究所
開拓研究本部 主任研究員研究室 眞貝細胞記憶研究室
協力研究員(研究当時) 加藤 雅紀(かとう まさき)
(現 生命医科学研究センター トランスプリクトーム研究チーム 上級研究員)
主任研究員 眞貝 洋一(しんかい よういち)
京都大学 ウイルス・再生医科学研究所
助教 竹本 経緯子(たけもと けいこ)
報道担当
理化学研究所 広報室 報道担当
京都大学 総務部 広報課 国際広報室
補足説明
-
- 内在性レトロウイルス(ERV)
- トランスポゾンの一種である。ゲノム中のおそらくレトロウイルス由来の配列であり、ヒトゲノム中の約8%、マウスゲノムの約10%を占めると考えられる。外来性レトロウイルスが生物に感染し、生殖細胞ゲノムに取り込まれ、次世代に受け継がれて生物ゲノム中に定着したもの。多くのERVは内部の配列に変異を持つものが多く、トランスポゾンとしての活性を失っているものが多い。ERVはEndogenous Retrovirusesの略。
-
- 自己免疫疾患
- 何らかの免疫異常によって自分の身体を構成する物質を異物のように認識し、自己抗体や自己に反応するリンパ球を作り、自分の組織を攻撃する疾患。代表的なものに関節リウマチなどあるが、根本的な治療法は見つかっていない。
-
- DNAのメチル化
- DNAを構成する塩基のシトシン(C)にメチル基を付加する反応。遺伝子の制御領域の高度なメチル化が、遺伝子発現を抑制すると考えられている。
-
- ノックアウト
- 目的の遺伝子を人為的に欠失させること。
-
- 転写因子
- DNAに結合するタンパク質。遺伝子の発現量を調節する機能があり、発生や成長に重要な役割を担う。
-
- トランスポゾン
- 細胞内でゲノム上の位置を移動(転移)することができるDNAの単位。動く遺伝子。ヒトゲノムの半分以上を占める。
-
- エピジェネティック修飾
- 遺伝情報を伝達するDNAの塩基配列自体には変更を加えず、DNAのメチル化やヒストン修飾などの化学修飾(メチル基やアセチル基、リン酸基等が可逆的に付加されること)を介して、ゲノムDNAの機能に変化をもたらす修飾のこと。
-
- ヒストン修飾
- ヒストンとは染色体を構成する主要なタンパク質である。染色体は、ヒストンH2A、H2B、H3、H4のそれぞれ2分子からなる八量体に、DNAが左巻きに1.65回巻きついた複合体を基本単位(ヌクレオソーム)として存在している。ヌクレオソームから各ヒストンのアミノ酸末端領域が突出しており、メチル化やアセチル化、リン酸化などの修飾を受けることにより、染色体構造に変化をもたらし、遺伝子発現の抑制や活性化を調節する。
-
- ES細胞
- 脊椎動物の初期胚が持つ、全ての種類の体細胞へ分化する能力を多能性という。多能性を持ち、試験管内で培養して無限に増やすことができる細胞を多能性幹細胞という。ES細胞は、哺乳類の着床前胚(胚盤胞)に存在する内部細胞塊から作製された多能性幹細胞。
-
- 次世代シーケンサー
- サンガー法を利用した蛍光キャピラリーシークエンサーである「第一世代シークエンサー」と対比させて使われる用語。機種によるが、一度に107~1010(1,000万~100億)個のDNAの配列を決定できる。以前はヒトのゲノム配列を決定するのに14年程度かかったが、次世代シークエンサーを用いると、数日で決定できる。
-
- 線維芽細胞
- 結合組織を構成する細胞の一つ。コラーゲン、エラスチン、ヒアルロン酸といった真皮の成分を作り出す。核小体が明瞭な楕円形の核を有し、細胞質は塩基好性を示す。
-
- VL30ファミリー
- ERVのファミリーの一種。げっ歯類のみに存在する。
-
- LTR
- ウイルスゲノムの両末端に存在する、数百塩基の長い繰り返し配列。ウイルスの転移活性に必要なほか、さまざまな転写調節配列が存在する。LTRはLong terminal repeatの略。
-
- MAPK経路
- MAPキナーゼ(mitogen-activated protein kinase, MAPK)経路は細胞の増殖、分化、ストレス応答などに関わるセリン/セレオニン プロテインキナーゼファミリーのシグナル伝達経路であり、真核生物のあいだで広く保存されている。
-
- ノックダウン
- mRNAの分解促進でタンパク質の量を著しく低下させることにより、遺伝子の機能を大きく減衰させる方法。遺伝子破壊以外の操作により行う場合が多い。

図1 Setdb1遺伝子のノックアウトとERVの脱抑制
いくつかのマウス細胞でSetdb1遺伝子をノックアウトすると野生型細胞(WT)に比べてERVの発現量が上昇(脱抑制)するものがある(マゼンタ色のほうが緑色に比べて発現量が高い)。どの種類のERVが脱抑制するかはそれぞれの細胞で特徴を有する。
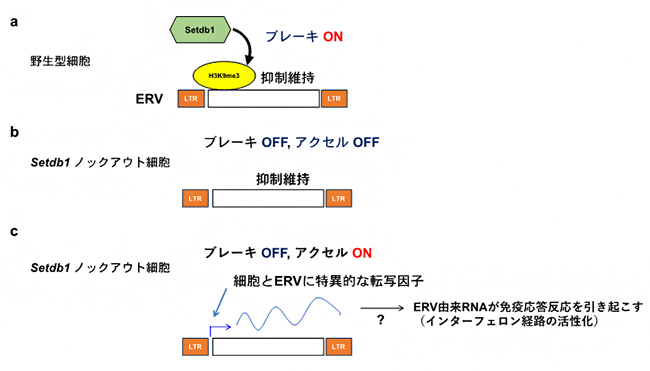
図2 本研究で提唱したモデル
a) 野生型の細胞では、ヒストンメチル化酵素Setdb1がERV領域にヒストンH3K9me3修飾を入れることによってブレーキの役割を果たし、ERVの発現を抑制する。
b) Setdb1ノックアウト細胞では、H3K9me3修飾がなくなりブレーキが外れる。ただし、アクセルとなるものがない場合、ERVの発現は誘導されないままである。
c) Setdb1ノックアウト細胞で、それぞれのERVの発現に必要な転写因子がその細胞で発現もしくは活性化されているときのみ、ERVが脱抑制される。