2019-05-14 理化学研究所,兵庫県立大学,大阪大学,日本医療研究開発機構
理化学研究所(理研)生命機能科学研究センター翻訳構造解析研究チームの伊藤拓宏チームリーダー、岩崎わかな専任研究員、タンパク質機能・構造研究チームの横山武司研究員、兵庫県立大学大学院工学研究科の今高寛晃教授、町田幸大助教、大阪大学蛋白質研究所の多田隈尚史助教らの共同研究グループ※は、細胞に感染したC型肝炎ウイルス(HCV)[1]がヒトの翻訳装置を乗っ取る新しい仕組みを発見しました。
本研究成果は、新たな抗ウイルス薬の開発などに貢献すると期待できます。
HCVが引き起こすC型肝炎は、肝がんにつながる重大な病気です。1本鎖RNAをゲノムに持つHCVは、感染した細胞内で自身のRNAを宿主の翻訳装置であるリボソーム[2]に読み取らせ、ウイルス増殖に必要な多数のタンパク質を効率良く合成します。ウイルスタンパク質の効率良い合成には、HCVのRNA配列に存在する「IRES[3]」と呼ばれる領域が寄与していることは分かっていましたが、IRESがどのように翻訳効率を向上させているかは謎でした。
今回、共同研究グループは、クライオ電子顕微鏡[4]による立体構造解析や蛍光顕微鏡による1分子観察[5]などの最先端の計測技術により、ヒトタンパク質を合成している最中のリボソームに、HCVのIRESが結合し、次に始まる翻訳反応でHCVゲノムを優先的に読み取らせていることを明らかにしました。この乗っ取り機構は、HCVのみならずその近縁のウイルスでも保存されていると考えられ、ウイルスの感染力の源となっているといえます。
本研究は、米国の科学雑誌『Molecular Cell』のオンライン版(5月9日付け:日本時間5月10日)に掲載されました。
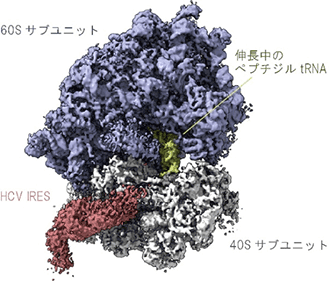
図 翻訳反応中のリボソームにHCV IRESが結合しているのを捉えたクライオ電子顕微鏡構造
- ※共同研究グループ
- 理化学研究所
- 生命機能科学研究センター
- 翻訳構造解析研究チーム
- チームリーダー 伊藤 拓宏(いとう たくひろ)
- 専任研究員 岩崎 わかな(いわさき わかな)
- 技師 西本 まどか(にしもと まどか)
- 技師 髙橋 真梨(たかはし まり)
- 技師 坂本 恵香(さかもと あやこ)
- タンパク質機能・構造研究チーム
- チームリーダー 白水 美香子(しろうず みかこ)
- 研究員 横山 武司(よこやま たけし)
- 創薬タンパク質解析基盤ユニット
- テクニカルスタッフI 米持 まゆ美(よねもち まゆみ)
- 放射光化学研究センター
- 利用システム開発研究部門 生物系ビームライン基盤グループ
- 研究員 重松 秀樹(しげまつ ひでき)
- 兵庫県立大学 工学研究科 物質系工学専攻
-
- 教授 今高 寛晃(いまたか ひろあき)
- 助教 町田 幸大(まちだ こうだい)
- 研究員(研究当時) 重田 友明(しげた ともあき)
- 大阪大学 蛋白質研究所
-
- 教授 原田 慶恵(はらだ よしえ)
- 助教 多田隈 尚史(ただくま ひさし)
- ※研究支援
- 本研究は、日本学術振興会(JSPS)科学研究費補助金 新学術領域研究(研究領域提案型)「ピコルナウイルスの2Aペプチドの終止コドン非依存的翻訳終結の構造基盤(研究代表者:伊藤拓宏)」、「ウイルスが引き起こす非標準的な翻訳機構の構造基盤(研究代表者:伊藤拓宏)」、「AMPA型グルタミン酸受容体の速くて複雑な動的構造の活写(研究代表者:重松秀樹)」、「膜蛋白質の速くて複雑な動的機能構造を活写するハイブリッド構造解析基盤(研究代表者:重松秀樹)」、「ncRNA作動装置構築の分子動態解析基盤の開発(研究代表者:泊幸秀、研究分担者:多田隈尚史)」、同基盤研究(B)「HCV IRESによるリボソーム・ハイジャックの分子機構(研究代表者:伊藤拓宏)」、「動物RNAウイルス生活環の試験管内再現(研究代表者:今高寛晃)」、「分子間距離で紐解く、遺伝子発現反応場の再構成(研究代表者:多田隈尚史)」、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)「統合的構造解析に向けた高難度複合体の生産支援と高度化(研究代表者:白水美香子)」、および武田科学振興財団ライフサイエンス研究奨励「真核生物の翻訳開始制御機構の解明(研究代表者:伊藤拓宏)」による支援を受けて行われました。
1.背景
ウイルスは、生物と同様に自身のゲノム(DNAまたはRNA)と、それを格納する構造体を持っています。しかし、ウイルスは自分のみでは増殖することができず、宿主細胞を利用して自己複製を行います。そこでウイルスは、細胞に侵入したり、細胞の持つタンパク質を利用したりするための巧妙な仕組みを多く備えています。ある種のウイルスは細胞を機能不全へと導き、最終的に病気を引き起こすことが知られています。HCVはC型肝炎の原因ウイルスであり、1本鎖RNAをゲノムとして持っています。
遺伝情報が記されているメッセンジャーRNA(mRNA)の配列に基づいてアミノ酸を連結し、タンパク質を合成する過程を「翻訳」と呼びます。翻訳反応系は多くのタンパク質と核酸が関わり、細胞のエネルギーの約半分を消費する複雑な反応系です。ウイルスが細胞内で増殖して、再び構造体を形成して細胞外へと出るためには、細胞内で働いている翻訳反応を利用してウイルスタンパク質を作り出す必要があります。細胞内の翻訳反応を担う翻訳装置は、「リボソーム」という巨大なタンパク質・核酸複合体です。リボソームは大小ニつのサブユニットからなる翻訳装置であり、真核生物の場合はそれぞれ「60Sサブユニット」、「40Sサブユニット」と呼ばれています。
真核生物のmRNAの末端には「キャップ[6]」という構造が付加されており、ここに翻訳の開始に関わるタンパク質「翻訳開始因子[7]」群(数十種類よりなる)と、40Sサブユニット、60Sサブユニットが集まります。こうして、60Sサブユニットと40Sサブユニットに挟まれた形で、mRNA上の翻訳開始点から翻訳反応が始まります。一方、HCVをはじめとするある種のウイルス由来のRNAはキャップ構造を持たず、リボソームと限られた種類の翻訳開始因子のみを用いてウイルスタンパク質の合成を効率良く開始します。
HCVは、ゲノム1本鎖RNAがそのままmRNAとして機能しますが、その末端に存在する「IRES」と呼ばれる配列が、効率の良い翻訳開始を担っています。IRESの役割については、多くの研究が精力的に進められています。これまでの知見では、IRESと翻訳開始因子のリボソーム上の結合部位が同じ場所であることから、宿主のmRNAの翻訳反応に使われていない空の40SサブユニットにIRESが結合することによって、HCVゲノムRNAの翻訳開始が誘導されると考えられてきました。しかし、この仮説を直接立証した観察はありませんでした。
2.研究手法と成果
共同研究グループはまず、HCVのIRESがどのような状態のリボソームに結合しているかを解析しました。その結果、従来から唱えられていた通り、HCVのIRESは40Sサブユニットに結合していましたが、意外なことに、すでに他のmRNAを翻訳中のリボソームにも結合していることを見いだしました。
そこで、その状態を詳しく調べるために、通常のヒト細胞の中で起こっているのと同じように、キャップ構造を持つmRNAと翻訳中のリボソームを試験管内に準備し、そこにHCVのIRESを加えた試料をクライオ電子顕微鏡で観察しました。するとIRESが、進行中のmRNAの翻訳反応を邪魔することなく、40Sサブユニットに結合している構造を捉えることに成功しました(図1)。
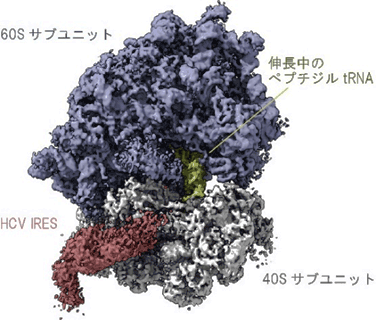
図1 翻訳反応中のリボソームに結合するIRESを捉えたクライオ電子顕微鏡像
mRNAのキャップ構造に依存して宿主細胞の翻訳を開始したリボソームに結合したC型肝炎ウイルス(HCV)のIRES(赤)と、合成途中のタンパク質(ペプチド)を結合したペプチジルtRNA(緑)が同時に観察されたことから、IRESは進行中のmRNAの翻訳反応を邪魔することなく、40Sサブユニットに結合しているのが分かる。HCVのゲノムRNAは1本鎖であるため、分子内の相補的な塩基同士が水素結合し、IRESの領域は特に複雑な立体構造をとる。
さらに、蛍光顕微鏡を用いた1分子観察からも、IRESが確かに翻訳反応中のリボソームに結合していることを確認できました。これらの結果から、IRESは40Sサブユニットに結合しながら、宿主のmRNAの翻訳反応が終結するのを待ち構え、その後にリボソームを「乗っ取る」と考えられます。
それでは、HCVゲノムを翻訳中のリボソームに対しても、IRESはリボソームに結合し続けることができるのでしょうか。この問いに答えるため、IRESに依存して翻訳を開始した場合についても試験管内で試料を調製し、クライオ電子顕微鏡で観察しました。すると、やはりIRESが翻訳反応中のリボソームの40Sサブユニットに結合していることが分かりました。
また、生化学的な実験により、HCVゲノム自身の翻訳(IRESに依存した翻訳)の開始は、キャップ構造を持つmRNAを翻訳中のリボソームを用いた方が、翻訳反応に使われていない空のリボソームを用いるよりも、迅速に進むことが明らかになりました。この結果とクライオ電子顕微鏡による観察結果と併せると、従来の「使われていない40SサブユニットにHCVのIRESが結合して、HCVのタンパク質合成が開始される」モデル(図2のⅥ→Ⅲ→Ⅳ→Ⅴの流れ)よりも、「細胞のmRNAを翻訳中のリボソームにHCVのIRESが結合し乗っ取る」(図2のⅠ→Ⅱ→Ⅲ→Ⅳ→Ⅴ)場合の方が、より効率よく頻繁に起こると考えられます。さらに、HCVゲノムの翻訳に使われている40Sサブユニットを、次のラウンドの翻訳の際にもう一度使う(図2のⅤ→Ⅲ)といったシステムも存在しうることが示されました。
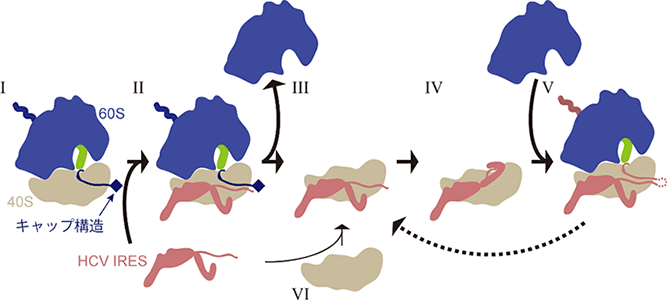
図2 HCVのIRESが翻訳装置リボソームを乗っ取る過程の模式図
翻訳中のリボソームを構成する60Sサブユニット(青)、40Sサブユニット(灰)、ペプチジルtRNA(緑)および翻訳されるmRNAを模式的に示す。mRNAのキャップ構造依存的に翻訳中のリボソーム(Ⅰ)の40SサブユニットにHCVのIRESが結合し(Ⅱ)、翻訳が終了すると(Ⅲ)、結合していたIRESは下流のウイルスRNAの翻訳を誘導し(Ⅳ)、ウイルスタンパク質の合成が開始する(Ⅴ)。同じHCVのIRESと40Sサブユニットのペアが、次のラウンドの翻訳をもう一度担っているかもしれない(Ⅴ→Ⅲ)。翻訳に使われていないリボソームに、HCVのIRESが結合する場合もある(Ⅵ)。
3.今後の期待
クライオ電子顕微鏡をはじめとした先端的な計測手法と、試験管内再構成という生化学的な手法の組み合わせは、日本の構造生物学研究の強みの一つです。本研究は、HCVのIRESが、これまで想定されていなかった方法で反応中の翻訳装置リボソームを乗っ取っていることを示した点で画期的です。今回明らかになった機構は、HCVのIRESと同じような構造を持つウイルスに共通すると考えられます。従って、IRESとリボソームの40Sサブユニットとの相互作用を阻害する薬剤を開発できれば、効果的な抗ウイルス薬になる可能性があります。
翻訳は、生物が持つ最も基本的な反応の一つです。その機構の解明は分子生物学の古典的な課題ですが、従来は見過ごされてきた新しい翻訳機構や翻訳制御機構が翻訳反応系に潜んでいる可能性があります。今後のさらなる研究によって、それらの機構が明らかになっていくと期待できます。
4.論文情報
- タイトル
- HCV IRES captures an actively translating 80S ribosome
- 著者名
- Takeshi Yokoyama, Kodai Machida, Wakana Iwasaki, Tomoaki Shigeta, Madoka Nishimoto, Mari Takahashi, Ayako Sakamoto, Mayumi Yonemochi, Yoshie Harada, Hideki Shigematsu, Mikako Shirouzu, Hisashi Tadakuma, Hiroaki Imataka, and Takuhiro Ito
- 雑誌
- Molecular Cell
- DOI
- 10.1016/j.molcel.2019.04.022
5.補足説明
- [1]C型肝炎ウイルス(HCV)
- 1本鎖RNAウイルスで、約9.6 kbのゲノムが三つの構造タンパク質(Core、E1、E2)と七つの非構造タンパク質(p7、NS2、NS3、NS4A、NS4B、NS5A、NS5B)をコードしている。通常、ヒトとチンパンジーにしか感染することができず、全世界で約1億7000万人がHCVに感染していると推測されている。HCVはhepatitis C virusの略。
- [2]リボソーム
- メッセンジャーRNAにコードされた遺伝子情報を、タンパク質を構成するアミノ酸の配列へと「翻訳」する、細胞内のタンパク質合成工場。コア領域はRNAで構成され、その外側をタンパク質が取り囲む、巨大なRNAタンパク質複合体。大サブユニットと小サブユニットの二つのサブユニットから構成され、真核生物の場合はそれぞれ60Sサブユニットと40Sサブユニットとも呼ばれる。
- [3]IRES
- 真核生物の通常の翻訳開始過程は、mRNAの5’末端のキャップ構造を翻訳開始因子群が認識し、リボソームを呼び込む。一方で、IRES配列を持つmRNAは、IRES RNAが立体構造を組むことで、直接リボソームを呼び込む。そのため、キャップ構造非依存的に翻訳過程を開始できるので、一部のRNAウイルスが、自身の遺伝子から効率良くタンパク質を生成するのに用いている。IRESは、Internal Ribosome Entry Siteの略。
- [4]クライオ電子顕微鏡
- 生体試料を液体エタンにより急速凍結し、ガラス状の氷に閉じ込め、透過型電子顕微鏡を用いて直接観察する手法。画像処理技術を駆使することで、リボソームなどの超分子複合体や、タンパク質複合体の三次元立体構造を得ることができる。
- [5]1分子観察
- 蛍光分子1個が出す光を捉える顕微鏡により、分子の挙動を1分子レベルで追跡するイメージング技術を用いた観察。
- [6]キャップ
- 真核生物のmRNAの5’末端に特徴的な構造。核酸の重合は通常5’→3’で結合が進むが、mRNAの5’末端は修飾反応によりグアノシンが5′-5’結合となっている。mRNAの5’末端を保護するためや、翻訳開始因子とmRNAの結合に関わっている。
- [7]翻訳開始因子
- 細胞内でタンパク質の合成(翻訳)を行うリボソームが、合成を開始する際に協調的に働くタンパク質群。真核生物の翻訳開始因子は真核生物型開始因子(eIF; eukaryotic Initiation Factor)と呼ばれる。
6.発表者・機関窓口
発表者
- 理化学研究所 生命機能科学研究センター
- 翻訳構造解析研究チーム
- チームリーダー 伊藤 拓宏(いとう たくひろ)
- 専任研究員 岩崎 わかな(いわさき わかな)
- タンパク質機能・構造研究チーム
- 研究員 横山 武司(よこやま たけし)
兵庫県立大学 工学研究科 物質系工学専攻
教授 今高 寛晃(いまたか ひろあき)
助教 町田 幸大(まちだ こうだい)
大阪大学 蛋白質研究所
助教 多田隈 尚史 (ただくま ひさし)
機関窓口
理化学研究所 生命機能科学研究センター センター長室
報道担当 山岸 敦(やまぎし あつし)
理化学研究所 広報室 報道担当
兵庫県立大学 姫路工学キャンパス 経営部総務課
後藤 綾一(ごとう りょういち)
大阪大学 蛋白質研究所 庶務係
和田 由美(わだ ゆみ)
日本医療研究開発機構(AMED)創薬戦略部 医薬品研究課