2024-01-24 東京大学
発表のポイント
- タンパク質の合成過程(翻訳)での植物の無機栄養の感知とそれに伴うタンパク質合成過程の変化が分子レベルで解明されました。これまで知られていなかった80Sリボソーム複合体がmRNA上を滑って移動するプロセスが翻訳制御に重要であることが明らかになりました。
- 翻訳を通じた植物の無機栄養の欠乏に対する反応の分子機構が初めて明らかにされました。
- この翻訳制御は植物の栄養吸収を担う遺伝子を栄養条件に応じて厳密に発現させるために不可欠な仕組みであり、この仕組みを人為的に変化させることによって、植物の栄養吸収能力を高めたり、栄養をあまり必要としない作物の開発につながる可能性があります。
今回明らかになったホウ素濃度に応じたリボソームの挙動の模式図
発表内容
東京大学大学院農学生命科学研究科の藤原 徹教授、理化学研究所生命機能科学研究センター伊藤 拓宏チームリーダー、および理化学研究所開拓研究本部 岩崎信太郎主任研究員、東北大学大学院生命科学研究科 横山武司助教らの共同研究グループは、構造学的、生化学的解析から、AUG-stop配列を介した植物のホウ素に応答した翻訳開始制御の新しい仕組みを明らかにしました。この仕組みを通じて植物は土壌からの栄養の吸収を制御し、正常な生育を維持しています。
植物の成長や作物生産には土壌の栄養が不可欠ですが、土壌の栄養濃度は植物にとって適切とは限りません。不適切な栄養条件に置かれた植物は不足する栄養をより良く吸収するために、栄養の輸送体の蓄積量を増加させたりします。植物が輸送体の蓄積量を栄養条件に応じて増やすためには、栄養条件を感知する必要があります。これまでの著者らの研究で植物の栄養素の一つホウ素を吸収するための輸送体の蓄積を増やすために、植物は細胞質のホウ素濃度を感知して、特定のタンパク質の合成量を変化させることがわかっていましたが、ホウ素がどのように感知されるのかについては分かっていませんでした。今回、ホウ素を与えたリボソームの分子構造解析や生化学的な解析を通じて、ホウ素がリボソームの挙動を変化させ、それによって輸送体タンパク質の合成量を制御していることが明らかになりました。
研究グループではホウ素を土壌から吸収するのに働くホウ酸輸送体NIP5;1を2006年に見出しました。NIP5;1をコードするmRNAの蓄積は、環境中のホウ素濃度によって厳密に制御されていることを2011年に見出し、さらにこの制御にはNIP5;1の非翻訳領域に存在する開始コドンと終止コドンのみからなる、最小のuORF(AUG-UAA 以下AUG-stopと呼びます)が必要であること、細胞内のホウ素濃度が高くなるところの配列上でリボソーム(注1) が停滞し、それに伴ってmRNAが分解することを2016年に明らかにしてきました(https://www.a.u-tokyo.ac.jp/topics/2016/20161020.html)。しかし、細胞質のホウ素濃度が高まることが、どのように翻訳過程に影響を及ぼすかについての分子メカニズムは明らかではありませんでした。
真核生物のリボソームは、40Sと60Sという2種類の大きさの異なるサブユニットから構成されています。リボソームのタンパク質合成は翻訳開始、伸長、終結の3つの主要段階によって行われます。翻訳開始では、複数の翻訳開始因子が結合したmRNAの5’キャップ(注2)に、開始tRNA(注3)と結合した40Sサブユニットが結合し(48S複合体)、この複合体が5’キャップから下流に移動し、最初の開始コドン(AUG)を見つけ、60Sサブユニットをリクルートすることで、80S開始複合体を形成し、翻訳が始まります。その後、翻訳が進み、終結コドン(UAA, UAG, UGA)までたどり着くと、翻訳終結因子(eRF1, 注4))がリボソームに結合し、加水分解を引き起こして、ペプチドをtRNAから解離させます。その後、リボソームはmRNAから解離し、リサイクルされます。
真核生物では、5’キャップから開始コドンまでの領域は5′-非翻訳領域(5′-UTR)と呼ばれ、43S複合体が開始コドンを探して移動していきます。この領域ではしばしば短いORF(uORF)が存在し、翻訳される場合があります。uORFで翻訳が完結し、下流のORFは翻訳されない場合もありますが、uORFで翻訳が完結しても再びリボソームが下流へ移動し、次の開始コドンで翻訳を再開する場合があります。この現象はreinitiationと呼ばれ、60Sサブユニットのみが解離し、40サブユニットがmRNA上に残って、下流へ再び移動を始め、開始コドンに到達し、60Sサブユニットをリクルートして、翻訳を再開する仕組みと考えられています。また、uORF上でリボソームが配列や小分子化合物特異的に停滞し、翻訳が途中で止まることがあります。この場合、別の因子が停滞しているリボソームと結合し、mRNAの分解およびリボソームが解離します。
今回、私たちは、NIP5;1の非翻訳領域に存在するAUG-stop配列上で、ホウ素濃度依存的にリボソームがどのように停滞しているのかをクライオ電子顕微鏡(Cryo-EM, 注5)を用いた構造解析によって、また、AUG-stop上で停滞したリボソームが、どのように下流のORFの翻訳を再開しているのかを生化学的解析によって明らかにすることを目的としました。
まず、コムギ胚芽無細胞タンパク質合成系(注6)を用いて、タンパク質を合成させ、高ホウ素条件/ホウ素なし条件で、AUG-stop上で停滞しているリボソームを精製し、Cryo-EMを用いて、リボソームの立体構造の解析を行いました。その結果、リボソームには開始tRNAと翻訳終結因子であるeRF1が結合し ていることが分かりました(図1A)。eRF1は、ホウ素によってその構造が変化しており、ホウ素非存在下では、Cryo-EMの密度が弱く(図1B)、eRF1はリボソーム内で固定されず、動いている状態にあることがわかり、一方、ホウ素存在下では、eRF1が固定され、eRF1のGGQモチーフ(注7)と開始tRNAのCCA end(注8)が結合した状態で存在していることが明らかとなりました(図1C)。この状態は、ちょうど翻訳終結反応の過程の、ペプチドの加水分解が起こる状態と一致していました。このことから、ホウ素はeRF1を固定するために働いていることがわかりました。
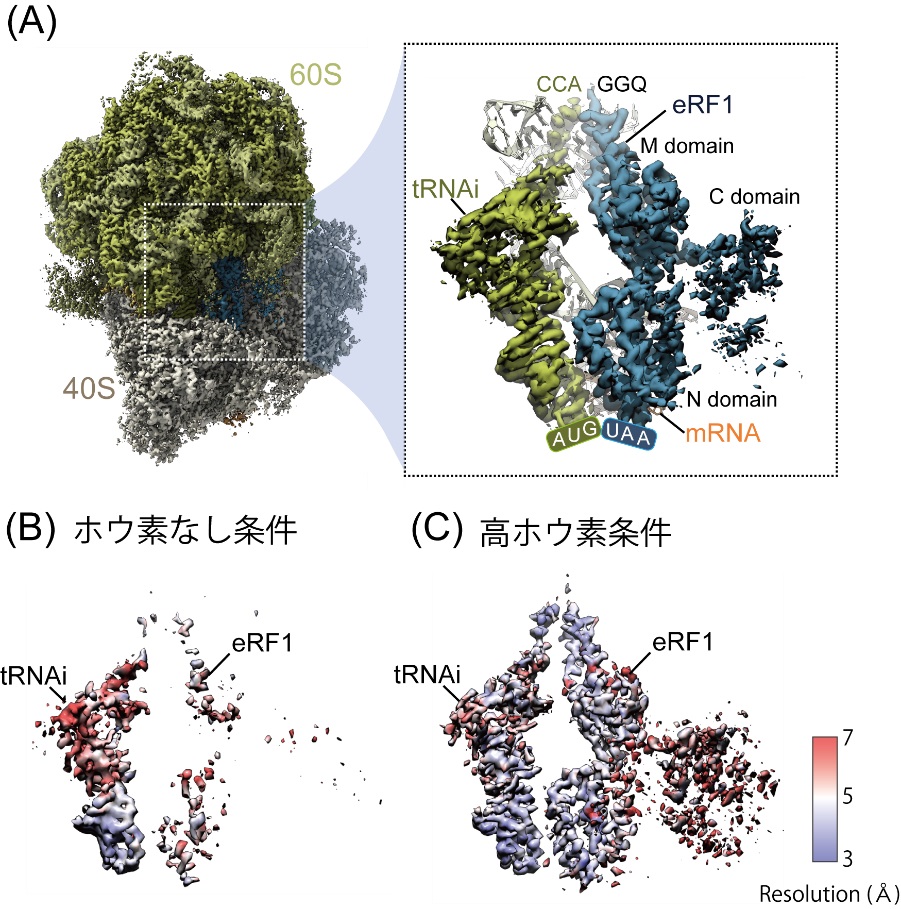
図1:Cryo-EMによるAUG-stop上で停滞している80Sリボソームの立体構造。
(A) 80Sに開始tRNA(tRNAi)とeRF1が結合している。(B)(C)開始tRNAとeRF1だけを抽出している。ホウ素がない条件では、eRF1のCryo-EM密度が弱く、形がほとんど見えない(B)。高ホウ素条件では、eRF1がはっきりと見える(C)。色の変化は局所的な分解能を示し、より青い部分は分解能が高く、より赤い部分は分解能が低い。
次にAUG-stop上でリボソームが停滞した後、どのようにして下流のORFが翻訳されるのかを明らかにするため、AUG-stopからmain ORFまでの領域(inter ORF)のリボソームがどのような状態(80Sか40Sか)で存在しているのかを、検出することにしました。Ribo-seq(80S が結合している領域を検出する方法、注9)とTCP-seq(40Sが結合している領域を検出する方法、注10)を組み合わせて解析した結果、ホウ素非存在下において、inter ORFでは、それより上流域と比較すると、40Sの存在比が少なくなり(図2A)、逆に80Sが多く存在していることが分かりました(図2B)。80Sはinter ORFでは翻訳しておらず、移動しているだけであること、ORFに到達すると、翻訳が再開していることもわかりました。つまり、inter ORFでは、80Sが翻訳せず移動している、「80S sliding」が起こっていることがわかりました。これまで知られている60Sが解離し、40Sが再び下流へ移動する、reinitiationとは異なるシステムでした。また、80S slidingが起こることは知られていましたが、今回の結果は、80S slidingが発現制御機構に機能的に関与していることを示した初めての例となりました。
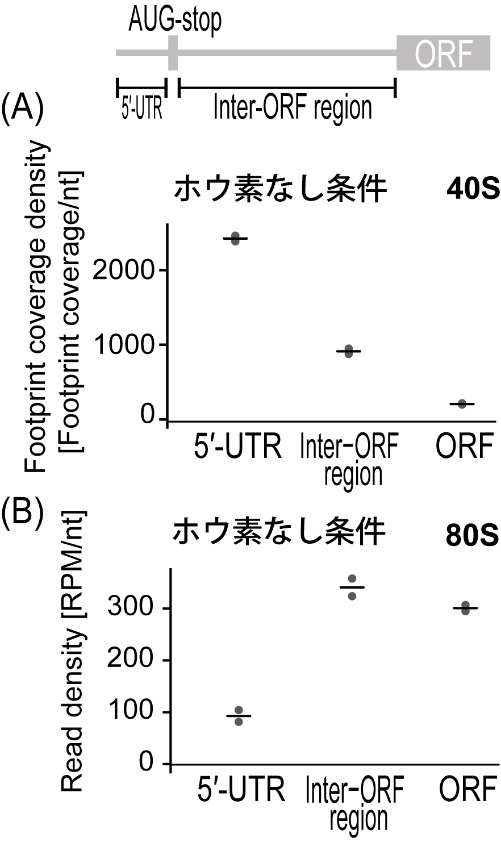
図2:NIP5;1上のリボソームの挙動。
(A)ホウ素非存在下の時のTCP-seqによるそれぞれの領域での40Sの存在比を示す(B)Ribo-seqによるそれぞれの領域での80Sの存在比を示す。
さらに、生化学実験から、高濃度のホウ素が、開始tRNAのメチオニンの加水分解を促進する働きがあることが分かり、ホウ素がeRF1を固定することによって、ゆっくりと加水分解が進行し、翻訳終結が促されていることが推察されました。ゆっくりと進む加水分解が、見かけ上長くリボソームが停滞しているように見えたのだと考えられます。
これらの実験は、NIP5;1のホウ素依存的な翻訳発現制御のメカニズムを明らかにしただけでなく、80S slidingが機能的に起こっている、新しい翻訳開始制御を初めて明らかにしたものです(図3)。
このような精密な制御を通じて、植物は土壌からの栄養吸収を最適化していると考えられます。この制御を人為的にチューニングすることができれば、植物の栄養吸収や生育の改善につながる可能性が考えられます。無機栄養は食糧生産に不可欠ですが、肥料は有限な資源を使って製造されており、肥料の利用は環境にも悪影響を及ぼす側面があります。そのため、肥料をなるべく使わずに生産を維持する農業の実現が求められています。作物生産に肥料が必要な一つの原因は植物の栄養吸収能力に限界があるためです。タンパク質の合成は全ての生物に不可欠なプロセスですが、今回の発表は植物ではタンパク質の合成段階で無機栄養を感知して合成を制御することを明らかにするものです。今回の発見はこれまでに無い植物での栄養の感知とそれに基づいたタンパク質の合成量制御を人為的に改変し、より栄養欠乏に感度の高い作物の作出を通じて、肥料を減らしても食糧生産を維持する技術開発につながる可能性があります。

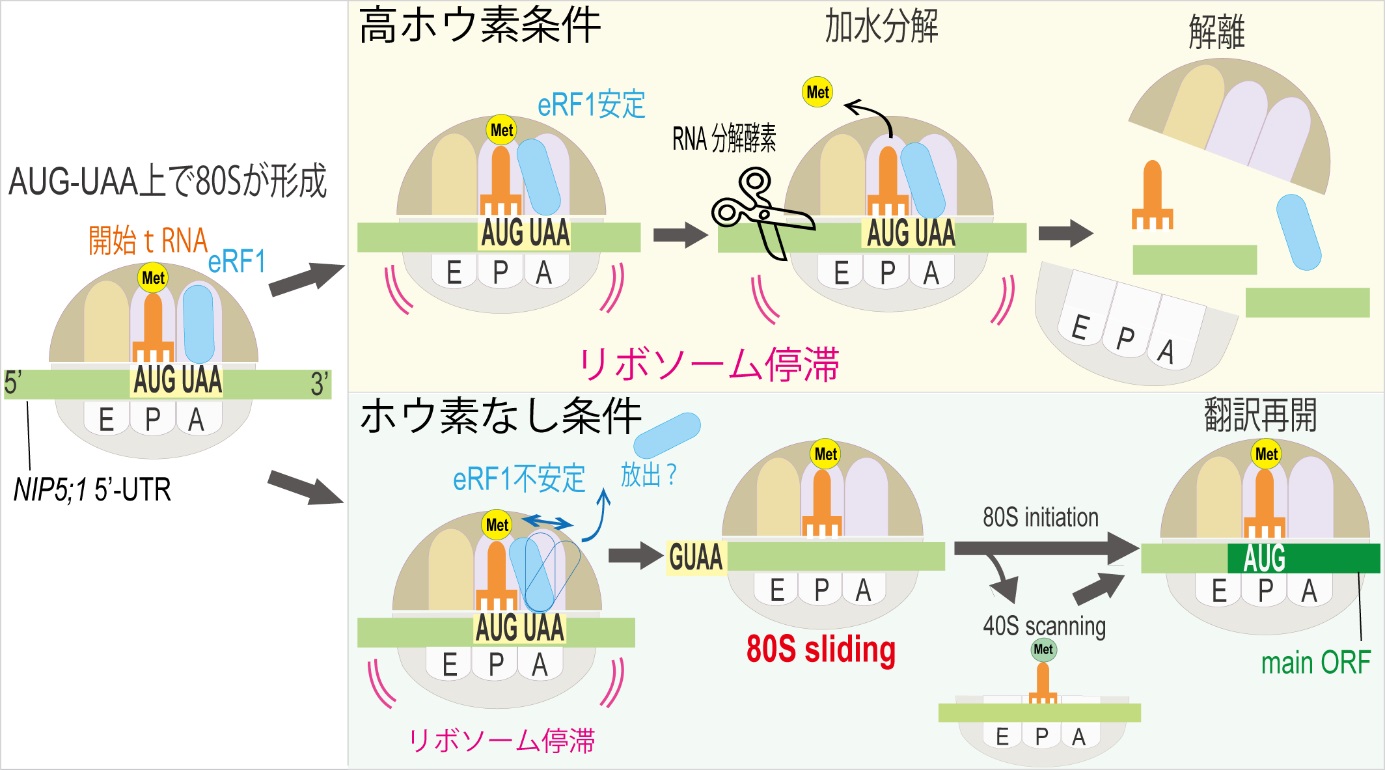
図3:NIP5;1のホウ素依存的な翻訳開始制御機構の模式図。
AUG-UAA配列上で80Sが形成され、eRF1がA部位に結合する。高ホウ素条件ではeRF1がAサイトで安定化しており、これがtRNAと結合しメチオニンの解離を促し、終結反応が起こる。終結反応の間に、mRNAが分解され、翻訳終結後、リボソームが解離する。一方、ホウ素なし条件では、eRF1が不安定で、 メチオニンの解離が起こらず、翻訳終結が起きない。その後、80S slidingが起こり、下流のORFの開始コドンをみつけて、翻訳が再開される。
発表者
東京大学大学院農学生命科学研究科 応用生命化学専攻
田中 真幸(特任助教:研究当時)
反田 直之(助教)
藤原 徹 教授
理化学研究所
開拓研究本部 岩崎RNAシステム生化学研究室
斉藤 大寛(大学院リサーチ・アソシエイト/研修生; 東京大学大学院新領域創成科学研究科メディカル情報生命専攻博士課程)
岩崎 信太郎(主任研究員)
生命機能科学研究センター 翻訳構造解析研究チーム
西本 まどか(テクニカルスタッフI)
伊藤 拓宏(チームリーダー)
生命機能科学研究センター タンパク質機能・構造研究チーム
津田 健吾(リサーチアソシエート:研究当時)
重松 秀樹(上級研究員:研究当時)(現 公益財団法人高輝度光科学研究センター構造生物学推進室 研究員)
白水 美香子(チームリーダー)
東北大学大学院生命科学研究科 分子化学生物学専攻
横山 武司(助教; 理化学研究所生命機能科学研究センタータンパク質機能・構造研究チーム 研究員:研究当時)
発表雑誌
- 雑誌
- Nature chemical biology (2023)
- 題名
- Boric acid intercepts 80S ribosome migration from AUG-stop by stabilizing eRF1 at the A site
- 著者
- Mayuki Tanaka, Takeshi Yokoyama, Hironori Saito, Madoka Nishimoto, Kengo
Tsuda, Naoyuki Sotta, Hideki Shigematsu, Mikako Shirouzu, Shintaro Iwasaki*, Takuhiro Ito*, and Toru Fujiwara* *責任著者 - DOI
- 10.1038/s41589-023-01513-0
- URL
- https://www.nature.com/articles/s41589-023-01513-0
研究助成
本研究は、科研費「基盤研究(S) 植物の栄養感知機構の解明と栄養応答統御(研究代表者:藤原徹)(課題番号:19H05637)」、「学術変革領域(A) 周囲環境応答としての植物成長特性の力学的最適化の柔軟性(研究代表者:藤原徹)(課題番号:18H05490)」、「基盤研究(C) AUG-UAAを介したリボソーム停滞のホウ素栄養制御機構(研究代表者:田中真幸)(課題番号:18K06278)」、「基盤研究(B) 複合体間の動的な相互作用による翻訳制御の構造基盤(研究代表者:伊藤拓宏)(課題番号:19H03172)」、「学術変革領域(A) 非ドメイン型バイオポリマーの立体構造・相互作用解析(研究代表者:伊藤拓宏)(課題番号:21H05281)」、「基盤研究(B) ウイルス感染時に誘導されるRNAサイレンシング活性化機構の解明(研究代表者:竹田 篤史、研究分担者:岩崎信太郎)(課題番号:19H02959)」、「学術変革領域(B) 新規Disome-Seq法: パラメトリックなリボソーム渋滞の網羅的探索(研究代表者:岩崎信太郎)(課題番号:20H05784)」、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)「統合的構造解析に向けた高難度複合体の生産支援と高度化(研究代表者:白水美香子)(課題番号:JP20am0101082)」、革新的先端研究開発支援事業(AMED-CREST)「プロテオスタシスの理解と革新的医療の創出」研究開発領域(研究開発総括:永田和宏)における研究開発課題「神経変性疾患におけるアグリゲーションと翻訳の陰陽(研究開発代表者:岩崎信太郎、研究開発分担者:伊藤拓宏)(課題番号:JP23gm1410001)」、理化学研究所独創的研究課題「細胞内環境の生物学」、科学技術振興機構(JST)戦略的創造研究推進事業 さきがけ 細胞の動的高次構造体「リボソームの動的分子構造と細胞内分布の統合的理解(研究代表者:横山武司)(課題番号:JPMJPR20EG)の支援により実施されました。また、クライオ電子顕微鏡測定は、東北大学未来型医療創成センター(INGEM)(AMED研究助成金JP20am0101095)及び理化学研究所 生命機能科学研究センター(BDR)のクライオ電子顕微鏡施設を利用しました。
用語解説
注1 リボソーム
mRNAの遺伝情報をタンパク質へ翻訳する装置。
注2 5’キャップ
真核生物のmRNAの5’末端にある化学的修飾構造。この構造上で翻訳開始複合体が形成される。
注3 開始tRNA
翻訳開始に必須なtRNAでメチオニンと結合した後40Sに結合する。
注4 真核生物翻訳終結因子 1(eRF1)
翻訳の終結に必要な因子。終止コドンがリボソームにくるとeRF1-3複合体が終止コドンを認識し結合する。その後、eRF1が合成したペプチド鎖の解離を引き起こす。
注5 クライオ電子顕微鏡
生体試料等を急速凍結し、凍結状態のまま、透過型電子線を用いて低温で観察する手法。生体試料を生理条件に近い環境で観察することができる。得られた2次元の粒子像から画像処理でその立体構造を再構成することができる。
注6 コムギ胚芽無細胞タンパク質合成系
小麦の胚芽の抽出液を用いて、目的タンパク質の遺伝情報を持つmRNAを添加し、試験管内でタンパク質を合成する実験系。
注7 GGQモチーフ
eRF1が持つモチーフの一つ。リボソームのPeptidyl Transferase Centerと呼ばれる場所と相互作用することで、tRNA からのペプチド鎖の解離を引き起こす。
注8 CCA end
tRNA の3’末端の配列。アミノ酸を受容するのに必要であるが、その他、ペプチジル転移反応に必須な配列。
注9 リボソームプロファイリング
80Sリボソームが結合する mRNA 配列を網羅的に同定する手法。
注10 Translation complex profile (TCP-seq)
走査リボソーム(40Sなど)が結合する mRNA 配列を網羅的に同定する手法。
問い合わせ先
〈研究に関する問合せ〉
東京大学大学院農学生命科学研究科 応用生命化学専攻
教授 藤原 徹(ふじわら とおる)
理化学研究所生命機能科学研究センター 翻訳構造解析研究チーム
チームリーダー 伊藤 拓宏 (いとう たくひろ)
理化学研究所開拓研究本部 岩崎RNAシステム生化学研究室
主任研究員 岩崎 信太郎(いわさき しんたろう)
東北大学大学院生命科学研究科・分子化学生物学専攻・応用生命分子解析分野
助教 横山 武司(よこやま たけし)
〈報道に関する問合せ〉
東京大学大学院農学生命科学研究科・農学部
事務部 総務課総務チーム 総務・広報情報担当(広報情報担当)
理化学研究所 広報室 報道担当
東北大学大学院生命科学研究科 広報室