ヒト・非ヒト霊長類のゲノムなどの高品質データを提供
2020-03-25 理化学研究所,東京都医学総合研究所,滋賀医科大学,実験動物中央研究所,慶應義塾大学,医薬基盤・健康・栄養研究所,日本医療研究開発機構
理化学研究所(理研)予防医療・診断技術開発プログラムの河合純副プログラムディレクターらの共同研究グループ※は、ヒト、カニクイザル、マーモセットの霊長類のゲノムと遺伝子モデル、遺伝子発現の情報を含むオミックス情報[1]の統合データベース「D3G(Database for Drug Development based on Genome and RNA)」を公開しました。
本データベースを利用することで、「核酸医薬[2]」の創薬研究における効果的な塩基配列の選択や有効性の評価、安全性の予測・解釈が容易になり、創薬に携わる研究者(アカデミア・企業)の業務が大きく推進され、創薬における新たなイノベーションにつながると期待できます。
アンチセンス医薬[3]やsiRNA医薬[4]に代表される核酸医薬は、種特異性が高いため、ヒトと遺伝的な類似度が高い非ヒト霊長類を用いた評価が有用と考えられます。しかし、これまで非ヒト霊長類の遺伝子情報の整備は限定的であるとともに体系的に整理されておらず、創薬のボトルネックになっていました。
今回、共同研究グループは、創薬研究において利用される非ヒト霊長類モデル動物であるカニクイザルおよびマーモセットのゲノムDNAとRNAを独自の技術を駆使して解析することで、高品質かつ総合的なデータセットを構築しました。本データセットは全ゲノムDNA配列、遺伝子モデル、毒性評価試験の対象とされる全組織の遺伝子発現の情報を含み、塩基配列を検索するツールや手順とともにデータベースD3Gとして整備されました。
本データベースのα版(データコンテンツの限定版)は、2019年夏より関係コミュニティ(研究や創薬を目的とした組織や団体)に広く利用されていますが、今般、最新のデータコンテンツを掲載した正式版が2020年3月25日に「D3G(Database for Drug Development based on Genome and RNA)」ウェブページ から一般に公開されます。
また本データベースのデータは、オンプレミス運用を可能にする高速塩基配列検索ソフトウェア「GGGenomeパッケージ版(創薬パック)」(株式会社レトリバ)にも収録されています。
- ※共同研究グループ
-
- 理化学研究所
-
- 予防医療・診断技術開発プログラム
- 副プログラムディレクター 河合 純 (かわい じゅん)
- 生命医科学研究センター
- 予防医療・ゲノミックス応用開発ユニット
- ユニットリーダー 川路 英哉 (かわじ ひでや)
(東京都医学総合研究所 副参事研究員) - 特別研究員 廣瀬 直毅 (ひろせ なおき)
- テクニカルスタッフ 佐野 浩美 (さの ひろみ)
- 理研-IFOMがんゲノミクス連携研究チーム
- チームリーダー 村川 泰裕 (むらかわ やすひろ)
- 応用ゲノム解析技術研究チーム
- チームリーダー 岡崎 康司 (おかざき やすし)
- 客員主管研究員 八木 研 (やぎ けん)
- 技師 山本 由美子(やまもと ゆみこ)
- 分子配列比較解析チーム
- チームリーダー 工樂 樹洋 (くらく しげひろ)
- 技師 門田 満隆 (かどた みつたか)
- 技師 西村 理 (にしむら おさむ)
- 滋賀医科大学 動物生命科学研究センター 幹細胞・疾患モデル研究部門
- 教授 依馬 正次 (えま まさつぐ)
- 客員講師 清田 弥寿成(せいた やすなり)
- 特任助教 中家 雅隆 (なかや まさたか)
- 特任准教授 築山 智之 (つきやま ともゆき)
- 特任助教 岡村 永一 (おかむら えいいち)
- 特任助教 武藤 真長 (むとう まさなが)
- 実験動物中央研究所 マーモセット医学生物学研究部
- 部長 佐々木 えりか(ささき えりか)
- 室長 井上 貴史 (いのうえ たかし)
- 研究員 峰重 隆幸 (みねしげ たかゆき)
- 研究員 圦本 晃海 (ゆりもと てるみ)
- 研究員 渡部 聡朗 (わたなべ としあき)
- 技術員 李 佳穎 (リー・ジャーイン)
- 慶應義塾大学 理工学部 生命情報学科
- 教授 榊原 康文(さかきばら やすぶみ)
- 研究員 ワサンタン・ジャヤクマル
(Vasanthan Jayakumar) - 医薬基盤・健康・栄養研究所 霊長類医科学研究センター
- センター長 保富 康宏 (やすとみ やすひろ)
- 主任研究員 山海 直 (さんかい ただし)
- 情報・システム研究機構 データサイエンス共同利用基盤施設
-
- ライフサイエンス統合データベースセンター
- 特任助教 内藤 雄樹 (ないとう ゆうき)
背景
医薬品の開発では、期待した効果を発揮すること、副作用を可能な限り回避することの2点が重要です。「核酸医薬」は疾患の原因遺伝子を特定できれば、当該遺伝子の転写産物(RNA)と相補的に結合する核酸の配列を即座に設計できるという利点があることから、次世代の医薬品として注目を集めています。
設計する核酸の配列(核酸医薬の候補品)の効果や副作用の可能性を推し測るためには、ヒトや非臨床試験で用いられる動物のDNAおよびRNAの情報が必須であり、さらには、臓器ごとのRNAの配列や発現量に関する情報も重要です。現在、最も開発が進んでいる核酸医薬は、アンチセンス医薬です。アンチセンス医薬はmRNA[5](伝令RNA)のみならず、mRNAの前駆体であるpre-mRNA[5]にも作用することから、核酸の配列についてはpre-mRNAの情報も重要です。
医薬品の開発では、非臨床試験において有効性と安全性が評価されますが、アンチセンス医薬やsiRNA医薬はRNAと結合して作用するため、その作用機序を考慮した評価と解釈が必要です。具体的には、ヒトRNAに対してデザインした核酸医薬の塩基配列が動物のRNAにも相補的に結合するか(薬理作用を発揮するか)否かにより、動物を用いた有効性の評価が可能か、あるいはオンターゲット毒性[6]の評価が可能かを判断します。
オフターゲット効果に起因する狭義のオフターゲット毒性[6]については、ゲノム配列が異なる動物では評価ができません。したがって、ヒトRNAのデータベースを用いたin silico[7]とヒト細胞を用いたin vitro解析(遺伝子発現変動解析)を組み合わせた評価法などによって、オフターゲット遺伝子(非標的遺伝子)を特定し、毒性発現の可能性を考察することになります。ここで、毒性の発現が懸念される特定のオフターゲット遺伝子が特定され、かつ、当該遺伝子のカニクイザルやマーモセットのオルソログ(直系遺伝子)[8]に対しても核酸医薬が相補的に結合(作用)するのであれば、当該オフターゲット遺伝子については、カニクイザルやマーモセットを用いた安全性試験で毒性評価できることになります。
以上のように、核酸医薬の非臨床試験における有効性と安全性の評価には、ヒトならびに動物のRNA配列(mRNA およびpre-mRNA)の情報が重要であり、特に、進化的類縁性と遺伝的類似度の高い非ヒト霊長類については必須です。しかし、その整備はデータ測定という観点からも、利用できるデータベースという観点からも不十分であり、コミュニティ(研究や創薬を目的とした組織や団体)の大きな課題でした。DDBJ(日本DNAデータバンク)といった公共レポジトリ[9]は、あらゆる遺伝子配列の一次データを収集し保管することを目的として運営されていますが、コミュニティが速やかに利用できるデータセットを構成するためには、データごとの精度管理や適切な計算処理、利用形態を想定した編纂などのプロセスが必要となります。
研究手法と成果
共同研究グループは、コミュニティ(研究や創薬を目的とした組織や団体)が必要とするデータベースを開発するために、多くの製薬企業、規制関係者、学会の協力を得て、求められるデータコンテンツを明らかにし、カニクイザルとコモンマーモセットに関するDNAゲノムとRNAのデータを取得しました(表1)。

表1 本研究で新たに開発したゲノムDNAとRNAのデータ
取得したデータセットには、高品質のゲノム配列と毒性評価試験の対象とされる全組織の遺伝子発現情報という際立った特徴があります。
ゲノム配列については、一分子リアルタイムシーケンス[10]によるロングリード(長いDNA塩基配列)と、ゲノムDNAの核内3D構造に基づいた配列再構築[11]により、染色体に相当する長さを持つDNA塩基配列を、すでに知られている染色体の本数だけ得ることができました。つまり、本研究で取得したデータのみを用いて、染色体レベルでのゲノム配列構築に成功しました。この新しいゲノム配列に含まれるギャップ領域(塩基配列が読み取れていない領域)の個数は、カニクイザルやコモンマーモセット研究においてこれまで用いられてきたゲノム配列に比べ、百分の一に減らすことができました。これは、これまで集中的に解析が進められてきたヒトやマウスのゲノム配列に迫る品質のゲノム配列です。さらに、2生物種のそれぞれから、創薬の毒性評価試験の対象となる約50種類の全身の臓器を、RNAの分解を最小限に抑えながら採取し、RNA-seq[12]およびCAGE[13]のデータを取得することで、カニクイザルで18,333個、マーモセットで18,629個という、これもヒトやマウスと同等の完成度の遺伝子モデルを構築できました(表2)。これらは、核酸医薬の開発に重要な遺伝子塩基配列の情報と組織ごとの遺伝子発現の情報を提供します。

表2 D3Gに採録されているゲノムと遺伝子モデル
図1に示すように、全てのデータは、データベース「D3G(Database for Drug Development based on Genome and RNA) 」に統合され、ブラウザで視覚的に表示することができます。また、全てのデータセットを利用者のローカル環境へダウンロードできます。
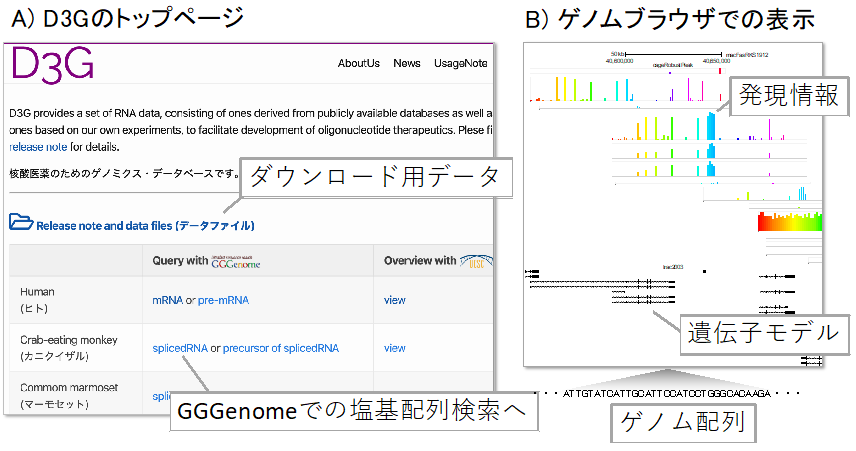 図1 D3Gのスナップショット
図1 D3Gのスナップショット
A)D3Gのトップページ。
B)D3Gで利用できる機能。
さらに、核酸医薬の開発において重要なオンターゲット遺伝子およびオフターゲット遺伝子の候補を容易に効果的に検索できるよう、短鎖配列の検索ツールGGGenomeをスムーズに利用できるリンクを設定するとともに(図2A)、ツールの利用手順を例示しました(図2B)。
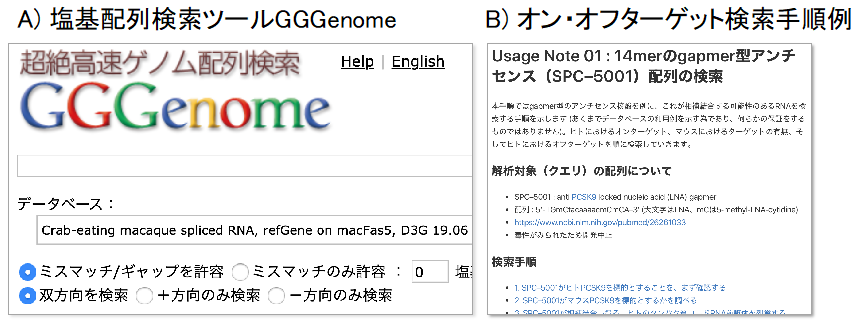 図2 D3Gの利用例
図2 D3Gの利用例
A) 相同性検索ツールGGGenome。
B)オン・オフターゲット探索の手順例。
今後の期待
本データベースは、ヒト・非ヒト霊長類およびマウスについて精度の高いオミックス情報を整理、統合し提供するものです。本成果は、核酸医薬のオフターゲット効果の予測・評価などに有用であり、国立医薬品食品衛生研究所などとの共同研究として日本分子生物学会誌『Genes to Cells』に発表した「RNA分解型アンチセンスのオフターゲット効果の評価法」に関する論文注1)においても、本データベースにおけるヒトpre-mRNAのデータセットが活用されています。
このように本成果は創薬に大きく貢献するものですが、それにとどまらず医科学や生物学などの基礎研究の基盤データを提供するものです。今後、データを充実させるとともに、疾患や遺伝子機能に関する情報を連携させることが期待されます。
注1)Tokuyuki Yoshida, Yuki Naito, Hidenori Yasuhara, Kiyomi Sasaki, Hideya Kawaji, Jun Kawai, Mikihiko Naito, Haruhiro Okuda, Satoshi Obika, Takao Inoue. Evaluation of off-target effects of gapmer antisense oligonucleotides using human cells. Genes to Cells, Vol.24, No.12, 827-835 (2019)
補足説明
- [1] オミックス情報
- 生命活動をになう生体分子の網羅的な情報の集合。DNAやRNAの塩基配列、遺伝子の発現情報、タンパク質の発現や構造などさまざまなものがある。
- [2] 核酸医薬
- 一般に、「核酸あるいは修飾核酸が十数~数十塩基連結したオリゴ核酸で構成され、タンパク質に翻訳されることなく直接生体に作用するもので、化学合成により製造される医薬品」を指す。核酸医薬は、RNAを標的とするものとタンパク質を標的とするものに大別され、前者についてはアンチセンスとsiRNAが代表的である。タンパク質を標的とする低分子医薬や抗体医薬による創薬シーズの枯渇が危惧されるなか、核酸医薬はRNAを標的とした創薬が可能であることなどから、次世代のモダリティとして注目を集めている。2020年3月までに、日米欧で11の核酸医薬が上市されている。
- [3] アンチセンス医薬
- 標的RNAと相補的に結合することで機能する核酸医薬で、修飾核酸を含む1本鎖DNAで構成される。RNA分解やスプライシング制御などの作用機序で有効性を発揮する。
- [4] siRNA医薬
- 標的mRNAと相補的に結合し、RNA干渉(RNAi)の原理でmRNA切断する核酸医薬。20塩基長程度の2本鎖RNAで構成される。
- [5] mRNA、pre-mRNA
- 「mRNA」は、ゲノムDNAの遺伝子領域鋳型に転写されるRNAのことであり、タンパク質に翻訳され得る塩基配列の情報を持つ。「pre-mRNA」は、スプライシングなどのさまざまな加工を受けて成熟した mRNA になる前の RNA 分子(mRNA前駆体)。 一般に真核生物では、タンパク質をコードする遺伝子の多くがイントロン(非コード領域)を持つが、スプライシングによりpre-mRNAから除去される。mRNAとpre-mRNAは核酸医薬の主要な標的となる分子である。
- [6] オンターゲット毒性、オフターゲット毒性
- 「オンターゲット毒性」は薬剤が標的分子に作用し、過大な薬理作用が起こった結果生じる毒性であり、一方、「オフターゲット毒性」は薬剤が本来の標的分子とは異なる分子に作用した結果生じる毒性を指す。RNAを標的とする核酸医薬については、作用機構の観点からオフターゲット毒性が大きく二つに分類される。すなわち、核酸医薬が標的RNA以外のRNAに作用した結果生じる毒性(いわゆるオフターゲット効果に起因する毒性)は「狭義のオフターゲット毒性」と呼ばれ、核酸医薬がRNA以外の生体成分(主にタンパク質と想定されている)に作用した結果生じる毒性は「広義のオフターゲット毒性」と呼ばれる。ヒト・非ヒト霊長類などのRNAデータベースは、核酸医薬のオンターゲット毒性ならびに狭義のオフターゲット毒性の予測・評価・解釈に有用である。
- [7] in silico
- 「コンピュータを用いて」を意味する。in vivo (生体内で)や in vitro (試験管内で)などに準じて作られた用語。
- [8] オルソログ(直系遺伝子)
- 生物種を越えて、共通祖先から種分岐を経て保持された、互いに類似した配列をもつ遺伝子のこと。
- [9] レポジトリ
- データの一元的な貯蔵庫。
- [10] 一分子リアルタイムシーケンス
- 一分子のDNAポリメラーゼによるDNA合成をリアルタイムに観察することで、核酸の塩基配列を決定する手法。長い塩基配列を読み取れる特徴を持つ。
- [11] ゲノムDNAの核内3D構造に基づいた染色体規模の配列再構築
- Hi-Cスキャフォールディング法を指し、クロマチン相互作用によって細胞の核内で空間的に近接しているゲノム領域を検出する。空間的に近接するゲノム領域は、同じ染色体上の近くに存在することが多い。この性質を生かして、ゲノム配列断片を染色体上の実際の位置関係の順に並べることにより(この過程をスキャフォールディングと呼ぶ)、染色体規模のDNA塩基配列につなぎ合わせることができる。今回は、理研発のiconHi-C(アイコニック)プロトコルに従って実施した。
- [12] RNA-seq
- RNA分子の断片を次世代シーケンサーで大規模に解読する手法。RNAの構造や発現量の解析などに用いられる。
- [13] CAGE
- RNA分子の5’端の30~数十ベースを、次世代シーケンサーで大規模に解読する手法。RNAの転写が開始される位置やその転写量を取得できる。国際プロジェクトFANTOMで利用されている。CAGEはCap-analysis of gene expressionの略。
謝辞
本研究を進める上で多くの方からご協力をいただきました。特に以下の方々にあつく感謝します(敬称略)。
- 国立医薬品食品衛生研究所 井上 貴雄、吉田 徳幸
- 日本製薬工業会 基礎研究部会 渡部 一人、木下 潔、太田 哲也
- 日本核酸医薬学会レギュラトリーサイエンス部会
- 理化学研究所 林崎 良英、ピエロ・カルニンチ、粕川 雄也、ホン・ヂョン・チョウ、デ・ホーン・ミヒル・ヤン・ラウレンス
- 独立行政法人 医薬品医療機器総合機構 毒性領域
- 株式会社レトリバ 河原 一哉、億田 正貴
- 株式会社ビッツ 武藤 勇
- 一般社団法人 予防衛生協会 棟居 佳子
発表者・機関窓口
発表者
理化学研究所 予防医療・診断技術開発プログラム
副プログラムディレクター 河合 純 (かわい じゅん)
機関窓口
理化学研究所 広報室 報道担当
東京都医学総合研究所 事務局 研究推進課
滋賀医科大学 総務企画課 広報係
実験動物中央研究所 広報室
慶應義塾 広報室
国立研究開発法人医薬基盤・健康・栄養研究所
霊長類医科学センター
AMEDについて
国立研究開発法人日本医療研究開発機構(AMED)
基盤研究事業部 バイオバンク課
ゲノム創薬基盤推進研究事業担当