モウセンゴケ科に属するコモウセンゴケ、ハエトリソウ、ムジナモの3種の ゲノム解読により判明
2020-05-15 基礎生物学研究所,総合研究大学院大学,金沢大学,宇都宮大学
食虫植物は葉で小動物を誘引、捕獲、消化、吸収し、栄養としています。このため、他の植物が生育できないような貧栄養地で生育することが可能です。食虫植物は植物の進化の中で、9回独立に進化したと推定されており、世界に約600種が知られています。基礎生物学研究所および総合研究大学院大学の長谷部光泰教授らを中心とした研究グループでは、2017年に壺型の捕虫葉を形成するフクロユキノシタのゲノムを解読し、消化酵素は耐病性遺伝子から進化した可能性が高いことを発表しました。しかし、耐病性遺伝子がどのようにして消化酵素へと進化できたのかは不明でした。今回、ゲルゴ パルファルビ大学院生、長谷部光泰教授らは、金沢大学西山智明助教、宇都宮大学玉田洋介准教授、およびヴュルツブルク大学(ドイツ)ライナー ヘドリッヒ教授などとの共同研究により、フクロユキノシタとは別の科であるモウセンゴケ科に含まれる、コモウセンゴケ、ハエトリソウ、ムジナモの3種のゲノム解読を行いました。その結果、モウセンゴケ科の祖先で全ての遺伝子がゲノム重複によって倍に増え、増えた遺伝子が消化酵素に加え、誘引、捕獲、吸収に関わる機能を進化させた可能性が高いことがわかりました。この成果は米国東部時間2020年5月14日付でCurrent Biology誌に掲載されました。
 図1:本研究でゲノム解読したモウセンゴケ科の3種。モウセンゴケ属コモウセンゴケ、ハエトリソウ属ハエトリソウ、ムジナモ属ムジナモ。
図1:本研究でゲノム解読したモウセンゴケ科の3種。モウセンゴケ属コモウセンゴケ、ハエトリソウ属ハエトリソウ、ムジナモ属ムジナモ。
【研究の背景】
食虫植物は小動物を誘引、捕獲、消化、吸収することで栄養を摂取し、貧栄養地で生育することができます。被子植物の5つの目で独立に食虫性が進化しましたが、特にナデシコ目ではいくつかの異なった食虫植物が進化しました。モウセンゴケ属(全世界に約200種)の種は、繊毛から消化液を含む粘液を出し、繊毛が動いて獲物を捕らえます(図1左)。ドロソフィルム属(ポルトガルなどに1種)とトリフィオフィルム属(シエラレオネなどに1種)の種は、モウセンゴケ属と同じように粘液を出しますが触毛は動きません。ハエトリソウ属(北米にハエトリソウ1種、図1中央)とムジナモ属(世界に点々とムジナモ1種、図1右)は、葉が瞬時に二つ折りになって獲物を挟み込み、葉の表面の分泌腺から消化液を出します。ウツボカズラ属(東南アジアを中心に約170種)は消化液を溜めた袋で獲物を捕らえます。2000年に基礎生物学研究所を中心とした研究グループは、モウセンゴケ属とハエトリソウ、ムジナモの葉緑体の光合成を担うrbcL遺伝子の塩基配列比較を行い、これら3属は、捕虫様式、捕虫葉形態が大きく異なるものの、近縁であることを発見し、同じモウセンゴケ科に含めることが妥当であることを示しました。
その後、2017年に基礎生物学研究所と総合研究大学院大学を中心とした研究グループは、オーストラリア南西部に自生し、袋型の捕虫葉を形成するフクロユキノシタのゲノムを解読し、袋の中の分泌腺から出る消化液に含まれる酵素は、食虫植物以外の植物では病気の原因となるウイルス、細菌、カビなどを分解する酵素を作る遺伝子(耐病性遺伝子)が転用されたものである可能性が高いことを発見しました。
一方で、食虫植物が進化するには、誘引、捕獲、消化、吸収の全てができる必要があります。しかし、これら多くの遺伝子がどうやってほぼ同じ時期に進化できたのかが不明でした。さらに、消化酵素遺伝子など消化や吸収に関わる遺伝子がどうして分泌腺で働くようになったのかも不明でした。
【研究の成果】
本研究では、モウセンゴケ科の3種である、モウセンゴケ属のコモウセンゴケ、ハエトリソウ、ムジナモの3種のゲノムを解読することに成功しました。その結果、モウセンゴケ科の祖先の段階で、ゲノム重複(ゲノムに含まれる遺伝子がすべて重複して倍数体になること)が起きたことがわかりました(図2)。ゲノム重複によって、耐病性遺伝子と同じ遺伝子ができ、もとからある遺伝子で耐病性の機能を保ちつつ、新しくできた遺伝子が消化酵素へと進化した可能性が高いことがわかりました(図3)。そして、ゲノム全体の遺伝子を調べたところ、消化酵素以外でも、誘引、捕獲、消化、吸収に関わるような279の食虫性関連遺伝子群がゲノム重複に伴い、近縁の非食虫植物と較べて増加していることがわかりました(注1)。
一方、3種ともこれまで報告された他の植物と較べ、最も遺伝子数が少ない部類であることもわかりました(注2)。このことは、ゲノム重複で遺伝子数が増えた後、3属が種分化するより前に、遺伝子数が大きく減少したことを示しています(図2)。その理由は、食虫性が進化することで、根からの養分吸収などが不必要となり、そこに関与していた遺伝子が欠失したためだと考えられます。
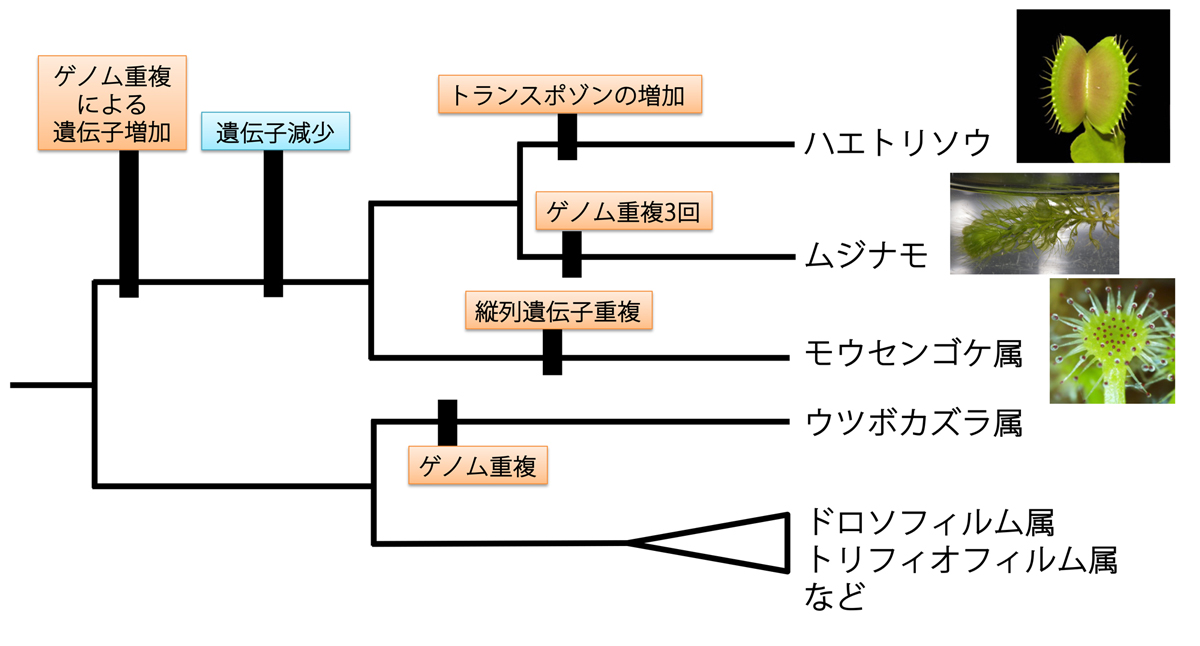 図2:食虫植物の進化過程で起こったゲノムの変化
図2:食虫植物の進化過程で起こったゲノムの変化
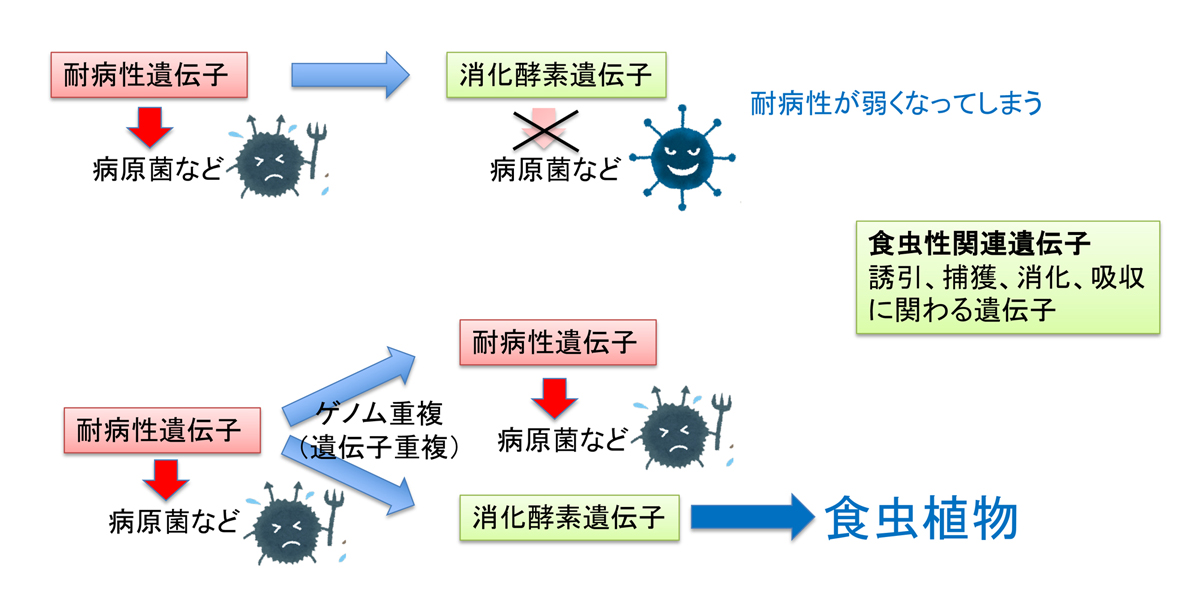 図3:遺伝子重複による食虫性の進化の模式図
図3:遺伝子重複による食虫性の進化の模式図
遺伝子数の急激な増減によって生まれた食虫性を持つ祖先から、コモウセンゴケ、ハエトリソウ、ムジナモがそれぞれ進化する過程でも、ゲノムに変化が見つかりました。コモウセンゴケでは、約400遺伝子に独自の遺伝子重複がおこっていました。ムジナモでは、ゲノム重複が3回おこっていることがわかりました。さらに、ハエトリソウではレトロトランスポゾン(酵素の働きによってゲノム上を動いたり、増幅したりする塩基配列)が大量に増幅し、ゲノムサイズが31.8億塩基対と、コモウセンゴケ(3.23億塩基対)、ムジナモ(5.09億塩基対)よりもずっと大きくなっていました。これらのことから、モウセンゴケ科の祖先で食虫性が進化した後、コモウセンゴケ、ハエトリソウ、ムジナモのそれぞれの系統でゲノムが大きく変化し、その結果として、現在の多様な捕虫葉形態を生み出したのではないかと推定されます。
さらに、ハエトリソウの消化腺で働く遺伝子の制御領域を調べるとその多くでWRKY(ワーキー)転写調節タンパク質の結合領域があることがわかりました。このことは、消化酵素遺伝子などの消化腺で働く遺伝子がWRKY転写調節タンパク質によって一体となって転写制御されている可能性が高いことを示しています。WRKY転写調節因子は普通の植物では病原菌やバクテリアの感染に伴い、病害応答に関わる遺伝子の働きを誘導します。そこで、ゲノム重複などを介して増幅した複数のWRKY転写因子遺伝子の発現している場所を調べたところ、捕虫葉特異的に働いているWRKY転写因子遺伝子(WRKY6とWRKY29)を発見しました。この遺伝子はムジナモやコモウセンゴケのゲノムにも見つかったことから、これらのWRKY転写因子遺伝子が消化酵素遺伝子を消化腺特異的に働かせている因子である可能性が出てきました。
【研究の意義および今後の展開】
本研究でゲノム重複による遺伝子数の一時的な増加と、増えて自由度の上がった(余った)遺伝子が、食虫性という新しい機能を進化させる上で重要だったことがわかりました。特定の遺伝子の重複ではなく、ゲノム重複することで、いろいろな機能を持った遺伝子の自由度が上がり、誘引、捕獲、消化、吸収という食虫性のための多様な機能をほぼ同じ時期に進化させることが可能になったと考えられます。今後は、さらにゲノム解析を進め、食虫性に関わる遺伝子がもともとどのような機能を持っていたのか、またそれらの遺伝子が統合的に機能するために、どのような制御系の進化が必要だったのかを調べていく予定です。
(注1)獲物の誘引に関わる可能性のある遺伝子として、蜜に含まれるテルペノイドや二次代謝産物合成酵素、糖の輸送体の遺伝子がありました。獲物を認識することに関わる可能性のある遺伝子として、膜タンパク質、シグナル伝達系遺伝子、ジャスモン酸の生合成とシグナル伝達に関わる遺伝子がありました。消化に関わる可能性のある遺伝子として、タンパク質分解酵素、核酸分解酵素などの加水分解酵素の遺伝子がありました。そして、吸収に関わる可能性のある遺伝子として、窒素、リン酸、アミノ酸、糖、金属イオンの輸送体の遺伝子がありました。専門的には279オルソグループが見つかったということです。
(注2)コモウセンゴケはゲノム内に、約18,000遺伝子、ムジナモは約25,000遺伝子、ハエトリソウは約21,000遺伝子が見つかりました。非食虫植物である、シロイヌナズナには約28,000遺伝子、イネには約36,000遺伝子がゲノム中に見つかっています。単細胞の緑藻類であるクラミドモナスは約18,000遺伝子、寄生性で多くの遺伝子を失っているネナシカズラの仲間は約20,000遺伝子であることを考慮すると、今回調べた3種の遺伝子数はとても少ないことがわかります。葉から栄養を吸収するようになったためか、根形成や根の機能に関わる遺伝子が減少しています。
【研究グループ】
本研究は、日本の自然科学研究機構基礎生物学研究所、総合研究大学院大学、金沢大学、東海大学、岐阜大学、宇都宮大学、ドイツのヴュルツブルク大学、ハノーファー大学、ライプニッツ植物遺伝学・作物研究所、生物地球科学研究所、チェコ共和国のチェコ科学アカデミー植物研究所、サウジアラビアのキングサウード大学が参加した共同研究です。
【研究サポート】
本研究は、科学研究費補助金 新学術領域研究(22128001、22128002、17H06384、17H06390)等の補助を受けて実施されました。
【論文情報】
掲載誌:Current Biology
掲載日:米国東部時間2020年5月14日 午前11時
論文タイトル:Genomes of the Venus Flytrap and close relatives unveil the roots of plant carnivory
筆者:Gergo Palfalvi(自然科学研究機構 基礎生物学研究所、総合研究大学院大学)*, Thomas Hackl *(ヴュルツブルク大学)、Niklas Terhoeven(ヴュルツブルク大学)、柴田朋子(自然科学研究機構 基礎生物学研究所)、西山智明(金沢大学)、Markus Ankenbrand(ヴュルツブルク大学)、Dirk Becker(ヴュルツブルク大学)、Frank Förster(ヴュルツブルク大学)、Matthias Freund(ヴュルツブルク大学)、Anda Iosip(ヴュルツブルク大学)、Ines Kreuzer(ヴュルツブルク大学)、Franziska Saul(ヴュルツブルク大学)、上田千晴(自然科学研究機構 基礎生物学研究所、総合研究大学院大学)、福島健児(自然科学研究機構 基礎生物学研究所、総合研究大学院大学、ヴュルツブルク大学)、重信秀治(自然科学研究機構 基礎生物学研究所、総合研究大学院大学)、玉田洋介(自然科学研究機構 基礎生物学研究所、総合研究大学院大学、宇都宮大学)、Lubomir Adamec(ヴュルツブルク大学)、星良和(東海大学)、植田邦彦(金沢大学、岐阜大学)、Traud Winkelmann(ハノーファー大学)、Jörg Fuchs(ライプニッツ植物遺伝学・作物研究所)、Ingo Schubert(ライプニッツ植物遺伝学・作物研究所)、Rainer Schwacke(ドイツ生物地球科学研究所)、Khaled Al-Rasheid(ヴュルツブルク大学、キングサウード大学)、#Jörg Schultz(ヴュルツブルク大学)、#長谷部光泰(自然科学研究機構 基礎生物学研究所、総合研究大学院大学)、#Rainer Hedrich(ヴュルツブルク大学)
*:共同第1著者 #:共同責任著者
DOI: https://doi.org/10.1016/j.cub.2020.04.051
【本研究に関するお問い合わせ先】
自然科学研究機構 基礎生物学研究所
総合研究大学院大学 生命科学研究科 基礎生物学専攻(併任)
教授 長谷部光泰
【報道担当】
基礎生物学研究所 広報室
総合研究大学院大学 総合企画課広報社会連携係
金沢大学 総務部 広報室 広報係
宇都宮大学広報・地域連携室