2020-10-06 理化学研究所
理化学研究所(理研)環境資源科学研究センター生産機能研究グループの榊原均グループディレクター、木羽隆敏研究員、統合ゲノム情報研究ユニットの櫻井哲也ユニットリーダー(いずれも研究当時)らの国際共同研究グループは、キビ亜科[1]に属し、C4型光合成[2]を行うモデル植物であるエノコログサ[3](Setaria viridis)の高精度ゲノム解読を行い、有用形質遺伝子の同定に活用できるゲノムリソースを整備しました。
本研究成果は、トウモロコシやソルガム、サトウキビなどキビ亜科植物の有用遺伝子の探索やゲノム育種による品種改良、C4作物[2]の研究を加速すると期待できます。
今回、国際共同研究グループは、アワ(Setaria itarica)の近縁野生種であるエノコログサの参照ゲノム配列を高精度で解読するとともに、598種のエコタイプ[4]のゲノム配列を解析しました。このゲノムリソース情報を用いて、農業上重要な形質である穂からの種子の脱粒性[5]と葉の角度を制御する遺伝子を同定することにも成功しました。
本研究は、科学雑誌『Nature Biotechnology』オンライン版(10月5日付:日本時間10月6日)に掲載されます。

キビ亜科に属する雑草のエノコログサ(Setaria viridis)
背景
トウモロコシ、ソルガム、サトウキビなどキビ亜科に属するC4作物は、農業やバイオマス利用の主力作物です。今後数十年の間に世界が乾燥し、温暖化が進むにつれて、その重要性はさらに高まると考えられています。
現在、C4作物の育種は、大規模な圃場と人力によって行われていますが、比較的ライフサイクルが長く、また作物自体が大型であるため、実験には大変な労力と時間が必要でした。また、これらの作物はC4型光合成を行うため、イネなどのC3型光合成[2]を行う植物とは、光合成機構も異なります。そのため、キビ亜科植物でC4型光合成を行う実用的なモデル植物であるエノコログサのゲノム解読の必要性が叫ばれていました。
研究手法と成果
アワ(Setaria itarica)の近縁野生種で、雑草のエノコログサのゲノムは、これまで標準エコタイプであるA10.1のドラフトシークエンスによるゲノムアセンブリ(v1.0)が公開されていましたが、精度が低く有効活用されていませんでした。そこで国際共同研究グループは、A10.1ゲノムの高精度解析を行い、データの質を飛躍的に向上させたゲノムアセンブリ(v2.0)を新たに作成しました。
次に、北米に分布するエノコログサの598種のエコタイプについてゲノム配列解析を行い、一塩基多型(SNP)[6]を解析しました。エノコログサは人為的にユーラシア大陸から北米に伝わったとされていますが、SNP解析の結果、北米に分布するエノコログサは四つのサブグループに分類され、それらが異なる伝播の歴史を経ていることが分かりました(図1)。
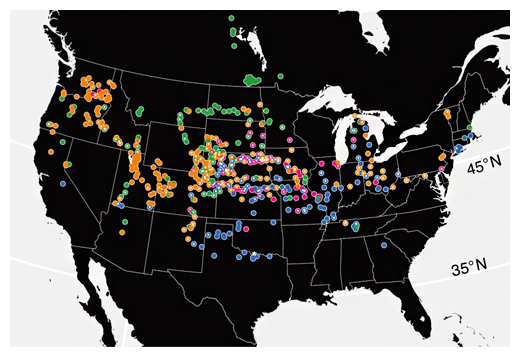
図1 SNP解析で明らかになった北米におけるエノコログササブグループの分布
SNP解析により、北米に分布するエノコログサは、赤・橙・緑・青の四つのサブグループに分類されることが分かった。
さらに、このゲノムリソース情報を活用し、約210種のエコタイプにおける穂からの種子の脱粒性と遺伝子型との関係をゲノムワイド関連解析(GWAS)[7]により調べ、MYB転写因子[8]をコードするSvLes1遺伝子を同定しました。エコタイプ間で高い脱粒性と低い脱粒性に対応する対立遺伝子をそれぞれSvLes1-1とSvLes1-2と名付け、ゲノム編集技術[9]を用いてSvLes1-1遺伝子の機能を破壊したところ、脱粒性が低下することが分かりました(図2)。

図2 エノコログサのSvLes1-1遺伝子機能破壊株
左はSvLes1-1遺伝子を持つ野生株で、脱粒性が高い。右はSvLes1-1遺伝子破壊株。脱粒性が低下したことが分かる。(Kellogg主任研究員、Huang研究員提供)
現在栽培されているアワは脱粒性が低いことが知られていますが、アワのSiLes1遺伝子にはコピア型のレトロトランスポゾン[10](copia38)が挿入されており、その影響でSiLes1遺伝子の機能が失われていることが分かりました。このことだけで、アワの低脱粒性を全て説明できるわけではありませんが、アワの栽培化過程の初期に、このレトロトランスポゾン挿入変異が低脱粒形質として選抜・固定化され、その後の過程で、より強い効果を持つ別の自然変異によって、低脱粒性がさらに強化されたものと考えられます。
さらに、598種のエコタイプの中に葉耳[11]と葉舌[11]が発達せず、葉が直立するものを見いだし、その原因遺伝子としてSvLiguless2(SvLg2)遺伝子を同定しました(図3)。この遺伝子は、トウモロコシで同様の形質を示す原因遺伝子として既に同定されているliguless2遺伝子のオルソログ[12]であることが分かりました。この結果は、エノコログサとトウモロコシの間で、表現型と遺伝子型が対応することを示しています。
なお本研究において、理研の研究チームはエノコログサゲノムの高精度解析のためのシークエンスデータの一部を提供しました。

図3 エノコログサの葉の角度が異なるエコタイプ
A10.1は標準エコタイプで、TB159は、葉耳と葉舌が発達しないために葉が直立するエコタイプ である。((A)はKellogg主任研究員、Huang研究員提供)
今後の期待
本研究により、エノコログサゲノムの高精度解析情報とそれに関連する多様性系統が、遺伝子探索に威力を発揮することが明らかになりました。これらのツールを組み合わせることで、農業上有用な形質や環境応答に関わる新たな遺伝子の発見が可能になると期待できます。
キビ亜科は草本植物の中で2番目に大きいサブファミリーであり、トウモロコシ、ソルガム、サトウキビ、スイッチグラスなど重要な農作物が含まれます。また、C4型光合成のサブタイプもトウモロコシ、ソルガム、サトウキビと同じであることから、C4型光合成研究のモデル植物としても有用です。さらに、これらキビ亜科の作物は、大型で世代時間が4、5カ月以上であるのに対し、エノコログサは小型で世代時間が8~10週間であることから、C4作物研究の迅速化にも貢献すると期待できます。
今回の研究成果は、国際連合が2016年に定めた17項目の「持続可能な開発目標 (SDGs) 」のうち、「2.飢餓をゼロに」と「15.陸の豊かさも守ろう」に大きく貢献するものです。
補足説明
1.キビ亜科
英名Panicoideae、草本植物の中で2番目に大きなサブファミリーであり、トウモロコシ、ソルガム、サトウキビ、スイッチグラスなど重要な農作物が含まれる。主に温帯および熱帯地域に分布している。
2.C4型光合成、C4作物、C3型光合成
「C4型光合成」とは炭酸固定回路であるカルビン-ベンソン回路に加え、二酸化炭素濃縮回路(ハッチ-スラック回路)を持つ光合成のこと。カルビン-ベンソン回路の初期産物が炭素原子三つの3ホスホグリセリン酸であるのに対し、ハッチ-スラック回路のそれは炭素原子四つのオキサロ酢酸であることから、前者を「C3型光合成」、後者をC4型光合成と呼ぶ。トウモロコシ、ソルガム、サトウキビ、スイッチグラスなどC4型光合成を行う作物を「C4作物」と呼ぶ。
3.エノコログサ
学名Setaria viridis、慣用英語名green foxtail。イネ科キビ亜科エノコログサ属の1年生草本で、春から秋にかけて広く見かける雑草。穀物のアワ(学名Setaria itarica、慣用英語名foxtail millet)の原種とされている。C4型光合成を行う。
4.エコタイプ
生物が生育する環境に適応する過程では、遺伝的な分化が、形態や生態の違いなどの表現型の違いが現れる。同じ種であるが、異なる環境に適応し、遺伝的に異なる性質を持つ生物群をエコタイプという。
5.脱粒性
植物の種子が結実後の成熟に伴い、穂から外れ落ちる性質。野生植物では種子の拡散は重要な生息範囲拡大戦略の一つであり、容易に脱粒する。一方で、農業生産では脱粒は収穫量減少の主要な原因となることから、多くの作物の栽培化過程において、脱粒性が喪失されている。
6.一塩基多型(SNP)
個体間で見られるゲノム塩基配列上の一塩基の違いのこと。SNPはSingle Nucleotide Polymorphismの略。
7.ゲノムワイド関連解析(GWAS)
ある集団内での個体間(系統間)での形質の違いと、ゲノム上のDNA塩基配列の違いとの関連を調べることにより、形質の違いと関連するDNAの多型を統計的に検出する解析手法。GWASはGenome-Wide Association Studyの略。
8.MYB転写因子
真核生物に広く保存された転写因子ファミリーの名前。転写因子とは、DNAに特異的に結合するタンパク質を指す。
9.ゲノム編集技術
配列特異的にゲノム配列を改変する技術。CRISPR/CasシステムやTranscription Activator-Like Effector Nucleases(TALEN)などがある。
10.コピア型のレトロトランスポゾン
ショウジョウバエのレトロトランスポゾンであるコピア因子と相同性の高いレトロトランスポゾンを指す。レトロトランスポゾンとは、ゲノム上を移動するDNA配列(トランスポゾン)のうち、移動するときにDNAからRNAに転写された後、再びDNAに逆転写されて別のDNA配列中に侵入するものである。
11.葉耳と葉舌
イネ科植物の葉は、先端側の葉身と基部側の葉鞘から構成される。葉身と葉鞘の接点にある組織をそれぞれ葉耳と葉舌と呼ぶ。
12.オルソログ
共通の祖先遺伝子から種分岐の過程で生じ、同じ機能を持つ遺伝子を指す。
国際共同研究グループ
理化学研究所 環境資源科学研究センター
生産機能研究グループ(研究当時)
グループディレクター(研究当時) 榊原均(さかきばら ひとし)
研究員(研究当時) 木羽 隆敏(きば たかとし)
統合ゲノム情報研究ユニット(研究当時)
ユニットリーダー(研究当時) 櫻井 哲也(さくらい てつや)
Danforth Plant Science Center(米国)
研究員 プ・ハン(Pu Huang)
研究員 マキシミリアン・フェルドマン(Maximilian Feldman)
研究員ユンキン・ユー(YunqingYu)
研究技術員 レイチェル・タバレス(Rachel Tavares)
主任研究員 ドミトリ・ヌシノウ(Dmitri A. Nusinow)
主任研究員 イバン・バクスター(Ivan Baxter)
主任研究員(研究当時) トマス・ブルートネル(Thomas P. Brutnell)
主任研究員 エリザベス・ケロッグ(Elizabeth A. Kellogg)(責任著者)
Hudson AlphaInstitute for Biotechnology(米国)
上級研究員 スジャン・マミディ(Sujan Mamidi)
研究員 アダム・ヒーリー(Adam Healey)
主任研究員 ジェーン・グリムウッド(Jane Grimwood)
グループリーダー ジェリー・ジェンキンス(Jerry Jenkins)
研究員 アビナッシュ・スリーダシャム(Avinash Sreedasyam)
研究員 ジョン・ロベル(John T. Lovell)
研究員 ジンシャー・ウー(Jinxia Wu)
主任研究員 ジェレミー・シュルツ(Jeremy Schmutz)
JointGenomeInstitute(米国)
プロジェクトマネージャー ケリー・バリー(Kerrie Barry)
研究員 シェンキャン・シュー(Shengqiang Shu)
上級研究員 シンディー・チェン(Cindy Chen)
研究員 ジェニファー・ジョンソン(Jenifer Johnson)
原論文情報
Sujan Mamidi, Adam Healey, Pu Huang, Jane Grimwood, Jerry Jenkins, Kerrie Barry, Avinash Sreedasyam, Shengqiang Shu, John Lovell, Maximilian Feldman, Jinxia Wu, Yunqing Yu, Cindy Chen, Jenifer Johnson, Hitoshi Sakakibara, Takatoshi Kiba, Tetsuya Sakurai, Rachel Tavares, Dmitri A. Nusinow, Ivan Baxter, Jeremy Schmutz, Thomas P. Brutnell, Elizabeth A. Kellogg, “A genome resource for green millet Setaria viridis enables discovery of agronomically valuable loci”, Nature Biotechnology, 10.1038/s41587-020-0681-2
発表者
理化学研究所
環境資源科学研究センター 質量分析・顕微鏡解析ユニット
客員主幹研究員 榊原 均(さかきばら ひとし)
(バトンゾーン研究推進プログラム植物新育種技術研究チーム客員主幹研究員、名古屋大学生命農学研究科教授)
客員主幹研究員 櫻井 哲也(さくらい てつや)
(高知大学総合科学系複合領域科学部門准教授)
合成ゲノミクス研究グループ
客員研究員 木羽 隆敏(きば たかとし)
(バトンゾーン研究推進プログラム植物新育種技術研究チーム客員主幹研究員、名古屋大学生命農学研究科准教授)
報道担当
理化学研究所 広報室 報道担当