2021-04-09 京都大学,国立国際医療研究センター,長崎大学,日本医療研究開発機構
概要
京都大学学際融合教育研究推進センター スーパーグローバルコース医学生命系ユニットの長﨑正朗 特定教授、Gervais Olivier研究員(研究当時)、国立国際医療研究センター ゲノム医科学プロジェクト 戸山プロジェクト長 徳永 勝士らのグループは、長崎大学大学院医歯薬総合研究科教授/国立病院機構長崎医療センター客員研究員の中村稔らのグループが世界規模で臨床研究を進めている、原発性胆汁性胆管炎(primary biliary cholangitis; PBC)(注1)(合計1,953人)の日本人遺伝子データベースと日本人の一般集団の全ゲノムデータベース(合計3,690人)との比較を行いました。その結果、日本人では今まで報告がない3箇所の新規領域を含む、合計7箇所の染色体(注2)上の疾患に関わる候補領域を、ほとんどヒトゲノム情報に適用されたことがないポリジェニック効果を考慮した手法、領域内遺伝率推定法(Regional Heritability Mapping法; RHM)(注3)による、ゲノム解析から同定しました。
また、再現性を確認するため、3か所の新規染色体対象領域に含まれるSNP(一塩基多型)のうち、統計量が一番有意であったSNPについて、前述の集団とは独立したPBC患者(合計220人)と、京都府立医科大学 大学院医学研究科 上田 真由美特任准教授から提供された一般集団の全ゲノム情報(合計271人)との間で遺伝型の頻度に有意差があるか解析を行いました。その結果、日本人では今まで報告がない3箇所の遺伝子領域(STAT4(注4)、ULK4(注5)、KCNH5(注6)) いずれについても疾患に関係することを再確認できました。
さらに、これら3か所の遺伝子のうち特に海外でも報告例がないULK4とKCNH5について、PBCの患者由来の遺伝子と非罹患群の遺伝子のトランスクリプトーム発現量を比較ました。その結果、ULK4を含む領域についてPBC罹患群で発現上昇していることを見出しました。
RHM法をヒト全ゲノム解析に適用することで新規の遺伝要因を同定したこと、独立した集団を用いてRHM法で同定された遺伝要因の再検証をする一連のスキームを確立したこと双方の成果が世界初になります。同手法は、PBC疾患以外の多因子疾患にも幅広く適用可能であり、今後さまざまな疾患にも適用することで一般的なゲノム情報解析手法では見逃されていた遺伝要因を新たに同定できると期待しています。
本研究は、科学雑誌「European Journal of Human Genetics」オンライン版(9日付:日本時間2021年4月9日)に掲載されます。
研究のポイント
- 日本人遺伝子データベース(PBC罹患群と一般集団)を比較解析し日本人のPBC(primary biliary cholangitis)疾患に関わる3つの新規遺伝子候補領域(STAT4,ULK4,KCNH5)を同定
- 同ゲノム解析に、領域内遺伝率推定法(Regional Heritability Mapping; RHM)法を活用
- 独立した日本人遺伝子データベース(PBC罹患群と一般集団)を比較解析し3か所の再現性を検証
- PBC罹患群と非罹患群との間でULK4とKCNH5の肝臓における遺伝子発現量を比較し、ULK4遺伝子がPBC罹患群で発現上昇することを確認
- RHM法をヒト全ゲノム解析に適用し新規の遺伝要因を同定したこと、および、独立した集団に対しRHM法で同定された遺伝要因を検証する一連のスキームを確立したこと双方が世界初
- 今後、同手法は幅広く新規の遺伝要因の探索に適用可能
背景
世界初のゲノムワイド関連解析(GWAS)法(注7)(参考文献1)が2002年に発表されて以来、数多くのGWAS研究が実施され、GWASのデータを集積したデータベースGWAS Catalog (注8)によると、GWASの論文が4000報以上発表されており、何百もの形質に対して延べ20万近くの関連遺伝子座が同定されています(参考文献2)。
また、2型糖尿病(注9)や高脂血症(注10)をはじめとし、GWASの結果を創薬に活用した成功例等が見られるようになってきており、疾患感受性遺伝子(注11)の同定が疾患病態の解明や創薬開発に役立つことが明らかになってきています(参考文献3、4)。そのため、今後もGWASにより得られた成果を活かすことでさまざまな疾患の治療法の開発に役立てられていくと期待しています。
しかし、通常GWASは、ゲノム全体をカバーする数十万個から数百万個の代表的な変異(注12)であるSNP(一塩基多型)(注13)に関する情報を利用し、特定の疾患や形質などとの関連を調べる統計手法であり、変異毎に検定を行います。つまり、各SNPに対する検定を個別に実施するため、各検定では対象となっている変異以外のマーカーの効果を基本的なモデルにおいて考慮をしていません。
一方、近年、大きな効果をもたらす個々の変異を探索することを研究対象としたGWASに比して、微小な働きを持つ多数の変異の集合体の効果を研究対象としたポリジェニックモデル(注14)が医学分野において注目を集めています(参考文献5、6)。
研究手法・成果
「日本人大規模全ゲノム情報を基盤とした多因子疾患関連遺伝子の同定を加速する情報解析技術の開発と応用」として、ヒトゲノム情報解析技術の開発を行い、その成果を、幅広く日本人を中心とした多因子疾患(注15)のゲノム情報について適用する研究を進めてきました。
その研究開発の中で、前述のGWAS法に対して、染色体上の単位領域に含まれる、微小な働きをもつ多数の変異の集合(ポリジェニック)としての効果を考慮する手法の1つ領域内遺伝性マッピング(RHM)法に着目をしました。さらに、RHM法の適用例として、希少難治性疾患(注16)の1つである原発性胆汁性胆管炎(PBC)を対象として以下の解析を行いました。
約66万SNPを搭載するSNPアレイ(注17)、ジャポニカアレイv1(注18)(参照文献7)、また、約60万SNPを搭載するSNPアレイ、Axiom Genome-Wide ASIアレイ(注19)を用いて計測を行った日本人のPBC罹患群(計1,953人)と日本人の一般集団の全ゲノムデータ(計3,690人)に対し、約2,000人の東北メディカル・メガバンク機構(注20)の全ゲノムリファレンスパネル(注21)を用いて未観測の領域の変異を推定しました(参考文献8)。さらに、一定以上の相関を持つ変異のうちの代表のSNPを抽出することで最終的に約100万か所の変異を抽出し、その変異群に対してGWAS法とRHM法それぞれを用いて解析を行いました。
その結果、図1に示すように、GWAS法を用いることで検出することができた染色体上の領域(図1下)に比べ、RHM法では新たにSTAT4、ULK4、KCNH5の3つの領域(図1上)について、統計的に有意な水準(注22)を超える領域として検出することができました。
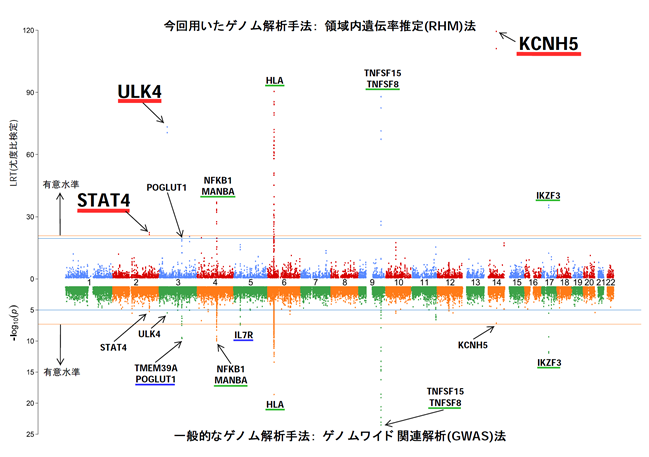
図1 PBC罹患群と一般集団とのゲノム情報解析結果の比較 提案手法RHM法(上)及び、一般的なGWAS法(下)による。新規領域を赤下線で表示している。詳細説明は参考参照
次に、得られた結果の信頼性を確認するため、日本人では今まで報告がない3箇所の領域 (STAT4、ULK4、KCNH5)の各領域に含まれる一番有意であった変異に対して、前述の集団とは独立したPBC罹患群(計220人)と一般集団(計271人)の変異の偏りについて検定を行ったところこれらの変異についてすべて有意な差を得ることができました。
さらに、STAT4についてはヨーロッパでPBC疾患との関連性が過去に報告されていましたが、残りの2つ(ULK4 および KCNH5)については日本人以外の集団おいても PBC 疾患との関連が報告されていない新規の遺伝子でした。これらの遺伝子は、従来の単一 SNP GWAS では検出できなかったことから、新たなヒト疾患感受性遺伝子の検出に RHM法が有効であることをヒトゲノムの実際のデータを用いて実証することができたといえます。
また、肝臓組織のmRNAマイクロアレイ解析(注23)により、PBC患者ではULK4の遺伝子発現レベルが高いことが明らかになり、ゲノム情報の解析によって得られた結果がPBC疾患に機能的に関連する可能性が高いことが示唆されました。
なお、シミュレーション研究(注24)では、複数の隣接する変異を統合することでゲノムの特定領域の遺伝的影響を推定する領域内遺伝率推定(RHM)法は、 GWAS法よりも多くの場合高い検出力を持つことが予測されていました(参考文献9、参考文献10)。しかし、これまでのところ、その利用は農業分野を中心に行われており、ヒト疾患における新規遺伝子発見のための全ゲノム領域への適用はなされていませんでした。
そのため、本報告は、RHM法をヒトゲノム解析に適用し、新規の疾患関連遺伝子を同定することに成功した世界初の成果となります。
波及効果、今後の予定
本研究においては、RHM法をPBC疾患に適用することで新たな遺伝要因を探索することを実証しましたが、本手法は、いままで解析が行われていたGWAS Catalogに登録されている4,000報以上の多因子疾患ゲノム情報についても適用できます。つまり、従来の単一 SNP GWASなどの一般的なゲノム解析手法において見逃されていた遺伝的要因を同様に新たに同定していくことができます。
一方、同手法の解析には、大規模な電算機資源(注25)が必要となるため、より計算量を削減した形での計算実装を行う改良を進めることや富岳(注26)などのより大規模な計算機に適した計算アルゴリズムと実装にするなどが考えられます。
なお、1つの変異の効果が強い場合などGWAS法でしか同一のサンプル数で有意水準に到達できない変異が存在することもあるため(例:図1のPOGLUT1、IL7R)、RHM法はGWASの代替というわけではなく、併用する手法が有効であると考えています。
より詳細なRHMの内容については、「遺伝子医学」通巻36号「ポリジェニックモデルによるゲノム情報解析の最前線」(2021年発刊予定)でも説明予定ですので参照ください。
論文タイトルと著者
- タイトル:
- 領域内遺伝率推定法による日本人集団における原発性胆汁性胆管炎の新たな遺伝要因(STAT4、ULK4とKCNH5)の同定
Regional heritability mapping identifies several novel loci (STAT4, ULK4, and KCNH5) for primary biliary cholangitis in the Japanese population - 著者:
- Olivier Gervais, Kazuko Ueno, Yosuke Kawai, Yuki Hitomi, Yoshihiro Aiba, Mayumi Ueta, Minoru Nakamura, Katsushi Tokunaga, Masao Nagasaki
- 掲載誌:
- European Journal of Human Genetics
- DOI:
- 10.1038/s41431-021-00854-5
研究支援
PBC疾患のRHM遺伝要因のディスカバリー解析については主に、⽇本医療研究開発機構(AMED)のゲノム医療実現推進プラットフォーム事業先端ゲノム研究開発(GRIFIN)「日本人大規模全ゲノム情報を基盤とした多因子疾患関連遺伝子の同定を加速する情報解析技術の開発と応用(研究開発代表者:徳永勝士);JP19km0405205」、PBC疾患のレプリケーション解析は主に、ゲノム医療実現推進プラットフォーム事業 国際的データシェアリングに関する課題解決のための調査研究及び開発研究「クラウド計算環境を利用したゲノム医科学研究の倫理・技術課題の調査と実践(研究開発代表者:徳永勝士);JP20km0405501」による支援を受けて行われました。
プロトタイプの手法開発には、科研費(若手研究) 「ポリジェニックモデルによる慢性腎臓病・メタボリックシンドローム関連形質ゲノム解析(研究代表者:Olivier Gervais);18K14757」、PBCの試料、ゲノム情報、トランスクリプトーム情報の取得は、国立病院機構共同臨床研究「原発性胆汁性肝硬変の病態解明と新たな分子標的治療法の開発のためのゲノムワイド関連解析(研究代表者:中村稔)」、国立病院機構共同臨床研究「原発性胆汁性胆管炎の発症と重症化機構解明のための多施設共同研究(研究代表者:中村稔)」、国立病院機構共同臨床研究「原発性胆汁性胆管炎の新しい病型分類と創薬のための長期観察研究(研究代表者:中村稔)」、科研費(基盤研究C)「日本人原発性胆汁性肝硬変の病態形成に関わる遺伝因子同定のための網羅的遺伝子解析(研究代表者:中村稔)」、科研費(基盤研究B)「原発性胆汁性肝硬変の疾患感受性遺伝子による病態の解明と新しい分子標的治療法の開発(研究代表者:中村稔)」、科研費(基盤研究B)「原発性胆汁性胆管炎の発症と重症化機構解明のためのGWASを基盤とした統合解析(研究代表者:中村稔)」、科研費(若手研究B)「原発性胆汁性肝硬変の黄疸・肝不全型進行に関わる分子機構の解明(研究代表者:相葉佳洋)」、科研費(基盤研究C)「原発性胆汁性胆管炎の肝不全進行におけるカテプシンZの役割の解明(研究代表者:相葉佳洋)」を用いて行われました。電算機資源として、学際大規模情報基盤共同利用・共同研究拠点の支援「大規模ゲノム情報解析にむけた数値計算技術開発と実装(研究代表者:徳永勝士);190055-DAJ」、「ハイブリッドクラウド構築とゲノム情報解析の効率的な運用に関した研究(研究代表者:長﨑正朗); jh200047-NWH」、情報・システム研究機構 国立遺伝学研究所が有する遺伝研スーパーコンピュータシステム、東北大学東北メディカル・メガバンク機構のスーパーコンピュータを利用しました。
用語説明
- (注1)原発性胆汁性胆管炎 (Primary Biliary Cholangitis)
- 原発性胆汁性肝硬変とは、中高年女性に多い比較的まれな(患者総数は全国で約5–6万人と推定)胆汁うっ滞性肝疾患であり、進行すると黄疸、肝不全となり肝移植以外に救命方法がない難病である。その原因としては自己免疫的機序により肝臓内の小型胆管が破壊されることが考えられているが、詳細については未だ不明であり、根治的治療法も開発されていない。
- (注2)染色体
- 各個人の遺伝情報を保持する媒体。ヒト染色体は、22本の常染色体と、2本の性染色体から構成される。本解析では、常染色体を対象として解析を行っている。(図1参照)
- (注3)領域内遺伝率推定法 (Regional Heritability Mapping)
- 本研究において用いた特定領域の遺伝分散(遺伝率)の推定手法。染色体の特定領域の遺伝的効果を環境要因及び領域以外の全ゲノムによって生じるポリジーン効果と分けて推定する。下記の式であらわされるモデルでは、環境要因や主成分の効果(Xβ)を取り除き、全SNPによるポリジーン効果(Zu)と、染色体上の特定領域(SNP群、Zw)の効果を分けて推定することができる。従来のGWASの場合、各マーカーの効果を推定することになるが、下式のモデルでは例えば、特定の遺伝子に連鎖するSNP群を一つの領域として扱うことができるため、個々のSNPの効果ではなく、領域内の複数のSNPやその組み合わせの効果も含んで推定できる。つまりこの方法では、染色体の特定領域(連続したSNP群)によって引き起こされる遺伝分散(遺伝率)を推定し、有効なSNP群があるか、否かを検定することができる。隣接した多数のSNP(数十~数百個)群の効果を推定し、全ゲノムの各領域を「スキャン」するこのモデルをRegional Heritability Mapping法(領域内遺伝率推定法、RHM法)と呼ぶ(参考文献10,11)。
y=Xβ+Zu+Zw+e
yは解析対象となっている表現型(=疾患や量的形質、測定値)
Xβは環境要因(=遺伝以外の要因)
Zuは遺伝要因(=全SNPによるポリジーン効果)
Zwは遺伝要因(=特定のSNP群(数十~数百SNP)による効果)
eは誤差 - (注4)STAT4
- Signal Transducer and Activator of Transcription 4
ヒトゲノム上の遺伝子の1つ。海外ではすでにPBC疾患に関連することが報告されていたが、本研究で、日本人のPBC疾患においても関連することが初めて確認された遺伝子。 - (注5)ULK4
- Unc-51 Like Kinase 4
ヒトゲノム上の遺伝子の1つ。本研究で、日本人のPBC疾患に関連すると初めて同定された遺伝子。 - (注6)KCNH5
- Potassium Voltage-Gated Channel Subfamily H Member 5
ヒトゲノム上の遺伝子の1つ。本研究で、日本人のPBC疾患に関連すると初めて同定された遺伝子。 - (注7)ゲノムワイド関連解析 (Genome Wide Association Study; GWAS)
- 疾患の罹患有無や身長・体重などの量的な形質に影響がある遺伝的な要因を変異毎に統計的な偏りがないか網羅的に調査する手法。一般的には、SNPアレイ(近年では全ゲノムシークエンス)などで取得された各変異(SNPアレイの場合にはSNP)に対して行う。
- (注8)GWAS Catalog
- 国内外のGWASのデータを集積したデータベース
- (注9)2型糖尿病
- インスリンが出にくくなったり、インスリンが効きにくくなったりすることによって血糖値が一般集団の基準値より高い値になっている症状をさす。遺伝的な要因と生活習慣の双方が関連する。
- (注10)高脂血症
- 多因子疾患の1つ。中性脂肪やコレステロールの値が一般集団の基準値より高い値になっている症状をさす。遺伝的な要因と生活習慣の双方が関連する。
- (注11)疾患感受性遺伝子
- 多因子疾患の発症に関わる遺伝子のこと。通常、疾患に対して弱い効果しかない場合が多く、多くの要因が重なって疾患を形成する。
- (注12)変異
- 個人毎の染色体上の塩基配列の違いを指す。ヒト全ゲノムシークエンスによって得られた情報を大型計算機やクラウド環境などで計算をすることでヒトゲノム国際参照配列に対して個人毎に異なる部分(変異)の同定をすることができる。個人毎に、通常、国際参照配列に対して数百万か所の変異が検出される。
- (注13)SNP
- 変異のうち特に1塩基の集団中の差異を指す。一塩基多型(Single Nucleotide Polymorphism)が正式名称。
- (注14)ポリジェニックモデル (Polygenic Model)
- 標準的なポリジェニックモデルは測定値(疾患の発症の有無も含む)から環境要因を除き、遺伝要因(=ポリジーン)による効果を推定するための統計モデルである。
- (注15)多因子疾患
- 疾患のうち、ヒトゲノム中の多数の弱い効果をもつ遺伝要因が複合的に疾患の罹患に影響を与えているような疾患を多因子疾患と呼ぶ。2型糖尿病や高脂血症などがその例。
- (注16)希少難治性疾患
- 患者数が非常に少なく有効な治療法が存在しない難病
- (注17)SNPアレイ
- DNAから約数十万から約数百万か所の決められたSNPを計測することができる計測技術を指す。本解析においては、ヒトのDNAから数十万か所のSNPの計測を行っている。
- (注18)ジャポニカアレイ v1
- SNPアレイの1つ。日本人集団から構築した全ゲノムリファレンスパネルを活用し、日本人集団で共通しているSNPを主に対象とすることで設計をおこなった日本人集団に適したSNPアレイ。
- (注19)Axiom Genome-Wide ASIアレイ
- SNPアレイの1つ。東アジア人の集団を対象にすることで設計されたSNPアレイ。
- (注20)東北メディカル・メガバンク機構
- 宮城県、岩手県の健康調査を通じてバイオバンクを構築し、遺伝子の研究を発展させて、次世代型医療を創り出そうとしているプロジェクト。
URL:https://www.megabank.tohoku.ac.jp/ - (注21)全ゲノムリファレンスパネル
- 個人毎の国際参照配列に対する変異(個人毎に共通する変異もあるため)を統合することで構築することができるカタログ。多数の集団から構成される国際1,000人ゲノムプロジェクト(参考文献12)によって構築された全ゲノムリファレンスパネルや、日本人集団から構築した東北メディカル・メガバンク機構で構築された1,070人からなる全ゲノムリファレンスパネル(現在は数千人規模)などがある(参考文献8)。本プロジェクトでは、約2,000人の全ゲノムリファレンスパネルを用いている。
- (注22)統計的に有意な水準
- ある帰無仮説を考えた時に、間違ってその正しい帰無仮説を棄却してしまう危険性が起こりえる可能性を確率(有意水準)として表現したときに、その帰無仮説が有意水準以上の確率で差がある場合、統計的に有意な水準であるという。本研究の帰無仮説は、疾患群と一般集団の間で染色体上のある対象領域の遺伝的な効果に差がないという仮説を指す。
- (注23)mRNAマイクロアレイ解析
- ヒトゲノム中に含まれる約2万遺伝子のmRNAの発現量を一度に計測する技術。ここではヒト肝臓組織細胞中から抽出されたmRNAの計測を行っている。
※mRNA:メッセンジャーRNA (messenger RNA) 。RNAポリメラーゼによって遺伝子 (DNA) から 転写された、タンパク質の配列情報をコードするRNAをさす。 - (注24)シミュレーション研究
- ここでは数学的に定義が可能な理想的なモデルを仮定し、そのモデルに基づいて計算機上で観測データを生成し、さらに、元のモデルに含まれるパラメータの予測を複数の手法を用いて計算機上で行い、それらの手法間の予測性能を比較評価するような研究を指す。
- (注25)大規模な計算資源
- ここでは、スーパーコンピュータやクラウドコンピューティングを指す。
- (注26)富岳
- 日本のスーパーコンピュータの1つであり2021年度より本格稼働を予定している世界最高水準のコンピュータ。
参考文献
- Functional SNPs in the lymphotoxin-alpha gene that are associated with susceptibility to myocardial infarction. Ozaki K, Ohnishi Y, et al. Nat Genet. 32 (4): 650-4. 2002.
- The NHGRI-EBI GWAS Catalog of published genome-wide association studies, targeted arrays and summary statistics 2019. Buniello A, MacArthur J A L, et al. Nucleic Acids Res. 47 (D1): D1005-D1012. 2019.
- The Influence of Big (Clinical) Data and Genomics on Precision Medicine and Drug Development. Denny J C, Van Driest S L, et al. Clin Pharmacol Ther. 103 (3): 409-418. 2018.
- Genetics of rheumatoid arthritis contributes to biology and drug discovery. Okada Y, Wu D, et al. Nature. 506 (7488): 376-81. 2014.
- Research review: Polygenic methods and their application to psychiatric traits. Wray N R, Lee S H, et al. J Child Psychol Psychiatry. 55 (10): 1068-87. 2014.
- Genetic variance estimation with imputed variants finds negligible missing heritability for human height and body mass index. Yang J, Bakshi A, et al. Nat Genet. 47 (10): 1114-20. 2015.
- Japonica array: improved genotype imputation by designing a population-specific SNP array with 1070 Japanese individuals, Y. Kawai, T. Mimori, K. Kojima, N. Nariai, I. Danjoh, R. Saito, J. Yasuda, M. Yamamoto, and M. Nagasaki. J Hum Genet, 60(10): 581-587, 2015.
- M. Nagasaki, J. Yasuda, F. Katsuoka, N. Nariai, K. Kojima, Y. Kawai, Y. Yamaguchi-Kabata, J. Yokozawa, I. Danjoh, S. Saito, Y. Sato, T. Mimori, K. Tsuda, R. Saito, X. Pan, S. Nishikawa, S. Ito, Y. Kuroki, O. Tanabe, N. Fuse, S. Kuriyama, H. Kiyomoto, A. Hozawa, N. Minegishi, J. Douglas Engel, K. Kinoshita, S. Kure, N. Yaegashi, and M. Yamamoto. Rare variant discovery by deep whole-genome sequencing of 1,070 Japanese individuals. Nat Commun, 6: 8018, 2015.
- The power of regional heritability analysis for rare and common variant detection: simulations and application to eye biometrical traits. Uemoto Y, Pong-Wong R, et al. Front Genet. 4: 232. 2013.
- Localising loci underlying complex trait variation using Regional Genomic Relationship Mapping. Nagamine Y, Pong-Wong R, et al. PLoS One. 7 (10): e46501. 2012.
A global reference for human genetic variation. 1000 Genomes Project Consortium. Nature, 526(7571):68-74. 2015.
お問い合わせ先
京都大学学際融合教育研究推進センター スーパーグローバルコース医学生命系ユニット
京都大学大学院医学研究科附属ゲノム医学センター
特定教授 長﨑 正朗 (ながさき まさお)
研究員(当時) Gervais Olivier (じぇるべ おりびえ)
(現在)日本大学 国際関係学部 国際総合政策学科、助教
国立研究開発法人 国立国際医療研究センター
ゲノム医科学プロジェクト
戸山プロジェクト長 徳永 勝士(とくなが かつし)
副プロジェクト長 河合 洋介(かわい ようすけ)
研究員 植野 和子(うえの かずこ)
客員研究員 人見 祐基(ひとみ ゆうき)
(星薬科大学薬学部微生物学研究室特任講師)
長崎大学大学院医歯薬学総合研究科新興感染症病態制御学系専攻肝臓病学講座教授
独立行政法人国立病院機構 長崎医療センター臨床研究センター客員研究員
中村 稔(なかむら みのる)
独立行政法人国立病院機構 長崎医療センター臨床研究センター
研究員相葉 佳洋(あいば よしひろ)
報道に関する問い合わせ
京都大学総務部広報課国際広報室
国立国際医療研究センター(NCGM)
企画戦略局 広報企画室 広報係長 西澤 樹生(にしざわ たつき)
AMED事業に関するお問い合わせ
日本医療研究開発機構(AMED)
ゲノム・データ基盤事業部 ゲノム医療基盤研究開発課
ゲノム医療実現推進プラットフォーム事業(先端ゲノム研究:GRIFIN)担当