2023-06-13 国立成育医療研究センター
国立成育医療研究センター(所在地:東京都世田谷区大蔵、理事長:五十嵐隆)のエコチル調査研究部・遺伝子解析室の熊坂夏彦チームリーダーは、イギリスのサンガー研究所と共同で、個々の細胞の遺伝子発現を解析する新たなツール「GASPACHO(ガスパチョ)」を開発しました。
このツールを用いて、自然免疫[1]応答下における遺伝子発現にどのような個人差があるのかを調べたところ、ヨーロッパ人の集団ではOAS1(抗ウイルス免疫に関わる遺伝子)に一塩基多型[2]が存在し、その変異アレルによってOAS1の正常な発現が低下することで新型コロナウイルス感染症(COVID-19)に感染・重症化しやすくなることを突き止めました。この成果によって、COVID-19の発症メカニズムの一旦が明らかになるとともに、OAS1が存在するカスケード(細胞内で起こる様々な連鎖的な反応)が今後、治療標的になる可能性も示唆されました。
なお、今回新しく開発された一細胞の遺伝子発現を解析するツールは、自然免疫応答に限らず、細胞の初期発生やストレス応答など、さまざまな細胞状態の連続的な変化を捉えることができる非常に汎用なもので、多くの機能ゲノム研究で活用されることが期待されます。
本研究成果は、遺伝学の国際的な学術誌「Nature Genetics」に6月12日に掲載されました。
[1] 自然免疫:病原体(細菌やウイルスなど)が体内に入ってきた際に、免疫細胞がこれらを排除しようとする反応。
[2] 一塩基多型:ある集団において、個人間でDNAの塩基配列の違いがある場所のことを多型といい、そのうち1つの塩基が別の塩基に置き換わっていることを一塩基多型といいます。また一塩基多型において、ヒトの参照塩基配列に存在する塩基を参照アレル、存在しない塩基を変異アレルと呼びます。
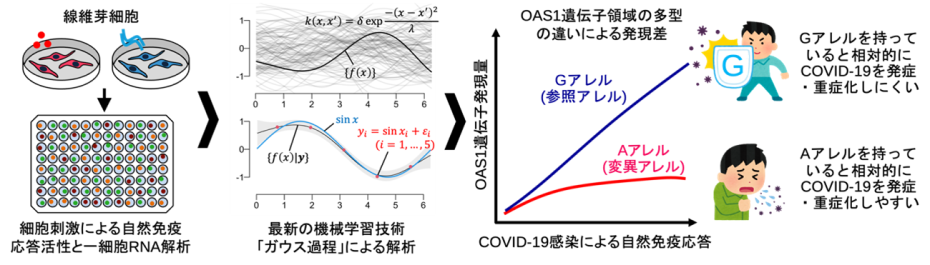
【図1:研究の概念図と主な発見】
プレスリリースのポイント
- 最新の機械学習技術である「ガウス過程 (Gaussian Process)」を一細胞RNA解析データに応用したツール:GASPACHO (GAuSsian Processes for leveraging Cell HeterOgeneity)を開発しました。
- このツールを用いて、複数の個体から得られた線維芽細胞の自然免疫応答下における遺伝子発現の個人差を解析しました。
- その結果、OAS1遺伝子内の一塩基多型rs10774671 (12番染色体のDNA配列の112,919,388番目の塩基GがA置き換わる変異)によって、免疫応答下で正常に遺伝子が発現しないこと、また、この遺伝子発現の低下によってCOVID-19に感染しやすくなることが明らかになりました。
- この一塩基多型は、COVID-19の発症および重症化に関わるリスク変異であることが国際的共同研究機関(The COVID-19 Host Genetics Initiative)によって報告されていましたが(発症および重症化のオッズ比=1.062および1.2)、なぜリスクを高めるのかに関しては正確に分かっていませんでした。なお、OAS1は細胞に感染したウイルスのRNAを分解・排除する機能の一翼を担っていることが知られており、西ナイルウイルス感染などにも影響を与えることが報告されています。
- この成果は、OAS1とそのカスケード(細胞内で起こる様々な連鎖的な反応)に存在する遺伝子が、今後COVID-19の発症や重症化に関する治療標的になる可能性を示唆するものです。
研究概要
- 最新の機械学習技術である「ガウス過程 (Gaussian Process)」を一細胞RNA解析データに応用したGASPACHO (GAuSsian Processes for leveraging Cell HeterOgeneity)というツールを開発しました。
- イギリス・サンガー研究所を中心とするヒト人工多能性幹細胞イニシアチブ(HipSci)に登録された68名のドナー(ヨーロッパ人)から得られた線維芽細胞に、ウイルス感染を模倣する細胞刺激(インターフェロンβと合成二重鎖RNA)を加え、自然免疫応答下における細胞の遺伝子発現を一細胞RNA解析技術によって定量化しました。
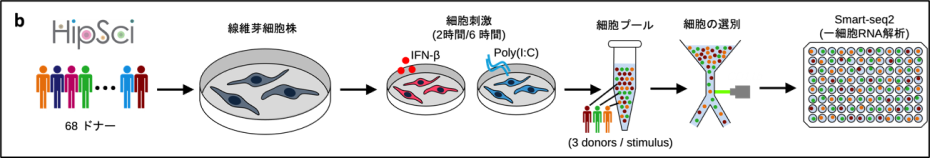
- 自然免疫応答に関わる潜在変数を抽出することに成功し、その変数に沿った遺伝的関連解析を行いました。そして免疫応答における個人間の遺伝子の発現の差を生み出す遺伝領域を網羅的に同定しました。
- その多くが、関節リウマチやアレルギーなどの自己免疫疾患と共局在化(原因となる多型を共有)していることがわかりました。そしてその一つがOAS1遺伝子で同定され、COVID-19の発症リスクのゲノムワイド関連解析で同定されたリスク多型(rs10774671G>A)と共局在化し ていることを発見しました。
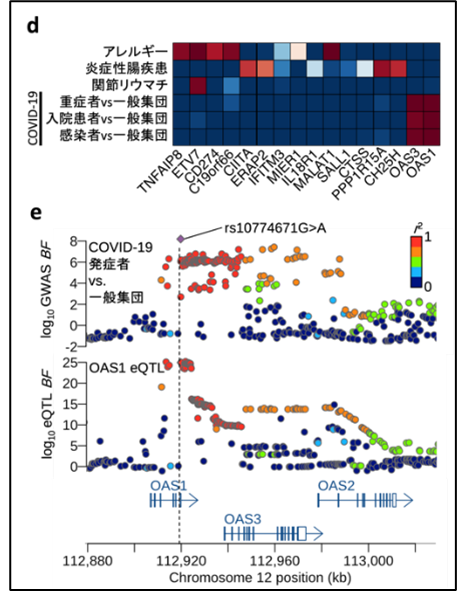
- OAS1は免疫応答に沿って発現が上昇しますが、GアレルからAアレルへの変異があることによって免疫応答下でその発現が低下することを確認しました。ここでAアレルがCOVID-19の発症と重症化のリスクを上昇させることが知られているため、結果としてOAS1の発現量の低下がCOVID-19の感染と重症化のリスクを上昇させることが示されました。また、次世代シークエンサーから得られたリード・カバレージ・データからは、アレルの変異によってOAS1の異なるアイソフォーム(どれもタンパク質レベルで機能していないという報告あり)が発現していることも明らかになりました。
- 追加検証として、実際にCOVID-19に感染した患者から得られた鼻腔内の粘膜細胞および血漿中の免疫細胞(CD16+単球)において、量的形質座位とアイソフォームの発現を追認しました。
発表論文情報
タイトル:Mapping interindividual dynamics of innate immune response at single-cell resolution
執筆者:Natsuhiko Kumasaka1,2, Raghd Rostom1,3, Ni Huang1, Krzysztof Polanski1, Kerstin B. Meyer1, Sharad Patel1, Rachel Boyd1, Celine Gomez1, Sam N. Barnett1, Nikolaos I Panousis1, Jeremy Schwartzentruber1,4, Maya Ghoussaini1,4, Paul A. Lyons5,6, Fernando J. Calero-Nieto7, Berthold Göttgens7, Josephine L. Barnes8, Kaylee B. Worlock8, Masahiro Yoshida8, Marko Z. Nikolić8,9, Emily Stephenson1,10, Gary Reynolds10, Muzlifah Haniffa1,10,11,12, John Marioni1,3,13, Oliver Stegle1,3,14,15, Tzachi Hagai16, Sarah A. Teichmann1,17
所属:
1. Wellcome Sanger Institute, Wellcome Genome Campus, Cambridge, UK
2. National Center for Child Health and Development, Medical Research Center for JapanEnvironment and Children’s Study, Tokyo, Japan
3. European Molecular Biology Laboratory, European Bioinformatics Institute, Wellcome Genome Campus, Hinxton, UK
4. Open Targets, Wellcome Genome Campus, Hinxton, Cambridgeshire CB10 1SD, UK
5. Cambridge Institute of Therapeutic Immunology and Infectious Disease, Jeffrey Cheah Biomedical Centre, Cambridge Biomedical Campus, Cambridge, UK
6. Department of Medicine, University of Cambridge, Cambridge Biomedical Campus, Cambridge, UK
7. Wellcome – MRC Cambridge Stem Cell Institute, University of Cambridge, Cambridge, UK
8. UCL Respiratory, Division of Medicine, University College London, London, UK
9. University College London Hospitals NHS Foundation Trust, London, UK
10. Biosciences Institute, Newcastle University, Newcastle upon Tyne, UK
11. NIHR Newcastle Biomedical Research Centre, Newcastle Hospitals NHS Foundation Trust, Newcastle upon Tyne, UK
12. Department of Dermatology, Newcastle Hospitals NHS Foundation Trust, Newcastle upon Tyne, UK
13. Cancer Research UK Cambridge Institute, University of Cambridge, Cambridge, UK
14. Division of Computational Genomics and Systems Genetics, German Cancer Research Center, Heidelberg, Germany
15. European Molecular Biology Laboratory, Genome Biology Unit, Heidelberg, Germany
16. Shmunis School of Biomedicine and Cancer Research, George S Wise Faculty of Life Sciences, Tel Aviv University, Tel Aviv, Israel
17. Theory of Condensed Matter Group, Cavendish Laboratory/Department of Physics, University of Cambridge, Cambridge, UK
掲載誌:Nature Genetics
DOI:10.1038/s41588-023-01421-y