2019-09-03 理化学研究所,株式会社ダナフォーム,京都大学
理化学研究所(理研)生命医科学研究センター理研-IFOMがんゲノミクス連携研究チームの平林茂樹大学院生リサーチ・アソシエイト(京都大学大学院医学研究科博士課程4年)、村川泰裕チームリーダー、予防医療・ゲノミクス応用開発ユニットのシュルティー・バーガット国際プログラム・アソシエイト、川路英哉開発ユニットリーダー、株式会社ダナフォームの松木悠部長代理らの国際共同研究グループ※は、遺伝子の発現スイッチとして働く「エンハンサー[1]」と呼ばれる非コードゲノム領域を、高感度かつ一塩基レベルで検出し、さらに活性度を測定する技術を新たに開発しました。
本研究成果は、個体発生などを制御する生体機構の解明につながるほか、次世代ゲノム医療に貢献すると期待できます。
エンハンサーには、さまざまな病気の発症に関連するゲノム変異が高度に濃縮されています。しかし、ヒトゲノムのどこに、どれだけの数のエンハンサーが存在して、どのように活性化されるかは不明でした。
今回、国際共同研究グループは、新しく合成されている最中の「Nascent RNA」に着目し、Nascent RNAを迅速かつ高純度で精製する生化学手法を開発しました。そして、この技術とCAGE法[2]を組み合わせた「NET-CAGE法」を確立し、がん細胞中に存在する約3万の活性化エンハンサーを高精細に同定、さらにその活性化動態やトポロジー構造[3]を解明しました。
本研究は、国際科学雑誌『Nature Genetics』オンライン版(9月2日付け:日本時間9月3日)に掲載されます。
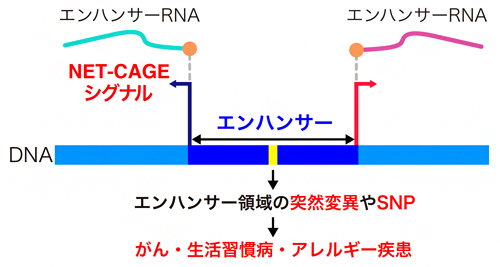
図 NET-CAGE法による疾患メカニズムの解明
※国際共同研究グループ
理化学研究所
生命医科学研究センター
理研-IFOMがんゲノミクス連携研究チーム
チームリーダー 村川 泰裕(むらかわ やすひろ)
(IFOM癌研究所グループリーダー)
大学院生リサーチ・アソシエイト 平林 茂樹(ひらばやし しげき)
(京都大学大学院 医学研究科 博士課程4年)
予防医療・ゲノミクス応用開発ユニット
開発ユニットリーダー 川路 英哉(かわじ ひでや)
(東京都医学総合研究所副参事研究員)
国際プログラム・アソシエイト シュルティー・バーガット(Shruti Bhagat)
(カロリンスカ大学院 博士課程3年)
副センター長 ピエロ・カルニンチ(Piero Carninci)
予防医療・診断技術開発プログラム
プログラムディレクター 林﨑 良英(はやしざき よしひで)
コーディネーター 伊藤 昌可(いとう まさよし)
株式会社ダナフォーム
部長 竹上 雄治郎(たけがみ ゆうじろう)
部長代理 松木 悠(まつき ゆう)
部員 金丸 愛(かねまる あい)
カロリンスカ大学
教授 ユハ・ケーレ(Juha Kere)
助教 片山 慎太郎(かたやま しんたろう)
京都大学大学院 医学研究科
血液・腫瘍内科学
教授 高折 晃史(たかおり あきふみ)
助教 白川 康太郎(しらかわ こうたろう)
京都大学大学院 医学研究科
医化学分野
教授 竹内 理(たけうち おさむ)
助教 植畑 拓也(うえはた たくや)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金若手研究A「エンハンサー同定の新技術を開発し、腫瘍に認める遺伝子発現異常の原因を解明する(研究代表者:村川泰裕)」、同基盤研究A「新規技術による白血病の包括的エンハンサー解析と分子病態解明(研究代表者:村川泰裕)」、同基盤研究B「ゲノムDNA転写開始活性の数理的構造を用いた制御領域リバースエンジニアリング(研究代表者:川路英哉)」、かなえ医薬振興財研究助成、小野医学研究財団研究助成、持田記念医学薬学振興財団研究助成金、日本応用酵素協会成人病の病因・病態の解明に関する研究助成、武田科学振興財団生命科学研究助成、日本医療研究開発機構(AMED)「オミックス解析による遺伝性網脈絡膜疾患、家族性緑内障、先天性視神経委縮症の病因・病態機序の解明(研究代表者:岩田岳)」による支援を受けて行われました。
背景
「エンハンサー」と呼ばれるゲノム領域は数百塩基の長さからなりますが、直接タンパク質を生成していません。エンハンサーに転写因子[4]が結合すると、標的遺伝子の発現にスイッチが入り、標的遺伝子が必要なタンパク質を生成することから、エンハンサーは遺伝子の発現制御において中心的な役割を担っているといえます。近年、活性化された状態のエンハンサー(活性化エンハンサー)の両端からは、エンハンサーRNA(eRNA)と呼ばれるノンコーディングRNA[5]が転写されていることが明らかになりました。
さらに、エンハンサーにはさまざまな病気に関連する一塩基多型(SNP)[6]が存在することや、エンハンサーのゲノム変異が発がんに関わることが報告されています(図1)。そのため、ヒトゲノム上に散らばっている活性化エンハンサーを、高感度かつ効率的に同定する技術の確立が急務となっています。
これまで、エンハンサーを同定・定量する技術として、ChIP-Seq法[7]やCAGE法が知られています。しかしChIP-seq法では、シグナルがブロードなため、一塩基レベルの高分解能でエンハンサーを同定できません。一方CAGE法では、転写されたRNAの5’末端の塩基配列を一塩基レベルで同定・定量することができます。従ってCAGE法を用いて、eRNAの転写開始点を同定すれば、高塩基解像度で活性化エンハンサーを検出できます注1)(図1)。
しかしeRNAの多くは、合成直後に細胞の核内で活発に分解されます。細胞内のトータルRNAを用いた従来のCAGE法では、細胞質に運ばれ安定的に蓄積している多量のメッセンジャーRNA(mRNA)のシグナルが大部分を占めることから、eRNAのシグナルはごくわずかしか捉えられないという問題がありました。そのため、活性化エンハンサーの同定感度は低く、ヒトゲノムに存在する活性化エンハンサーの数、動作メカニズム、トポロジー構造の詳細は不明でした。
注1)Andersson et al. Nature 2014 507(7493): 455-461 An atlas of active enhancers across human cell types and tissues
研究手法と成果
国際共同研究グループは、従来のCAGE法の問題点を解決するために、細胞内のトータルRNAではなく、RNA分解を受ける前のRNAポリメラーゼ[8]により新しく合成されている最中の新生鎖RNA(Nascent RNA)に着目しました。このNascent RNAには、半減期の短い不安定なeRNAが高い割合で含まれています。そして、細胞から細胞内分画化の工程のみで、Nascent RNAを迅速かつ高純度に回収・精製する生化学手法を開発しました。この技術と従来のCAGE法を組み合わせたところ、エンハンサーの検出感度を飛躍的に向上させることに成功し、この技術を「NET-CAGE(Native Elongating Transcript-CAGE)法」と名付けました(図2)。NET-CAGE法では、簡便な手順により、再現性の高いデータを産出でき、多検体の解析が可能です。また、生細胞だけでなく、凍結保存された細胞や組織に対しても適用できます。
さらに、NET-CAGE法と通常のCAGE法を組み合わせることで、異なる転写開始点から転写されるRNAの半減期を推定することにも成功しました。
次に、開発したNET-CAGE法を5種類の代表的なヒトがん細胞株(白血病細胞、リンパ芽球細胞、乳がん細胞、子宮頸がん細胞、肝がん細胞)に適用した結果、約3万の活性化エンハンサーが、一塩基レベルの高分解能で網羅的に同定されました。これで、FANTOMコンソーシアム[9]の第5期プロジェクトで同定されたものと合わせると、活性化エンハンサーは合計約9万に上ります。
また、複数のエンハンサーが連結したゲノム領域の内部構造である「スーパーエンハンサー」を一塩基レベルで明らかになりました。さらに、プロモーター[1]は多種の細胞で共通に使われているのに対して、エンハンサーは極めて細胞特異的に活性化されることが分かりました(図3)。
NET-CAGE法では、安定性の高いmRNAだけでなく、eRNAなど半減期の短いノンコーディングRNAの転写開始点と、さらに、これら全転写開始点からの(合成と分解のバランスではない)真の転写量がゲノムワイドに計測できます。そこで、NET-CAGE法により、細胞が刺激を受けた際の、エンハンサーとプロモーターの活性化度を経時的に厳密計測し、刺激に応答してエンハンサーとプロモーターは、多くの場合、同時に活性化することを新たに見いだしました。さらに、細胞特異的な遺伝子発現を可能にするエンハンサーとプロモーターのトポロジー構造も明らかにし、遺伝子発現制御の根幹をなすヒトゲノム構造を解明しました。
今後の期待
近年、ゲノム配列の違いによる個別化医療や予防医療が実現されつつあります。これまでDNA配列解析は、タンパク質をコードしている領域(ゲノム全体の約1%)が主に行われてきました。しかし、近年のゲノム解読技術の急速な進歩により、全ゲノムのDNA配列解読が臨床現場で行われつつあります。
個体発生や疾患発症に中心的な役割を担うエンハンサーには、病気のゲノム変異が高度に濃縮されています。本研究で開発したエンハンサー解析技術がもたらす新しい知見は、次世代ゲノム医療の推進に大きく貢献すると期待できます。
さらに、国際共同研究グループは今後、NET-CAGE法を臨床検体に適用し、がんエンハンサー・ゲノム転写ネットワークの包括的な理解を目指しています。臨床データ、ゲノム配列情報、RNA発現情報、そして従来法では見えなかったエンハンサーの活性度などのエピジェネティクス[10]情報も含めた統合解析により、がんの発生・維持の根源的な分子メカニズムを解明し、新しい治療標的やバイオマーカーの同定を目指します。
原論文情報
Shigeki Hirabayashi, Shruti Bhagat, Yu Matsuki, Yujiro Takegami, Takuya Uehata, Ai Kanemaru, Masayoshi Itoh, Kotaro Shirakawa, Akifumi Takaori-Kondo, Osamu Takeuchi, Piero Carninci, Shintaro Katayama, Yoshihide Hayashizaki, Juha Kere, Hideya Kawaji and Yasuhiro Murakawa, “NET-CAGE Characterizes the Dynamics and Topology of Human Transcribed Cis-regulatory Elements”, Nature Genetics, 10.1038/s41588-019-0485-9
発表者
理化学研究所
生命医科学研究センター 理研-IFOMがんゲノミクス連携研究チーム
チームリーダー 村川 泰裕(むらかわ やすひろ)
(IFOM癌研究所グループリーダー)
大学院生リサーチ・アソシエイト 平林 茂樹(ひらばやし しげき)
(京都大学大学院 医学研究科 博士課程4年)
生命医科学研究センター 予防医療・ゲノミクス応用開発ユニット
開発ユニットリーダー 川路 英哉(かわじ ひでや)
(東京都医学総合研究所副参事研究員)
国際プログラム・アソシエイト シュルティー・バーガット(Shruti Bhagat)
(カロリンスカ大学院博士課程3年)
株式会社ダナフォーム
部長代理 松木 悠(まつき ゆう)
報道担当
理化学研究所 広報室 報道担当
株式会社ダナフォーム
京都大学 総務部 広報課 国際広報室
補足説明
-
- エンハンサー、プロモーター
- どちらも遺伝子発現を制御する機能を持つDNA配列。主に遺伝子の上流や下流に位置し、遺伝子の転写効率を変化させるDNAの特定の配列のうち、転写効率を著しく高める部分をエンハンサーという。これに対し、ゲノムDNA上でRNAとして転写される領域の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーターという。
-
- CAGE法
- 理研が独自に開発した手法で、耐熱性逆転写酵素やmRNAのCap構造を捕捉する技術を組み合わせて転写産物の5’末端の塩基配列を決定する実験手法。この塩基配列を読み取ってゲノム配列と照らし合わせて、どこから転写が始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる。CAGEはCap Analysis Gene Expressionの略。
-
- トポロジー構造
- 細胞核内にて高度に折り畳まれ、立体的に区域化されたゲノム構造のこと。エンハンサーやプロモーターといった領域は、区域内で空間的に隣接して相互作用しており、遺伝子発現制御の一翼を担う。
-
- 転写因子
- DNAに配列特異的に結合するタンパク質で、プロモーターやエンハンサーといった転写制御領域に結合し、RNAポリメラーゼによる遺伝子の転写を活性化あるいは不活性化する。
-
- ノンコーディングRNA
- タンパク質を構成するアミノ酸配列をコードするmRNAと異なり、タンパク質をコードしないRNAの総称。エピジェネティクス(塩基配列に依存しない遺伝子の調節機構)や転写、翻訳といった生物の活動の中枢をなす反応、幹細胞性の維持など、さまざまな働きに関与するノンコーディングRNAが次々に報告されており、その重要性に注目が集まっている。
-
- 一塩基多型(SNP)
- ヒトゲノムは30億塩基対のDNAからなるが、個々人を比較するとそのうちの0.1%の塩基配列の違いがある。これを遺伝子多型という。遺伝子多型のうち一つの塩基が、ほかの塩基に変わるものを一塩基多型と呼ぶ。SNPはSingle Nucleotide Polymorphismの略。
-
- ChIP-Seq法
- クロマチン免疫沈降法と次世代シーケンサーを組み合わせた網羅的解析方法。特定の転写因子やヒストン修飾に対する抗体により免疫沈降を行って、それらに結合しているDNA断片を回収し、次世代シーケンサーを用いてそのタンパク質がゲノム上のどの場所に結合しているかをゲノムワイドかつ網羅的に解析する方法。ChIPは、chromatin immunoprecipitationの略。
-
- RNAポリメラーゼ
- DNAの塩基配列を読み取って、相補的なRNAを合成する酵素で、遺伝子発現(セントラルドグマ)の第一段階をつかさどる生命活動に必須の酵素。
-
- FANTOMコンソーシアム
- FANTOMは理化学研究所が主宰する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センターの林﨑良英グループディレクター(現 予防医療・診断技術開発プログラム プログラムディレクター)が中心となり2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。第5期のプロジェクトとなるFANTOM5では、さまざまな哺乳類細胞のゲノム上の遺伝子制御部位の活性を測定し、転写状態やプロモーター活性の全容を明らかにする研究が進められた。現在のFANTOM6には20カ国、100以上の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。FANTOMは、Functional ANnoTation Of Mammalian genomeの略。
-
- エピジェネティクス
- DNA配列の変化によらず、遺伝子の発現を制御するメカニズムの総称。狭義ではDNAのメチル化やヒストン修飾による制御を指すことが多い。
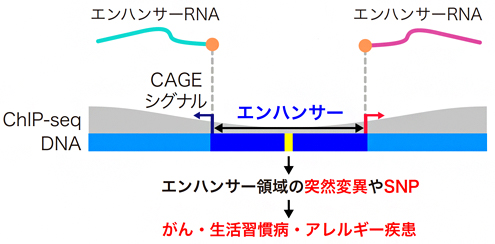
図1 CAGE法による高塩基解像度のエンハンサー領域決定法
エンハンサー領域の両端から、エンハンサーRNA(eRNA)が発現している。CAGE法はeRNAの転写開始点(オレンジ丸)を同定することで、エンハンサー領域を一塩基レベルで検出できる。ChIP-seq法では、このような高塩基解像度での同定が困難である。エンハンサー領域の突然変異によりがん化が促進される例や、エンハンサー領域の一塩基多型(SNP)が生活習慣病・アレルギー疾患のなりやすさに関連する例が報告されている。
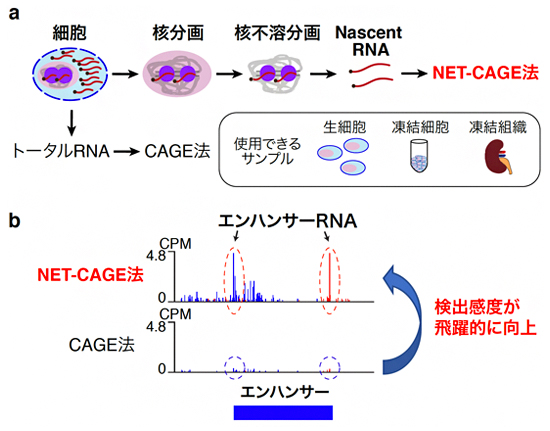
図2 NET-CAGE法の概略
(a) NET-CAGE法の工程を示す。生細胞、凍結細胞、凍結組織などの細胞から、生化学的な手法により細胞質分画を除き、核分画を濃縮する。高濃度の塩と尿素を用いて、核分画から新しく合成中のRNA(Nascent RNA)が濃縮されている核不溶分画を抽出する。この核不溶分画から精製されたNascent RNAに、CAGE法を実施する。
(b) エンハンサー領域のNET-CAGE法とCAGE法のデータの一例を示す。縦軸はeRNAの発現量を示しており、CAGE法のデータと比較してNET-CAGE法のデータでは、eRNAの検出感度が飛躍的に向上していることが分かる。
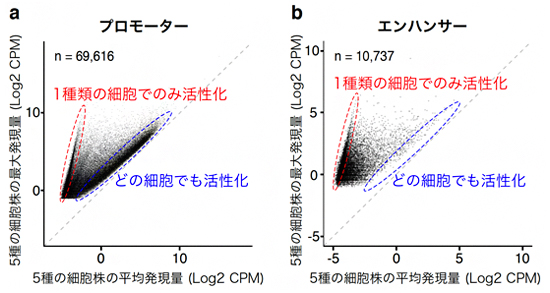
図3 がん細胞株におけるプロモーターとエンハンサーの活性化パターンの比較
5種類のがん細胞株(白血病細胞、リンパ芽球細胞、乳がん細胞、子宮頸がん細胞、肝がん細胞)におけるプロモーター(a)とエンハンサー(b)の活性化パターンを示す。5種類の細胞株の発現量がほぼ同じである場合、平均発現量(横軸)と最大発現量(縦軸)はほぼ同じ値となり、青の点線で囲まれた領域にプロットされる。つまり、5種類のどの細胞でも活性化していることを意味する。一方、5種類の細胞株のうち、1種類の細胞株のみで発現している場合、赤の点線で囲まれた領域にプロットされる。つまり、細胞特異的に活性化していることを意味する。多くのプロモーターは、青の点線で囲まれた領域に存在し、どの細胞でも活性化している。一方エンハンサーは、青の点線で囲まれた領域にほとんど存在せず、むしろ赤の点線で囲まれた領域に多く存在しており、細胞特異的に活性化していることが分かる。