2019-03-04 国立遺伝学研究所,名古屋大学,科学技術振興機構
私たちの体は約40兆個の細胞からできています。そして、それぞれの細胞には全長約2メートルにも及ぶ生命の設計図ヒトゲノムDNAが収納されています。DNAの収納構造については、近年、多くのことが分かってきた一方で、生きた細胞でのDNAのふるまい(動き)についてはほとんど分かっていませんでした。
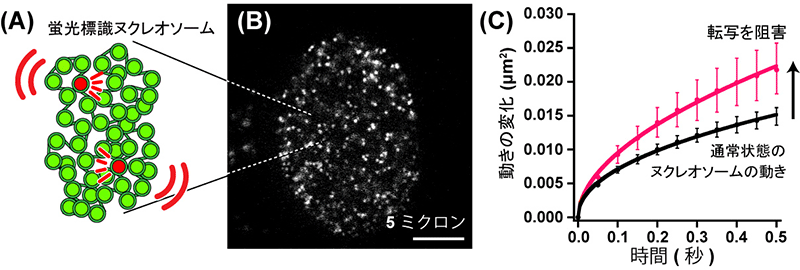
このたび、情報・システム研究機構 国立遺伝学研究所 永島 崚甫 総研大大学院生、日比野 佳代 助教、前島 一博 教授らのグループは、名古屋大学 大学院工学研究科 S.S.Ashwin 特任助教、笹井 理生 教授と共同で、光学顕微鏡の分解能を超える超解像蛍光顕微鏡注1)を駆使することで、生きた細胞内のDNAの動きを観察することに成功しました(図1、動画1)。一般に、ゲノムDNAの遺伝情報が読み出される転写注2)が起きる際、DNAを含む高次構造であるヌクレオソームは緩くなり、よりダイナミックに動くと考えられてきました。しかし本研究で調べたところ、転写を阻害するとDNAの動きが逆に活発化することが明らかになりました(図1C、動画2)。さらに、転写の際にDNA上で働くRNAポリメラーゼII注3)や他の転写因子注4)が塊(ハブ)を作ってDNAの動きを抑える様子が示されました(図3A)。この結果は、ハブを作ることでゲノムDNAは連結されてネットワーク化し、DNAの動きを抑え、効率的に転写を行う可能性を示唆するものです。
本研究の結果によって、遺伝情報がゲノムDNAからどのように読み出されるのかについての理解が進むとともに、転写の変化で起きるさまざまな細胞の異常や関連疾患の理解につながることが期待されます。
本研究成果は、米国科学雑誌「Journal of Cell Biology」に2019年3月4日午後11時(日本時間)に掲載されます。
本研究は、科学技術振興機構(JST) 戦略的創造研究推進事業(CREST)(JPMJCR15G2)、日本学術振興会(JSPS) 科研費(16H04746)、武田科学振興財団、RIKEN Pioneering Project、NIG-JOINT(2016-A2(6))、総研大2017年度学生派遣事業の支援を受けました。
<研究の背景>
私たちの体は約40兆個の細胞から構成されています。それぞれの細胞には、生命の設計図とも称される全長約2メートルのヒトゲノムDNAが収納されています。このDNAは直径2ナノメートル注5)のとても細い糸で、「ヒストン」という樽状のタンパク質に巻かれることで、直径約11ナノメートルの「ヌクレオソーム」を作っています(図2上段)。ヌクレオソームと、これに結合する他のタンパク質を合わせて「クロマチン」と呼びます。長い間、このヌクレオソームはらせん状に規則正しく折りたたまれ(クロマチン線維)、さらに巻かれて階層構造を作るとされてきました(図2中段左)。国立遺伝学研究所 前島教授らは、2008年よりクライオ電子顕微鏡注6)やX線散乱注7)の実験によって、定説のような規則正しい階層構造は存在せず、ヌクレオソームが不規則に、そしてダイナミックに細胞内に収められていることを提唱してきました(図2中段右)。それ以降、生きた細胞でのヌクレオソームのふるまいについて理解が求められてきましたが、ヌクレオソームの動きは非常に小さく、従来の光学顕微鏡を用いて観察することは困難でした。
<研究内容>
本研究では、1個1個のヌクレオソームを観察できる超解像蛍光顕微鏡を駆使することで、ヌクレオソームのふるまいを生きたままの細胞で観察することを実現しました(図1A)。具体的には、細胞1個に3000万個程度存在するヌクレオソーム(図2上段)をまばらに蛍光で目印(標識)を付けることにより(図1A、B)、個々のヌクレオソームの動きを正確に調べることが可能となりました。その結果、多数のヌクレオソームが核内でダイナミックに動く様子を明らかにしました(図1C、動画1)。次にDNAの遺伝情報が読み出される「転写」が起きる際、ヌクレオソームの動きにどのような変化があるのかを調べました。一般に、転写が起きる部分では、クロマチンの折り畳みは緩くなり、ヌクレオソームはよりダイナミックに動くと考えられてきました。しかし本研究で調べたところ、転写を阻害するとヌクレオソームの動きが逆に活発化することが明らかになりました(図1C赤線、動画2)。さらに転写の際にDNA上でRNAポリメラーゼIIや他の転写因子が働くときに作られる塊(ハブ)を介して、クロマチンの動きが抑えられる様子が示されました(図3A)。この知見は細胞内のクロマチンの動きを再現した計算機シミュレーションの結果とも一致していました(図3B、動画3、動画4)。細胞核内のクロマチンは転写が起きる場所をハブとし、緩いネットワークを作っていると考えられます(図3A右)。この結果は、クロマチンはハブを作ることでヌクレオソームの動きを抑え、転写の作業効率を向上させる可能性を示唆するものです。
<今後の期待>
細胞の多くの活動は、DNA上の遺伝情報を読み出す転写から始まります。本研究で明らかになった転写ハブはクロマチンの動きを抑え、転写の制御・効率化に関わっていると考えられます。また、転写の異常によって、細胞にガン化・細胞死、さらには分化異常など、さまざまな細胞障害をもたらすことが近年分かってきました。本研究の成果によって、このような細胞の異常や関連疾患の理解が進むことが期待されます。
<動画>
- (動画1)https://youtu.be/tVXjDRKr404
- 超解像蛍光顕微鏡による核内のヌクレオソームの動きの動画。通常状態の細胞内のヌクレオソームの動き。1個1個のドットが1個1個のヌクレオソームを示している。 1コマ50ミリ秒。
- (動画2)https://youtu.be/JK79I7z4Upk
- 転写を阻害した際のヌクレオソームの動きの動画。通常の状態と比べて、ヌクレオソームの動きが活発化していることが分かる。1コマ50ミリ秒。
- (動画3)https://youtu.be/BXIHc88vbUQ
- 転写ハブが存在する状況でのクロマチンの動きの再現。緑のボールはクロマチンの塊、ピンクのボールは転写ハブ、赤いボールはRNAポリメラーゼIIを表している。転写ハブがないときに比べて、緑のボールの動きが抑えられているのが分かる。
- (動画4)https://youtu.be/CibfDvtE5LM
- 計算機シミュレーションによるクロマチンの動きの再現。緑のボールはクロマチンの塊を表しており、緑のボールが激しく動いているのが分かる。この動画は転写が阻害された状況を示している。
<参考図>
![]()
図1
(A)少数の蛍光標識されたヌクレオソーム(赤)。それらに注目することで個々のヌクレオソームの動きを調べることができる。(B)超解像蛍光顕微鏡による核内のヌクレオソーム画像。1個1個のドットが1個1個のヌクレオソームを示している。(C)転写を阻害するとヌクレオソームの動きが上昇する(赤線)。黒線が通常状態のヌクレオソームの動き。ヌクレオソームの動きは「平均二乗変位注8)」という量で表されている。
![]()
図2
(上段)DNAはヒストンに巻かれてヌクレオソームを形成する。ここでは3個のヌクレオソームが描かれているが、1つの細胞当たり、3000万個ものヌクレオソームが存在する。
(中段)DNAの細胞内収納に関するこれまでの定説(左)と現在の説(右)。
![]()
図3
(A)細胞の核(左)の中で転写が起きる際、DNA上で働くRNAポリメラーゼII(赤)や他の転写因子(青)が塊(ハブ、ピンク色)を作ってクロマチンの動きを抑える(A中央、右)。細胞核内のクロマチンは転写が起きる場所をハブとし、緩いネットワークを作っていると考えられる(A右)。(B)細胞内のクロマチン状態を再現した計算機シミュレーション。転写ハブを作ることでクロマチンの動きが抑えられていることが確かめられた。
<用語解説>
- 注1)超解像蛍光顕微鏡
- 通常の光(可視光)を用いて顕微鏡観察する場合は、200ナノメートル程度の大きさのモノを解像するのが限界(光の回折限界)である。しかし、超解像蛍光顕微鏡はこの限界を超えて(超解像)、より小さな構造まで観察することができる。本研究では、クロマチンのヌクレオソームをまばらに蛍光標識することで超解像を達成する方法を用いた。
- 注2)転写
- 生物のゲノムDNAの塩基配列(主に遺伝子)を基に、RNA(転写産物)を合成すること。遺伝情報が読み出されるための過程。
- 注3)RNAポリメラーゼII
- DNAの塩基配列を読み取って相補的なRNAを合成する反応(転写)を触媒する際、中心となって働く巨大なタンパク質複合体。図3の赤いボール。
- 注4)転写因子
- DNAの遺伝情報をRNAに転写する際、その領域に特異的に結合するスイッチタンパク質。DNA上の転写を制御する領域に結合し、転写を促進する。図3の青いボール。
- 注5)ナノメートル
- 1メートルの10の9乗分の1(10-9)。
- 注6)クライオ電子顕微鏡
- 生きた状態に近い生物材料を観察することができる電子顕微鏡。「生きている」状態を保存するために、細胞を急速凍結し、凍結した細胞を極低温下(-150度)で薄く切り(切片化)、その切片を極低温下でそのまま観察する。
- 注7)X線散乱
- タンパク質などが集まった構造体にX線を当てると、その構造体の規則性に応じた散乱パターンが得られる。このことを利用し、構造を調べる方法。大型放射光施設SPring-8で強力なX線である放射光を用いて実験を行った。
- 注8)平均二乗変位
- ある時間の間に粒子が移動した距離の二乗を平均した量。この量は、粒子が動く程度を表現するために使われる。
<論文情報>
タイトル:“Single nucleosome imaging reveals loose genome chromatin networks via active RNA polymerase Ⅱ”
(一分子ヌクレオソームイメージングはRNAポリメラーゼIIによるクロマチンネットワークの存在を明らかにする。)
著者名:Ryosuke Nagashima, Kayo Hibino, S. S. Ashwin, Michael Babokhov, Shin Fujishiro, Ryosuke Imai, Tadasu Nozaki, Sachiko Tamura, Tomomi Tani, Hiroshi Kimura, Michael Shribak, Masato T.Kanemaki, Masaki Sasai, and Kazuhiro Maeshima
<お問い合わせ先>
<研究に関すること>
前島 一博(マエシマ カズヒロ)
国立遺伝学研究所 遺伝メカニズム研究系 ゲノムダイナミクス研究室 教授
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部 ライフグループ
<報道担当>
国立遺伝学研究所 リサーチ・アドミニストレーター室 広報チーム
名古屋大学 総務部 総務課 広報室
科学技術振興機構 広報課